讲解视频内容请移步Bilibili:
https://space.bilibili.com/542601735
入群讨论请加vhochzeitstorte
请注明“核磁共振学习”
3D Slicer实现纤维追踪(大脑纤维束成像)
1、选择模块Tractography Seeding
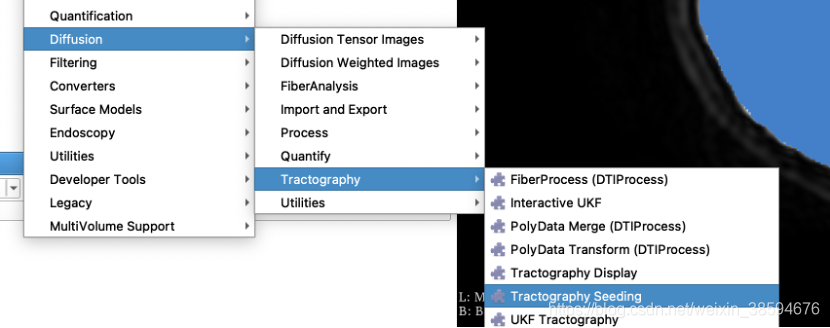
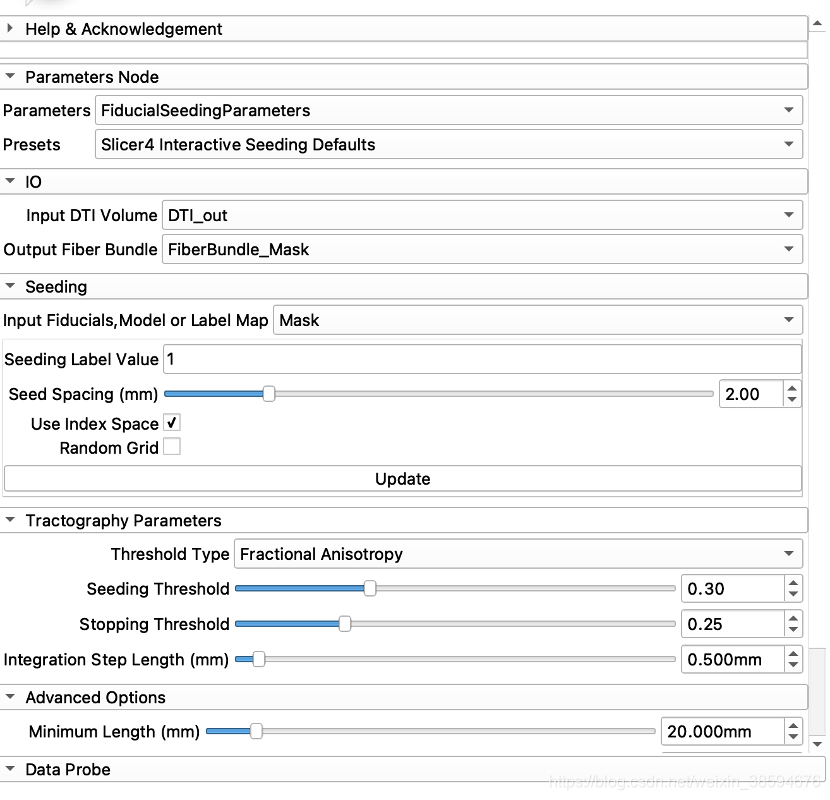
2、参数选择
Parmeters Node(参数节点):
Parameters(参数):选择现有设置或创建一个新的设置。
Presets(预设):
Slicer4 Interactive Seeding Defaults:默认的Slicer4交互种子。
Slicer3 Labelmap Seeding Defaults:默认的Slicer3标签映射种子。
Slicer3 Fiducial Seeding Defaults:默认的Slicer3基准点种子。
Input DTI Volume:输入DTI影像
Output Fiber Bundle:输出纤维束
Seeding(种子):
Input Fiducials,Model or Label Map:选择一个现有的基准点列表、模型或标签映射体数据作为跟踪的种子。
Seeding Label Value:种子标签值,默认为1。
Seed Spacing(mm):种子间距,表示选取种子间的距离,单位为毫米,注意功能该选项仅在没有使用索引空间(Use Index Space)时才生效。
Use Index Space:使用索引空间,勾选后在IJK中选择体素作为种子。
Random Grid:随机网格,随机选取种子点。
纤维束参数(Tratography Parameters)
Threshold Type:阈值类型,Fractional Anisotropy
Seeding Threshold:种子开始阈值。例如上面选择FA,则只有FA不小于该值时开始做纤维追踪。
Stopping Threshold:种子停止阈值。
Integration Step Length(mm):纤维追踪的步进长度。
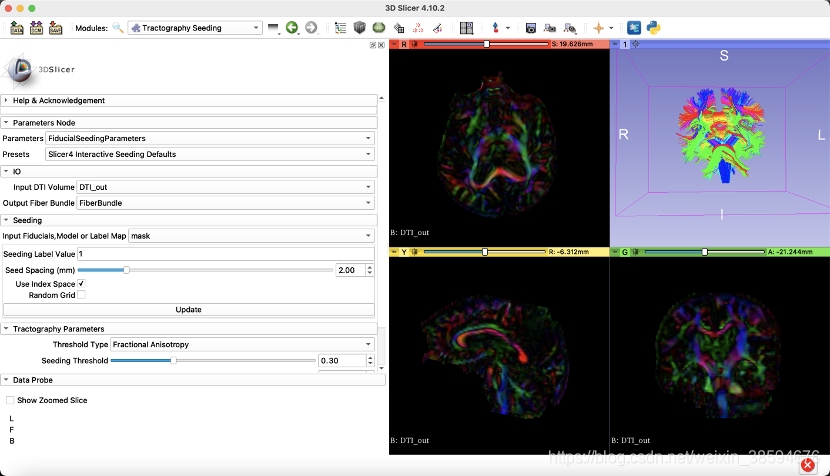
结果展示
下期预告:概率纤维追踪实践。





















 6007
6007











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








