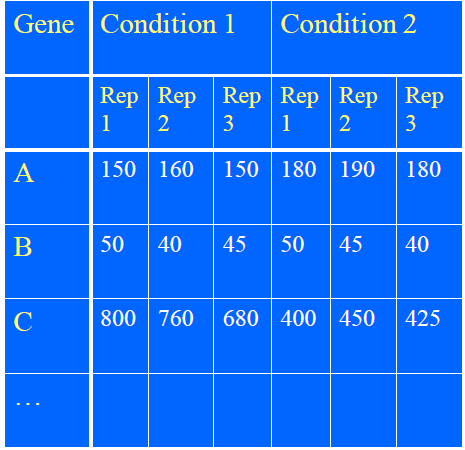
这里我们就要讲讲如何从海量的芯片数据中寻找到差异表达的基因。
首先,我们得知道为什么我们需要找这些差异表达的基因。其实在肿瘤的发生发展过程中,很多平时沉默的基因开始高表达,而原本那些正常表达的基因,它们的表达量可能就会下调。也恰恰这些与平时正常基因表达量发生变化的基因,它们的存在启动了肿瘤的发生。所以,如果我们要研究肿瘤发生的机制,研究这些差异表达的基因是必不可少的。
利用在线工具GEO2R寻找差异表达的基因
那么这里,小编给大家介绍一个简单且容易上手的在线工具——GEO2R。同样,我们根据上一讲给的网站点击进入(https://www.ncbi.nlm.nih.gov/geo/),我们输入gastric cancer,回车。
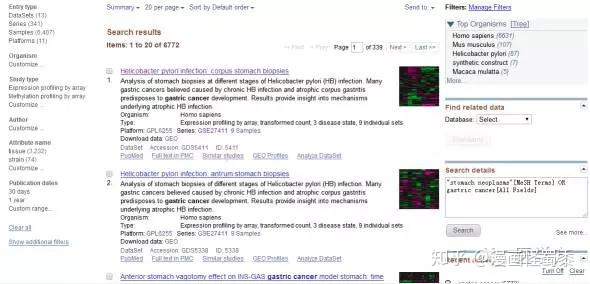
然后点击第一项,进入下面这个界面。
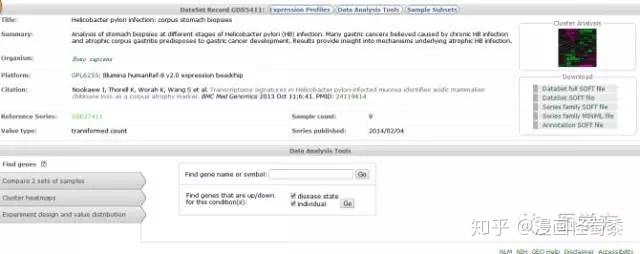
再点击GSE27411,进入以下界面。

然后点击Analyze with GEO2R。

点击Define groups,这里我们假设分为两组,分别是infected和uninfected。首先我们先输入infected,然后回车。接着再输入uninfected, 回车。完成后显示如下。
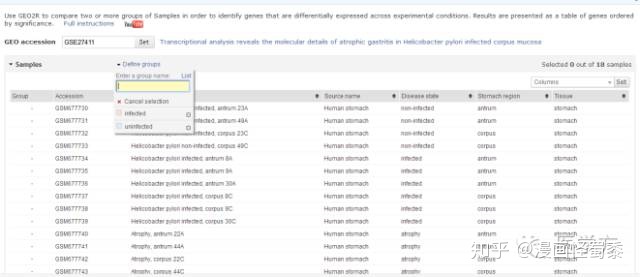
接下去我们可以看到下方有一个表格,其中前4行是unifected,接下去6行是infected。这里先点击第一行,条带会显示黄色,然后再点击Define groups里的uninfected,完成后原本黄色的条带会显示为粉红色,如下图。
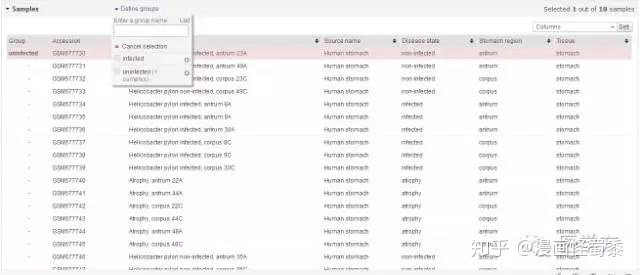
依次类推。。。
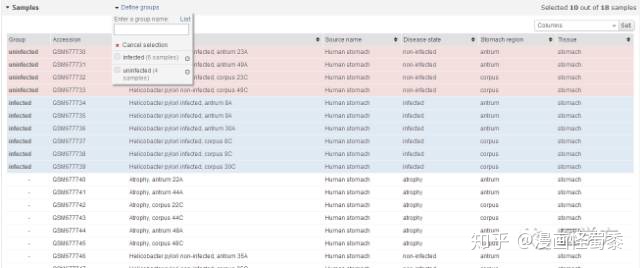
分类完成后,鼠标往下滑,点击Top 250,最后界面显示如下。
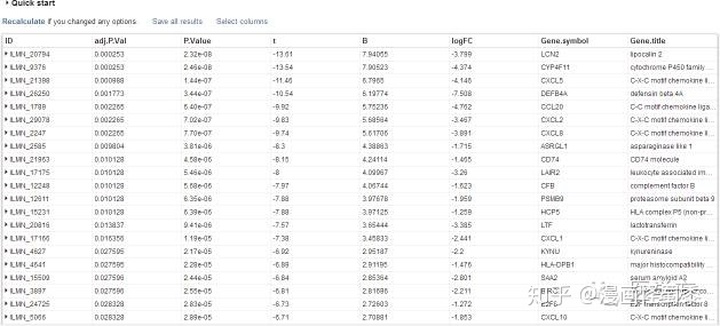
操作到现在,其实寻找差异基因的步骤已经基本结束了,但是大家或许对这个表格的解读还是存在疑惑,接下去,大家跟着小编一起来解读一下这个表格的具体内容。
这里,所有基因的排序是根据P.Val值从小到大依次排列的,P值越小,越有理由相信该基因在组与组之间存在差异表达,而adj.P.Val是经过校正后的P值,其意义与P值一样,但更加准确。B是经过bayes调整后得到的标准差的对数值,t是经调整后,所要比较的两组表达值经T检验后的t值。logFC指的是两组表达量间以2为底对数化的变化倍数。这么几个指标中,最最重要的是adj.P.val和logFC。至于结果的保存,经过小编个人操作后,建议直接在表格上选定,复制黏贴到excel。不过,界面中有一个save all results,点进去后如下图。

如果你从上图中选定后复制黏贴到excel中,你会发现所有数据都集中在一个格子中,而不是每个数据都落在各自的格子里。比较不方便后续的数据处理。
最后提一点,在之前的那张表格里,仔细的同学会看到有些个别几行没有gene.symbol,如下图。
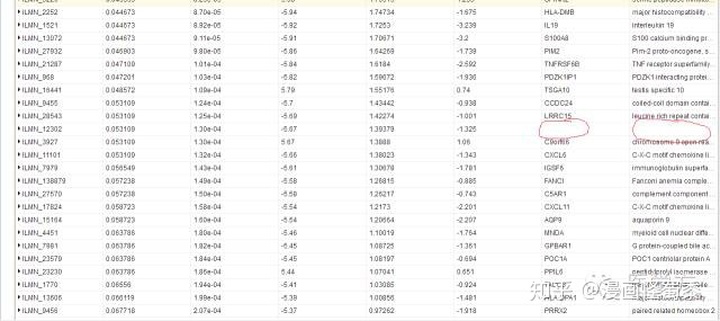
这是为什么呢?道理很简单,人类转录组有很多成分,除了我们平时讲的mRNA,还有非编码RNA,假基因RNA,核糖体RNA等等。基因芯片检测的时候,都会涉及到这些RNA,但他们中间有些要么不是基因,要么功能还没明确,甚至未被命名,所以一般在分析的时候都会把这些没有symbol的探针直接滤过就可以了。
如何应用GEO2R的在线工具来帮助寻找差异基因
今天小编再教大家一招,如何使用gene spring来再次实现这个功能。那么我们打开上一节最后生成的界面,如图。
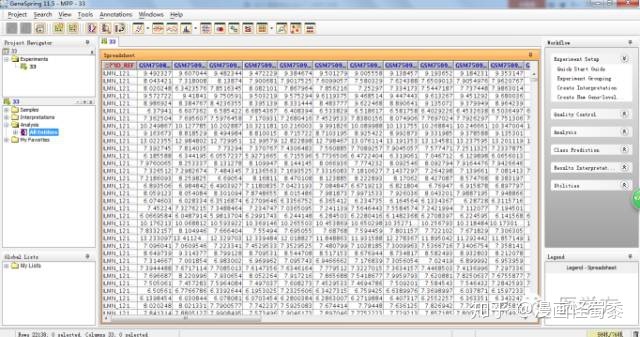
接下去我们先要对所有的样本进行分组,只有分了组,才能进行比较。首先我们点击右上角的Experiment grouping,然后选择Add Parameter。
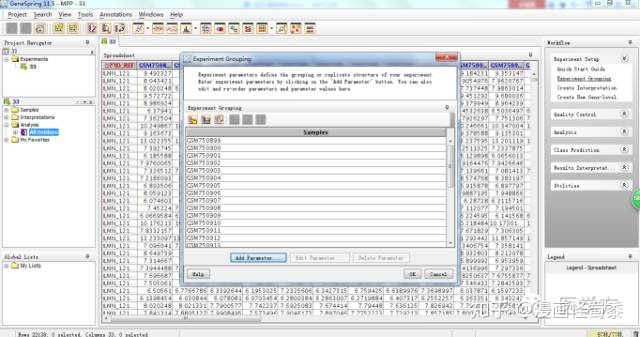
这里Parameter name中我们命名为group,参数类型选择非数值型。
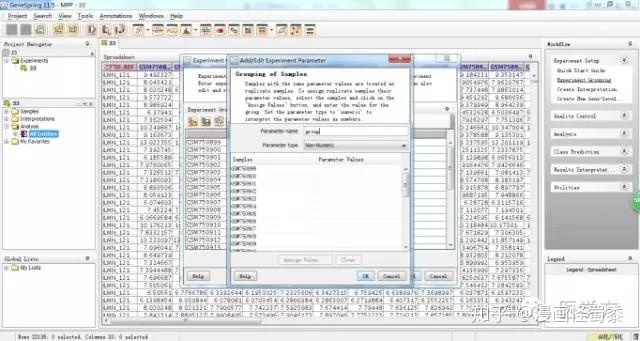
现在总共有32个样本,假设我们分为两组,分别为control和drug。先选择前16个样本,然后点击Assign value,出现对话框后我们命名为control。
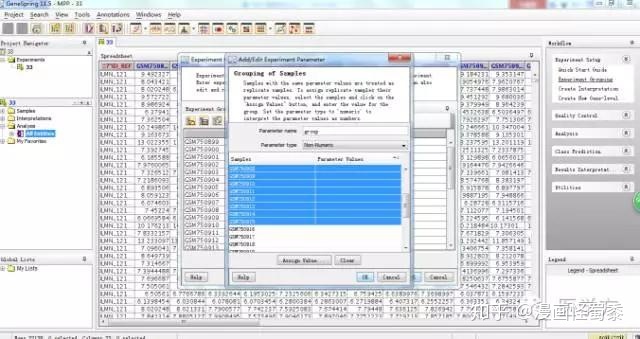
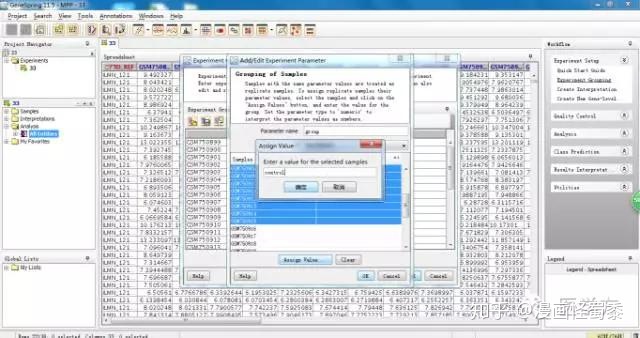
同样道理,我们用同样的方法给后16组命名为drug。如图,点击OK。
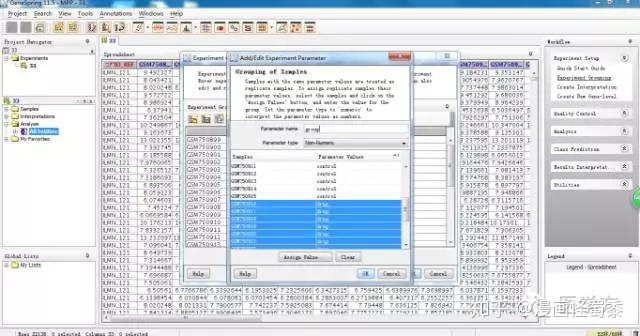
接着我们点击右上方的Create interpretation,这里我们选择非连续性。接下去几步都默认即可。
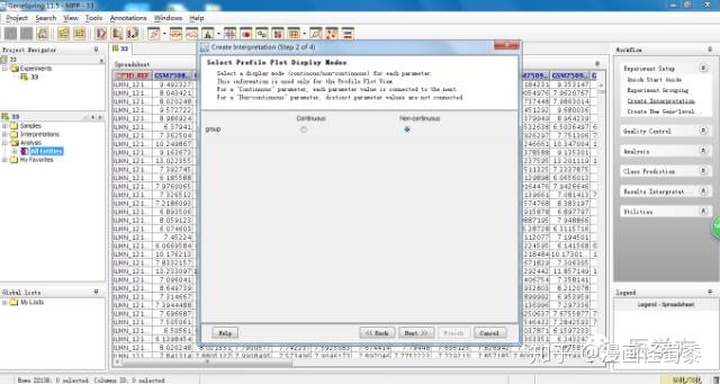
接着我们点击下图中红色圈的statistical analysis, 进行统计分析。
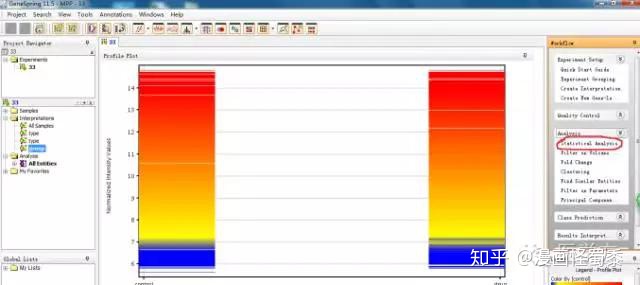
这里我们选择非配对T检验,具体需要根据每个实验的具体情况而定。
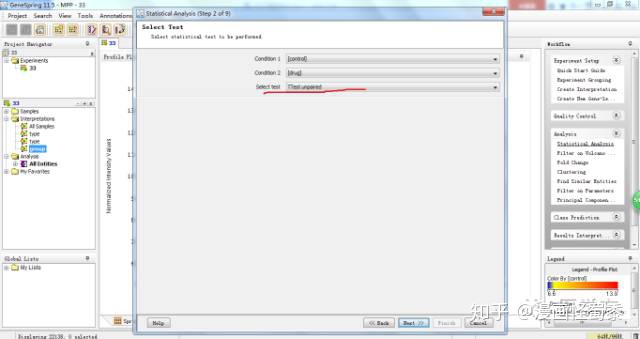
在这块我们假设,如果两组样本之间基因差异要有统计学意义,则p<0.05。当然如果最后发现差异基因有很多,你可以把p控制在小于0.01。
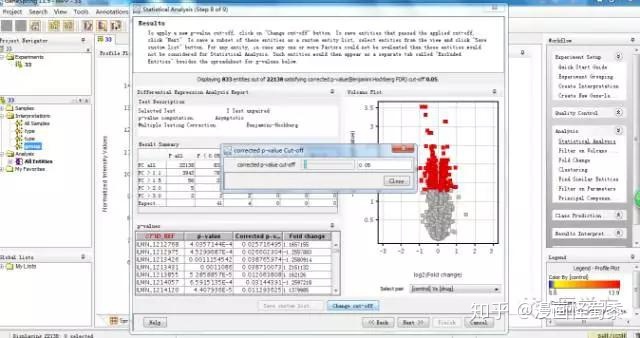
这里你可以发现有833个基因存在表达差异。其他默认。
之前我们筛选出来的都是有表达差异的基因,接下去我们要挖掘这些基因彼此间差异到底有多大呢,这个值是我们可以人为设定的。那接下去我们需要点击右侧的fold change,如图。
这一步我们默认。
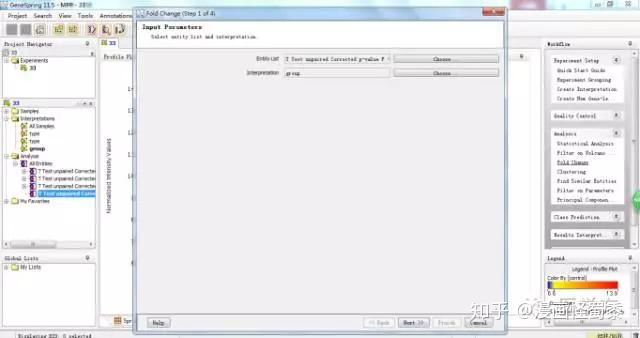
这个对话框里我们选择Pairs of conditions。
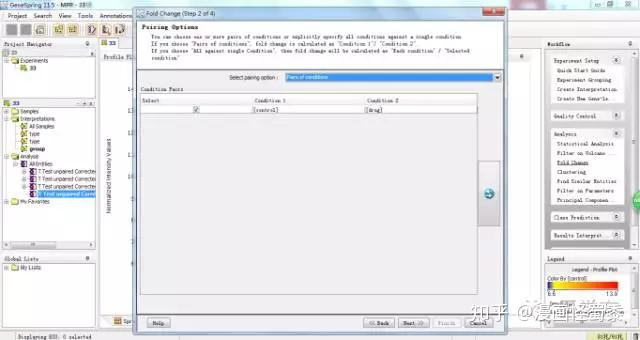
同样这里我们把差异控制在2倍以上。
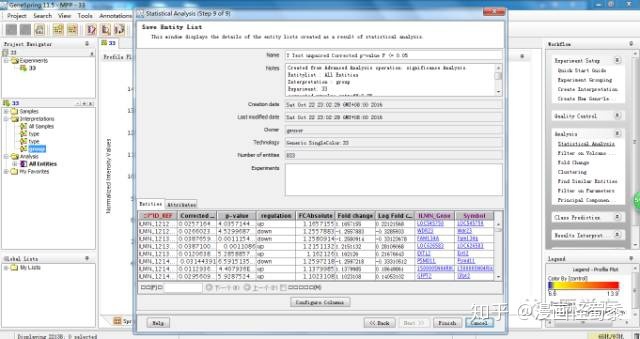
最后发现只剩下3个基因。其他步骤我们均默认。
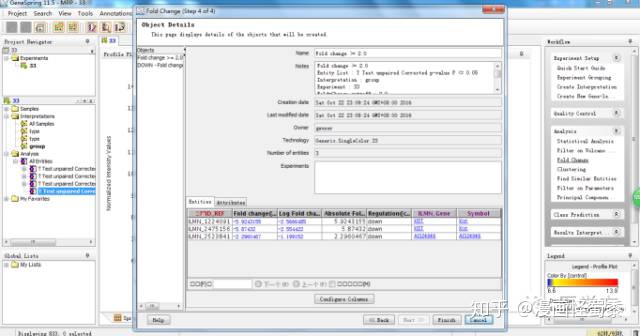
你会发现这3个基因均是表达下调的。
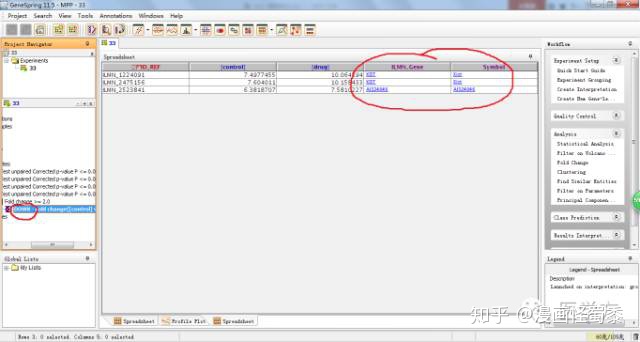
其实整个过程很简单,就是通过这个软件先进行分组,然后设定p值,找出有统计学差异的基因,在这个基础上,我们再人为设定差异的倍数,最终找到那几个差异表达最大的基因。




















 738
738











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








