STRING (search tool for recurring instances of neighbouring genes)是欧洲分子生物实验室(European Molecular Biology Laboratory, EMBL)Peer Bork团队开发的关于基因间功能关联关系的数据库。自2000年上线以来,目前已经更新到第11版。2019年的最新版包括了共5090个物种、24,584,628个蛋白、3,123,056,667个相互作用。
在网络药理学研究中,STRING数据库通常被应用于构建靶标蛋白之间的蛋白-蛋白相互作用网络。事实上,STRING的功能非常强大,它还有更多的用途,本期将通过几个例子,详细介绍这些用法。
一、蛋白名称及ID的批量影射
例1. 将蛋白名称转化为gene symbol.
中药网络药理学研究中,大家常用的一个中药数据库是TCMSP。但是这个数据库给出的靶标信息是靶标的蛋白名称,这种一长串的英文名称在后续的网络构建、分析、可视化中有诸多不便,通常需要把它转化成gene symbol。用STRING实现批量转换的方法如下图:
粘贴蛋白名称数据、选择物种、点击搜索:

结果下载:
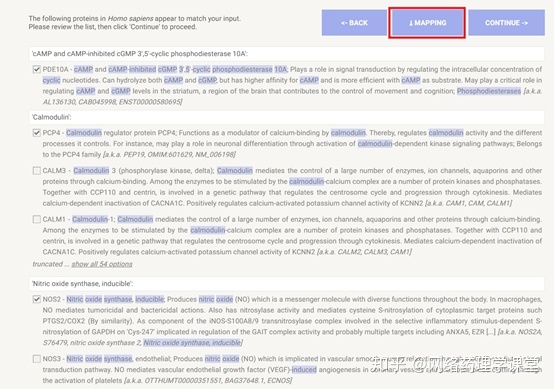
例2. 识别一列Uniprot ID 中非人源的蛋白。
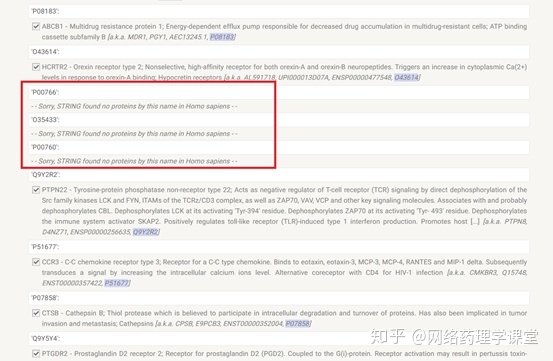
TargetNet平台预测化合物的靶标, 是将输入的化合物针对623个常用靶标进行预测,并给出预测分。但是,623个靶标中有一些不是人源蛋白。如何剔出这些靶标呢?可以将TargetNet输出的靶标的Uniprot ID粘贴到STRING,物种选择人,没有影射到的蛋白就是非人源蛋白。
粘贴Uniprot ID、选择物种、点击搜索:

STRING将UNIPROT ID影射到gene symbol, 没有影射到的就是非人源蛋白。
二、构建多个蛋白之间的相互作用网络:
首先介绍一下STRING数据库构建的网络中边的含义。根据STRING数据库的文档介绍,数据库里面的蛋白-蛋白相互作用代表的是蛋白之间功能的关联性。STRING的数据来自整合各种生物学数据,并包含大量预测的关联性,并使用Confidence Score将不同来源数据的可靠性整合成一个可靠性分值。STRING将低可靠性、中等可靠性、高可靠性、最高可靠性的分值分别设置为0.15, 0.4, 0.7,0.9。在构建网络是,它默认的可靠性分是0.4。
STRING里边的数据来源见下图:

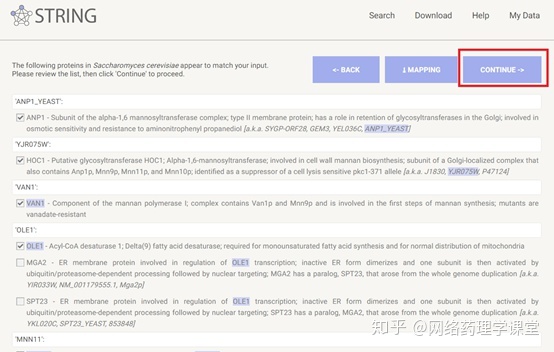
例3. 构建仅包含输入蛋白的相互作用网络。
粘贴蛋白list、选择物种、点击搜索:

点击继续:
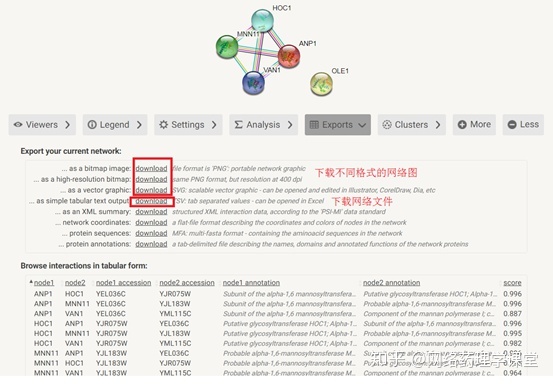
获得构建的网络:

下载网络:
例4. 构建不仅包含输入蛋白、还包含其它与当前蛋白关联最强的蛋白的相互作用网络
在例3的结果页面,点击“More”,得到加入了更多蛋白的网络。可以继续点击“More”, 持续加入更多蛋白。点击“Less”, 会删除刚加入的蛋白。见下图:

点击“Legend”, 会列出输入节点和加入的多余节点:


三、蛋白功能富集分析
STRING也可以象DAVID一样,做GO和KEGG等富集分析,而且操作更简便。
例5. 对构建的网络中的蛋白作功能富集分析。
在构建好网络的页面,点击“Analysis”,得到网络的基本拓扑指标、以及网络中蛋白的功能富集分析结果,并可以分项下载。见下图:


例6. 对转录组学、蛋白组学实验结果数据作功能富集分析
通常对转录组学、蛋白组学实验结果,需要分别设定fold change, abundance等的阈值,识别出显著差异表达的基因和蛋白,再对这部分基因或蛋白进行功能富集分析。STRING第11版新上线的这个新功能,可以将整个组学结果按fold change, 或abundance,或p-value值排序输入,进行功能富集分析。输入格式是2列,第1列是蛋白名字,第2列是该蛋白对应的数值,要求按从小到大或者从大到小的顺序排列输入数据。分析的结果会告诉你是列表的顶部、底部还是两侧数据富集在相应的功能上。
输入:

结果:将鼠标点在某个功能上,左上方会显示落在这个功能上的基因位于列表的位置,左下方显示落在这个功能上的基因位于全网络的位置。

结果下载:

本期分享到此,希望大家充分利用STRING强大的功能
参考文献:
Szklarczyk D, Gable AL, Lyon D, Junge A, Wyder S, Huerta-Cepas J, Simonovic M, Doncheva NT, Morris JH, Bork P, Jensen LJ, von Mering C.STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets. Nucleic Acids Res. 2019 Jan; 47:D607-613.







 STRING数据库由欧洲分子生物实验室开发,用于研究基因间的功能关联。本文详细介绍如何利用STRING进行蛋白名称转换、构建蛋白相互作用网络及功能富集分析等功能。
STRING数据库由欧洲分子生物实验室开发,用于研究基因间的功能关联。本文详细介绍如何利用STRING进行蛋白名称转换、构建蛋白相互作用网络及功能富集分析等功能。
















 9303
9303

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








