AlphaFold2源码解析(9)–模型之损失
损失函数和辅助头 该网络是端到端训练的,梯度来自主帧对齐点误差 (FAPE) 损失
L
F
A
P
E
L_{FAPE}
LFAPE和许多辅助损失。 每个示例的总损失可以定义如下

其中
L
a
u
x
L_{aux}
Laux是结构模块的辅助损失(中间结构的平均 FAPE 和扭转损失,定义在算法 20 第 23 行),
L
d
i
s
t
L_{dist}
Ldist是分布图预测的平均交叉熵损失,
L
m
s
a
L_{msa}
Lmsa是屏蔽 MSA 预测平均交叉熵损失,
L
c
o
n
f
L_{conf}
Lconf是 1.9.6 小节中定义的模型置信度损失,
L
e
x
p
L_{exp}
Lexp解析是 1.9.10 小节中定义的“实验解决”损失,
L
v
i
o
l
L_{viol}
Lviol是 1.9.11 小节中定义的违规损失。 最后两个损失仅在微调期间使用。为了降低短序列的相对重要性,我们将每个训练示例的最终损失乘以裁剪后残基数的平方根。 这意味着所有长于作物大小的蛋白质的权重相等,而较短的蛋白质则受到平方根惩罚。
FAPE、辅助、直方图和 MSA 损失的目的是将单独的损失附加到模型的每个主要子组件(包括配对和 MSA 最终嵌入)作为训练每个单元的“目的”指南. FAPE 和 aux 是 Structure 模块的直接结构术语。直方图损失确保Pair表示中的所有条目与相关的 i j ij ij 残基对具有明确的关系,并确保配Pair表示对结构模块有用(消融显示这只是一个很小的影响)。直方图也是一种分布预测,因此它是我们解释模型在域间交互中的置信度的一种方法。 MSA 损失旨在迫使网络考虑序列间或系统发育关系来完成 BERT 任务,我们打算以此作为一种方式来鼓励模型考虑类似协同进化的关系,而无需明确编码协方差统计(这是意图,但我们只观察到它提高模型准确性的结果)。非常小的置信度损失允许构建 pLDDT 值而不会影响结构本身的准确性——我们之前在训练后微调了这个损失,但从一开始就以小的损失进行训练同样准确。最后,“违规”损失会促使模型生成具有正确键几何形状并避免冲突的物理上合理的结构,即使在模型高度不确定结构的情况下也是如此。这可以避免在最终的 AMBER 松弛中出现罕见的故障或精度损失。在训练早期使用违规损失会导致最终准确度略有下降,因为模型过度优化以避免冲突,因此我们只在微调期间使用它。
各种损失权重是手动选择的,并且仅在 AlphaFold 开发过程中略微调整(通常在引入损失项时尝试每个损失系数的几个值,之后很少调整权重)。 我们在模型开发的早期对 FAPE、直方图和 MSA 损失的比率进行了一些调整,但在模型开发过程中并没有重新调整太多。 对这些权重进行自动化或更广泛的调整可能会提高准确性,但我们通常没有观察到对激励我们这样做的精确值的强烈敏感性。 下面我们提供了应用于 Evoformer 输出表示以获得辅助预测的单个损失和转换的详细信息。
def loss(module, head_config, ret, name, filter_ret=True):
if filter_ret:
value = ret[name]
else:
value = ret
loss_output = module.loss(value, batch)
ret[name].update(loss_output)
loss = head_config.weight * ret[name]['loss']
return loss
for name, (head_config, module) in heads.items():
......
total_loss += loss(module, head_config, ret, name)
侧链和主链扭转角损失
预测的侧链扭转角和骨架扭转角用单位圆上的点表示,即 a ⃗ ^ i f ∈ R 2 \hat{\vec{a}}^f_i \in R^2 a^if∈R2 且 ∣ ∣ a ⃗ ^ i f ∣ ∣ = 1 ||\hat{\vec{a}}^f_i||=1 ∣∣a^if∣∣=1 ,将它们以 R 2 R^2 R2中 L 2 L2 L2损失与真实扭转角 a ⃗ i t r u e , f \vec{a}^{true,f}_i aitrue,f进行比较。它在数学上等价于夹角差的余弦。
一些侧链部分是180旋转对称的,因此预测的扭转角 χ χ χ和 χ + π χ + π χ+π得到相同的物理结构。我们允许网络产生任意一个扭转角通过提供另一个角度 α ⃗ i a l t t r u t h , f = α ⃗ i t r u e , f + π \vec{\alpha}_i^{alt truth, f}=\vec{\alpha}_i^{true, f}+\pi αialttruth,f=αitrue,f+π, 对于所有的非对称构型,我们设 α ⃗ i a l t t r u t h , f = α ⃗ t r u e , f \vec{\alpha}_i^{alt truth,f}=\vec{\alpha}^{true,f} αialttruth,f=αtrue,f。
引入了一个小的辅助损失 L a n g l e n o r m L_{anglenorm} Langlenorm ,使预测点靠近单位圆。 这有两个原因:一是避免向量太靠近原点,这会导致数值不稳定的梯度。 另一个是虽然向量的范数不影响输出,但它确实影响网络的学习动力。 当查看梯度在归一化的反向传递中如何变换时,梯度将被非归一化向量的范数重新缩放。
由于模型是高度非线性的,这些向量的长度可以在训练过程中发生强烈变化,导致不期望的学习动态。加权因子是在特定的基础上选择的,测试了几个值,并选择最小的一个,使向量的范数保持稳定。在模型性能方面,我们没有观察到任何对精确值的强烈依赖。

两个角(α和β)用L2范数比较表示为单位圆上的点,在数学上等价于角差的余弦

第一个恒等式就是普通的余弦差公式。
def supervised_chi_loss(ret, batch, value, config):
"""Computes loss for direct chi angle supervision.
Jumper et al. (2021) Suppl. Alg. 27 "torsionAngleLoss"
Args:
ret: Dictionary to write outputs into, needs to contain 'loss'.
batch: Batch, needs to contain 'seq_mask', 'chi_mask', 'chi_angles'.
value: Dictionary containing structure module output, needs to contain
value['sidechains']['angles_sin_cos'] for angles and
value['sidechains']['unnormalized_angles_sin_cos'] for unnormalized
angles.
config: Configuration of loss, should contain 'chi_weight' and
'angle_norm_weight', 'angle_norm_weight' scales angle norm term,
'chi_weight' scales torsion term.
"""
eps = 1e-6
sequence_mask = batch['seq_mask']
num_res = sequence_mask.shape[0]
chi_mask = batch['chi_mask'].astype(jnp.float32)
pred_angles = jnp.reshape(
value['sidechains']['angles_sin_cos'], [-1, num_res, 7, 2])
pred_angles = pred_angles[:, :, 3:]
residue_type_one_hot = jax.nn.one_hot(
batch['aatype'], residue_constants.restype_num + 1,
dtype=jnp.float32)[None]
chi_pi_periodic = jnp.einsum('ijk, kl->ijl', residue_type_one_hot,
jnp.asarray(residue_constants.chi_pi_periodic))
true_chi = batch['chi_angles'][None]
sin_true_chi = jnp.sin(true_chi)
cos_true_chi = jnp.cos(true_chi)
sin_cos_true_chi = jnp.stack([sin_true_chi, cos_true_chi], axis=-1)
# This is -1 if chi is pi-periodic and +1 if it's 2pi-periodic
shifted_mask = (1 - 2 * chi_pi_periodic)[..., None]
sin_cos_true_chi_shifted = shifted_mask * sin_cos_true_chi
sq_chi_error = jnp.sum(
squared_difference(sin_cos_true_chi, pred_angles), -1)
sq_chi_error_shifted = jnp.sum(
squared_difference(sin_cos_true_chi_shifted, pred_angles), -1)
sq_chi_error = jnp.minimum(sq_chi_error, sq_chi_error_shifted)
sq_chi_loss = utils.mask_mean(mask=chi_mask[None], value=sq_chi_error)
ret['chi_loss'] = sq_chi_loss
ret['loss'] += config.chi_weight * sq_chi_loss
unnormed_angles = jnp.reshape(
value['sidechains']['unnormalized_angles_sin_cos'], [-1, num_res, 7, 2])
angle_norm = jnp.sqrt(jnp.sum(jnp.square(unnormed_angles), axis=-1) + eps)
norm_error = jnp.abs(angle_norm - 1.)
angle_norm_loss = utils.mask_mean(mask=sequence_mask[None, :, None],
value=norm_error)
ret['angle_norm_loss'] = angle_norm_loss
ret['loss'] += config.angle_norm_weight * angle_norm_loss
帧对齐点错误(FAPE)
帧对齐点误差(Frame Aligned Point Error, FAPE)对一组预测局部帧 { T i } \{T_i\} {Ti}下的一组预测原子坐标 { x ⃗ j } \{\vec{x}_j\} {xj}与对应的地面真值原子坐标 x ⃗ j t r u e {\vec{x}^{true}_j} xjtrue和真值局部帧 { T i t r u e } \{T^{true}_i\} {Titrue}进行评分。最终的FAPE损失对所有主链和侧链框架中的所有原子进行评分。此外,在结构模块的每一层中使用一个更便宜的版本作为辅助损耗。
为了表述损失,我们计算了相对于坐标系 T i T_i Ti的原子位置 x ⃗ j \vec{x}_j xj和相应的真原子位置 x ⃗ j t r u e \vec{x}^{true}_j xjtrue相对于真坐标系 T i t r u e T^{true}_i Titrue的位置。偏差计算为稳健的L2范数。( ϵ \epsilon ϵ是一个添加的小常数,以确保梯度在数值上表现良好。这个常数的确切值并不重要,只要它足够小。我们在实验中使用了 1 0 4 10^4 104和 1 0 12 10^{12} 1012的值)。由此产生的 N f r a m e s × N a t o m s N_{frames} \times N_{atoms} Nframes×Natoms偏差用长度刻度 Z = 10 A ˚ Z = 10 Å Z=10A˚的l1损失进行惩罚,以使损失无单位。
在本节中,我们表示Å中的点位置和超参数,尽管损失对单位的选择是不变的。
我们现在讨论在真实结构和预测结构的整体刚性变换下损失的行为。首先,我们应该注意到 x ⃗ i j \vec{x}_{ij} xij在刚性运动(不包括反射)下是不变的;因此,如果通过任意旋转和平移,预测结构与真实值不同,损失将保持不变。然而,由于局部框架的构造方式,由于局部框架的z轴转换为伪向量,因此在反射下损失不是不变的。这意味着从结构构建框架的方式不受我们所做的精确选择的限制,但只要它们在预测结构和目标结构之间以一致的方式构建,就可以有所不同。

def frame_aligned_point_error(
pred_frames: r3.Rigids, # shape (num_frames)
target_frames: r3.Rigids, # shape (num_frames)
frames_mask: jnp.ndarray, # shape (num_frames)
pred_positions: r3.Vecs, # shape (num_positions)
target_positions: r3.Vecs, # shape (num_positions)
positions_mask: jnp.ndarray, # shape (num_positions)
length_scale: float,
l1_clamp_distance: Optional[float] = None,
epsilon=1e-4) -> jnp.ndarray: # shape ()
assert pred_frames.rot.xx.ndim == 1
assert target_frames.rot.xx.ndim == 1
assert frames_mask.ndim == 1, frames_mask.ndim
assert pred_positions.x.ndim == 1
assert target_positions.x.ndim == 1
assert positions_mask.ndim == 1
# Compute array of predicted positions in the predicted frames.
# r3.Vecs (num_frames, num_positions)
local_pred_pos = r3.rigids_mul_vecs(
jax.tree_map(lambda r: r[:, None], r3.invert_rigids(pred_frames)),
jax.tree_map(lambda x: x[None, :], pred_positions))
# Compute array of target positions in the target frames.
# r3.Vecs (num_frames, num_positions)
local_target_pos = r3.rigids_mul_vecs(
jax.tree_map(lambda r: r[:, None], r3.invert_rigids(target_frames)),
jax.tree_map(lambda x: x[None, :], target_positions))
# Compute errors between the structures.
# jnp.ndarray (num_frames, num_positions)
error_dist = jnp.sqrt(
r3.vecs_squared_distance(local_pred_pos, local_target_pos)
+ epsilon)
if l1_clamp_distance:
error_dist = jnp.clip(error_dist, 0, l1_clamp_distance)
normed_error = error_dist / length_scale
normed_error *= jnp.expand_dims(frames_mask, axis=-1)
normed_error *= jnp.expand_dims(positions_mask, axis=-2)
normalization_factor = (
jnp.sum(frames_mask, axis=-1) *
jnp.sum(positions_mask, axis=-1))
return (jnp.sum(normed_error, axis=(-2, -1)) /
(epsilon + normalization_factor))
AlphaFold 的手性特性及其损失
在本节中,我们将详细查看全局反射下各个组件的变换属性

在这种全局反射下,帧的坐标也以非平凡的方式变化。简单的代数表明

其中旋转矩阵
R
i
R_i
Ri的非平凡变换来自于算法21中的叉乘。旋转矩阵的非平凡变换也意味着局部点KaTeX parse error: Unexpected end of input in a macro argument, expected '}' at position 15: T^{-1}_i \dot \̲v̲e̲c̲{x}_j在反射下不是不变的。全局反射的作用是只反射局部坐标KaTeX parse error: Unexpected end of input in a macro argument, expected '}' at position 15: T^{-1}_i \dot \̲v̲e̲c̲{x}_j的z分量。

在下面,我们用大罗马字母表示一组框架和点,例如
X
=
(
{
x
⃗
i
}
,
{
T
j
}
)
X=(\{\vec{x}_i\},\{T_j\})
X=({xi},{Tj})
特别地,这意味着FAPE和IPA都可以区分蛋白质的全局反射,假设刚性帧与算法21的底层点相关,例如

这在退化情况之外是一个很大的正值。对于FAPE可以区分手性的更一般的证明,而不管框架是如何构造的,请参阅下一节
AlphaFold中还有其他手性来源。原子位置是根据主骨架和预测的χ角组合计算的,这个过程总是会生成一个左手分子,因为它使用了χ角之外的理想值(即CB总是在左手位置)。AlphaFold几乎可以完全生成主链原子的手性对,但它不能构建侧链的手性对。该模型对χ角值的损失较小,且这些值在反射下不是不变的。

为了测试FAPE手性的重要性,我们使用dRMSD损失代替FAPE训练了一个模型,我们在图9中显示了CASP14集上的结果。这里我们可以看到lDDT-Cα的性能仍然很好,我们注意到lDDT-Cα是一个不能区分相反手性分子的规则。
然而,如果用dRMSD损失进行训练,GDT显示出双峰分布,其中两种模式之一仅比基线AlphaFold略差,而另一种模式具有非常低的精度(Suppl。图9)。这表明第二模式是由反手性分子组成的。为了测试这一点,我们计算了GDT,这是用于计算GDT的结构的镜面反射的GDT。这些镜像结构也显示了GDT值的双峰性。最后,取结构和其镜像的GDT的最大值产生一致的高GDT。这证实了使用dRMSD损耗训练的AlphaFold经常产生镜像结构,而FAPE是确保预测结构正确手性的主要组成部分。
FAPE(X,Y) = 0 的配置
为了理解实现零FAPE损失的点,我们将引入一个类似rmsd的辅助度量,它只对点而不是帧起作用

T
T
T是恰当的刚性变换。然后,我们可以显示FAPE的下界,而不考虑帧的值
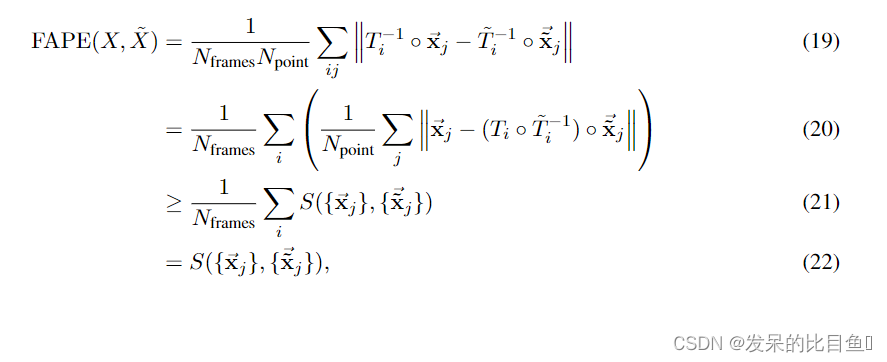
因为S函数在所有固有刚变换中使括号内的量最小化
T
i
T
⃗
i
−
1
T_i\vec{T}_i^{-1}
TiTi−1是一个固有刚变换。这个不等式简单地表明,在相同的距离函数下,所有局部帧的平均点误差不小于最佳单全局对齐的点误差。
如果 R M S D ( { x ⃗ i } , { x ⃗ ^ i } ) = 0 RMSD(\{\vec{x}_i\}, \{\hat{\vec{x}}_i\}) = 0 RMSD({xi},{x^i})=0,则值 S ( { x ⃗ i } , { x ⃗ ^ i } ) S(\{\vec{x}_i\}, \{\hat{\vec{x}}_i\}) S({xi},{x^i})为零,这表明只有当 R M S D ( { x ⃗ i } , { y ⃗ ^ i } ) = 0 RMSD(\{\vec{x}_i\}, \{\hat{\vec{y}}_i\}) = 0 RMSD({xi},{y^i})=0时, F A P E ( X , X ⃗ ) = 0 FAPE(X, \vec{X}) = 0 FAPE(X,X)=0才有可能。我们可以将 F A P E ( X , X ⃗ ) = 0 FAPE(X, \vec{X}) = 0 FAPE(X,X)=0的所有对描述为 R M S D ( { x ⃗ i } , { y ⃗ ^ i } ) = 0 RMSD(\{\vec{x}_i\}, \{\hat{\vec{y}}_i\}) = 0 RMSD({xi},{y^i})=0且KaTeX parse error: Double superscript at position 6: T^{i}^̲{-1}\dot \vec{T…是实现零RMSD的刚性运动的所有对。 特别是,如果点集是非简并的,FAPE损失总是具有非零值手性对,而不管框架是如何构造的。
FAPE 的度量属性
在本节中,我们将展示理想化的FAPE(即使用 ϵ \epsilon ϵ= 0)具有伪度量的所有属性,这是拓扑学中使用的广义距离函数。为了减少杂波,证明没有截断,但当 ∣ ∣ ∙ ∣ ∣ ||\bullet|| ∣∣∙∣∣被视为裁剪向量范数时,证明同样有效。对数学细节不感兴趣的读者可以跳过这一节。对数学细节不感兴趣的读者可以跳过这一节。
由定义方程可以简单地证明: 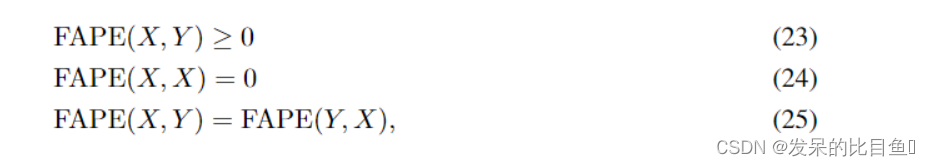
剩下的伪度量性质就是三角不等式了
F
A
P
E
(
X
,
Z
)
≤
F
A
P
E
(
X
,
Y
)
+
F
A
P
E
(
Y
,
Z
)
FAPE(X,Z) ≤FAPE(X,Y )+FAPE(Y,Z)
FAPE(X,Z)≤FAPE(X,Y)+FAPE(Y,Z)。
三角形不等式的证明如下,使用\tildes和overbar来指定不同的配置。
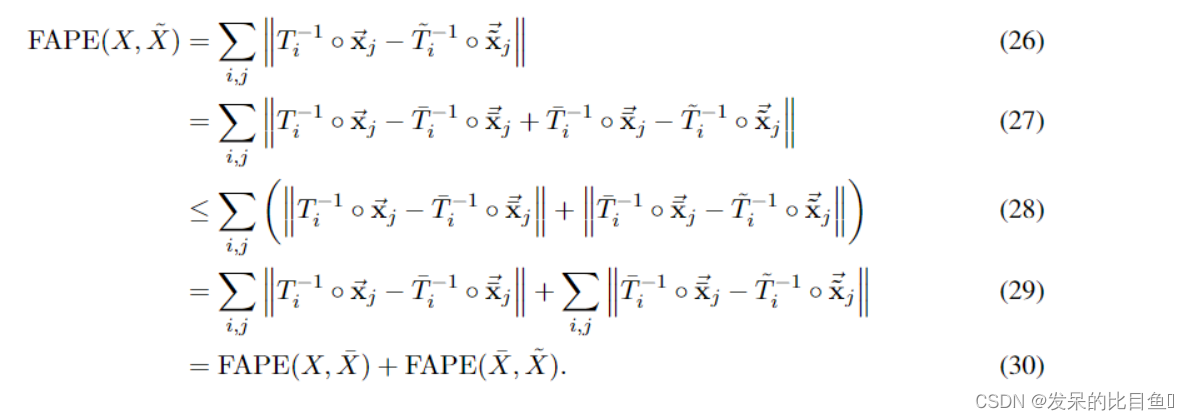
模型置信度预测(pLDDT)
我们的模型通过预测每个残基lDDT-Cα分数来提供内在模型精度估计。我们称这个置信度为 pLDDT. 算法29通过从结构模块(算法20第30行)中获得最终的单一表示来计算它,并将其投射到50个bin(每个bin覆盖2 lDDT- Cα范围)
class PredictedLDDTHead(hk.Module):
def __init__(self, config, global_config, name='predicted_lddt_head'):
super().__init__(name=name)
self.config = config
self.global_config = global_config
def __call__(self, representations, batch, is_training):
act = representations['structure_module'] #[N_res, 384]
act = hk.LayerNorm(
axis=[-1],
create_scale=True,
create_offset=True,
name='input_layer_norm')(
act)
act = common_modules.Linear(
self.config.num_channels,
initializer='relu',
name='act_0')(
act)
act = jax.nn.relu(act)
act = common_modules.Linear(
self.config.num_channels,
initializer='relu',
name='act_1')(
act)
act = jax.nn.relu(act)
logits = common_modules.Linear(
self.config.num_bins,
initializer=utils.final_init(self.global_config),
name='logits')(
act)#[N_res, 50]
# Shape (batch_size, num_res, num_bins)
return dict(logits=logits)
def loss(self, value, batch):
# Shape (num_res, 37, 3)
pred_all_atom_pos = value['structure_module']['final_atom_positions']
# Shape (num_res, 37, 3)
true_all_atom_pos = batch['all_atom_positions']
# Shape (num_res, 37)
all_atom_mask = batch['all_atom_mask']
# Shape (num_res,)
lddt_ca = lddt.lddt(
# Shape (batch_size, num_res, 3)
predicted_points=pred_all_atom_pos[None, :, 1, :],
# Shape (batch_size, num_res, 3)
true_points=true_all_atom_pos[None, :, 1, :],
# Shape (batch_size, num_res, 1)
true_points_mask=all_atom_mask[None, :, 1:2].astype(jnp.float32),
cutoff=15.,
per_residue=True)
lddt_ca = jax.lax.stop_gradient(lddt_ca)
num_bins = self.config.num_bins
bin_index = jnp.floor(lddt_ca * num_bins).astype(jnp.int32)
# protect against out of range for lddt_ca == 1
bin_index = jnp.minimum(bin_index, num_bins - 1)
lddt_ca_one_hot = jax.nn.one_hot(bin_index, num_classes=num_bins)
# Shape (num_res, num_channel)
logits = value['predicted_lddt']['logits']
errors = softmax_cross_entropy(labels=lddt_ca_one_hot, logits=logits)
# Shape (num_res,)
mask_ca = all_atom_mask[:, residue_constants.atom_order['CA']]
mask_ca = mask_ca.astype(jnp.float32)
loss = jnp.sum(errors * mask_ca) / (jnp.sum(mask_ca) + 1e-8)
if self.config.filter_by_resolution:
# NMR & distillation have resolution = 0
loss *= ((batch['resolution'] >= self.config.min_resolution)
& (batch['resolution'] <= self.config.max_resolution)).astype(
jnp.float32)
output = {'loss': loss}
return output
TM-score 预测
上面的pLDDT头部预测了lDDT-Cα的值,这是一个成对操作的局部误差度量,但在设计上对使用单个全局旋转和平移可以对齐的残差比例不敏感。这可能不利于评估模型是否对大型链的整体域置信度。在本节中,我们将开发一个全局叠加度量TM-score的预测器
我们表示基本真实结构的Cα原子
X
t
r
u
e
=
{
X
⃗
j
t
r
u
e
}
X^{true}=\{\vec{X}^{true}_j\}
Xtrue={Xjtrue},它的骨干框架
{
T
i
t
r
u
e
}
\{T_i^{true}\}
{Titrue}, 预测结构的Cα原子通过
X
=
{
x
}
⃗
j
X = \vec{\{x\}}_j
X={x}j以及相应的骨干帧
{
T
i
}
\{T_i\}
{Ti}。设残差个数为
N
r
e
s
N_{res}
Nres,并假设所有残差都在基本真值中求解.表示
T
a
l
i
g
n
=
(
R
a
l
i
g
n
,
t
⃗
a
l
i
g
n
)
T^{align}=(R^{align}, \vec{t}^{align})
Talign=(Ralign,talign)为任意刚性变换,TM-score定义为

在上面,我们用
N
r
e
s
N_{res}
Nres但序列对齐
(
T
i
T
i
t
r
u
e
−
1
)
(T^i{T^{true}_i}^{-1})
(TiTitrue−1)的离散最大集替换了所有可能对齐的连续最大值。这显然是原始极大值的下界,当全局叠加与任何剩余的主干完全对齐时,这个上界变得很紧。然后,利用刚性变换
T
i
T_i
Ti对矢量差进行对称化。请注意,公式36中的表达式与FAPE密切相关,除了f函数略有不同,FAPE具有平均值而不是最大值(即FAPE考虑所有1-残差对齐而不是仅考虑最佳对齐)。
接下来,我们假设正确的结构
X
t
r
u
e
X^{true}
Xtrue是未知的,并且我们有一个可能结构的分布,我们为我们的预测
X
X
X寻求
T
M
−
s
c
o
r
e
TM-score
TM−score的期望值

在最后一行中,我们将最大期望替换为期望的最大值(由于Jensen不等式的下界),并使用期望的线性。
成对矩阵 e i j = ∣ ∣ T i − 1 ∘ x ⃗ j − T i t r u e − 1 ∘ x ⃗ j t r u e ∣ ∣ e_{ij}=||T_i^{-1}\circ \vec{x}_j-{T_i^{true}}^{-1}\circ \vec{x}_j^{true}|| eij=∣∣Ti−1∘xj−Titrue−1∘xjtrue∣∣是一个非对称矩阵,它捕获了残基 j j j的Cα原子位置的误差,当预测结构和真实结构使用残基 i i i的主框架对齐时。神经网络可以很容易地预测其元素的概率分布。为此,我们将 e i j e_{ij} eij分配到64bin中,覆盖0至31.5Å的范围,每个bin宽度0.5Å。在训练期间,最终的bin还捕获任何较大的错误。我们将 e i j e_{ij} eij计算为对表示 z i j z_{ij} zij的线性投影,然后是一个softmax。我们对CASP模型进行了微调,以使用平均分类交叉熵损失额外预测 e i j e_{ij} eij,权重为0.1(也尝试了0.01和1.0的权重,但这些权重分别降低了pTM精度或结构预测精度)。就像pLDDT预测一样,我们只在分辨率在0.1 Å和3.0 Å之间的non-NMR 示例上训练这个预测模块。他花了大约16个小时进行微调,处理了 3 ⋅ 1 0 5 3\cdot 10^5 3⋅105个样本。
使用
e
i
j
e_{ij}
eij,我们将TM-score近似为

其中期望是由
e
i
j
e_{ij}
eij定义的概率分布。我们发现pTM是一个真实tm得分的精确预测器,这是意料之中的,因为在推导pTM时使用的两个近似都是下界。
请注意,给定
e
i
j
e_{ij}
eij的全链预测,我们可以通过简单地限制所考虑的残基的范围来获得残基D的任何子集(例如特定的域)的TM-score预测

其中
∣
D
∣
|D|
∣D∣是集合
D
D
D中的残差数。通过小的修改,可以从
e
i
j
e_{ij}
eij矩阵中推导出一个类似的表达式来估计GDT、FAPE或RMSD,尽管我们不进一步研究这个问题。此外,
f
(
e
i
j
)
f(e_{ij})
f(eij)的期望值的二维图像可以很好地显示结构中的自信域填充。
直方图预测
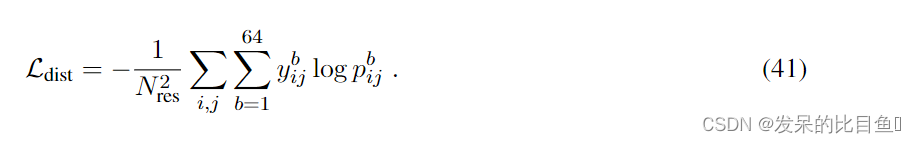
我们将对称的对表示
(
z
i
j
+
z
j
i
)
(z_{ij}+z_{ji})
(zij+zji)线性投影到64个距离bin中,并用softmax获得bin概率
p
i
j
b
p_{ij}^b
pijb。bin的范围从2 Å到22 Å;它们与最后一个箱子的距离相等,也包括距离残基对。对于预测目标
y
i
j
b
y_{ij}^b
yijb,我们使用单热编码的bin残基距离,这是从除甘氨酸以外的所有氨基酸的真实β碳位置计算出来的,而甘氨酸则使用α碳。

屏蔽 MSA 预测
类似于常见的掩码语言建模目标,我们使用最终的MSA表示来重建先前被掩码的MSA值.我们考虑23类,其中包括20种常见氨基酸类型,一种未知类型,一种间隙令牌和一种掩码令牌。MSA表示
{
m
s
i
}
\{m_{si}\}
{msi}被线性投影到输出类中,通过一个softmax,并使用交叉熵损失进行评分
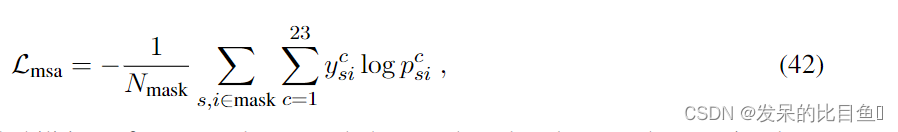
其中
p
s
i
c
p^c_{si}
psic是预测的类别概率,
y
s
i
c
y^c_{si}
ysic是单热编码的真实值,并对屏蔽位置进行平均。
实验解决”预测
该模型包含一个头部,用于预测原子是否在实验中以高分辨率结构分解。这个头部的输入是Evoformer Stack产生的单个表示
{
s
i
}
\{si\}
{si}. 单个表示用一个线性层和一个sigmoid投影到具有
i
∈
[
1
,
.
.
.
,
N
r
e
s
]
i \in [1, ..., N_{res}]
i∈[1,...,Nres]和
a
∈
S
a
t
o
m
n
a
m
e
s
a \in S_{atom names}
a∈Satomnames的原子概率
{
p
i
e
x
p
r
e
s
o
l
v
e
d
,
a
}
\{p^{exp resolved,a}_i\}
{piexpresolved,a}。我们在使用标准交叉熵对高分辨率x射线晶体和cryo-EM 结构(分辨率优于3Å)进行微调时训练这个头部。

其中
y
i
a
∈
{
0
,
1
}
y^a_i \in \{0,1\}
yia∈{0,1}表示基本真值,即如果残基
i
i
i中的原子
a
a
a在实验中被解析。
结构违规
从独立的骨架框架和扭转角(图3e)构建原子坐标(子小节1.8.4)为大多数原子键产生理想的键长和键角,但残基键间(肽键)的几何结构和避免原子碰撞需要学习。我们引入额外的损失来惩罚这些结构违规,以确保这些约束无处不在,也就是在没有真实原子坐标可用的区域。我们以一种无损失结构将通过lDDT度量的立体化学质量检查的方式构建了损失。
损耗使用flat-bottom L1 损失,惩罚超过一定容差阈值τ的违规行为。键长违反损失计算为

其中
l
p
r
e
d
i
l^i_{pred}
lpredi为预测结构中的键长,
l
l
i
t
i
l^i_{lit}
lliti为该键长的文献值。我们将公差τ设为12
σ
l
i
t
σ_{lit}
σlit,其中
σ
l
i
t
σ_{lit}
σlit是该键长的文献标准差。我们选择了因子12以确保所生产的键长通过立体化学质量检查lDDT度量,该度量在默认情况下也使用12的公差因子。
键角违反损失使用从键的单位向量点积计算出的角度余弦
c
o
s
α
v
1
⃗
^
T
v
2
⃗
^
cos \alpha \hat{\vec{v_1}}^T \hat{\vec{v_2}}
cosαv1^Tv2^,并在偏差上flat-bottom L1损失

其中
α
p
r
e
d
i
α^i_{pred}
αpredi为预测结构中的键角,
α
l
i
t
i
α^i_{lit}
αliti为该键角的文献值。角是这个结构中所有键角的个数。
N
a
n
g
l
e
s
N_{angles}
Nangles是这个结构中所有键角的个数。计算公差
τ
\tau
τ时,flat bottom从-12到12倍的文献标准偏差的键角。
碰撞损失使用单侧flat-bottom电位,只惩罚过短的距离

其中
d
p
r
e
d
i
d^i_{pred}
dpredi是预测结构中两个非键原子的距离,
d
l
i
t
i
d^i_{lit}
dliti是这两个原子根据其文献范德华半径的碰撞距离。
N
n
b
p
a
i
r
N_{nbpair}
Nnbpair是该结构中所有未成键原子对的数目。容差τ设置为1.5 Å。
所有这些损失一起构成了违规损失

我们只在微调训练阶段应用这种违反损失。在早期训练中打开它会导致训练动态的强烈不稳定性。


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










