Effective Hybrid Genetic Algorithm (EHGA)

STEP 1:输入图像
读取图像
X
(
i
,
j
)
X(i,j)
X(i,j),其中
i
,
j
∈
[
0
,
255
]
i,j\in[0,255]
i,j∈[0,255]
STEP 2:初始化初始种群
通过对图像
X
(
i
,
j
)
X(i,j)
X(i,j)执行DAMF,AWMF和FNAFSM得到由三个个体组成的初始种群:
D
=
{
X
D
A
M
F
,
X
A
W
M
F
,
X
N
A
F
S
M
}
D=\{X_{DAMF},X_{AWMF},X_{NAFSM}\}
D={XDAMF,XAWMF,XNAFSM}。接着,从集合
D
D
D随机选取两个个体,并对这两个个体采用像素合并:在两个选定的个体之间应用逐像素随机选择,得到一个新的个体并加入种群中,直到种群个数达到
N
p
N_p
Np
STEP 3:评价图像
适应值
fitness
(
F
)
=
λ
∣
F
−
X
∣
+
(
∑
Ω
1
+
β
2
∣
∇
F
∣
2
)
\text { fitness }(F)=\lambda|F-X|+\left(\sum_{\Omega} \sqrt{1+\beta^{2}|\nabla F|^{2}}\right)
fitness (F)=λ∣F−X∣+(Ω∑1+β2∣∇F∣2)
Ω
\Omega
Ω:图像的所有像素点组成的集合。
F
F
F:被评估的图像
X
X
X:噪声图
∇
F
\nabla F
∇F:全变分损失,相邻像素的差值,值越小,图像越平滑
∇
F
i
j
=
∣
F
i
+
1
,
j
−
F
i
,
j
∣
+
∣
F
i
,
j
+
1
−
F
i
,
j
∣
\nabla F_{ij}=\left|F_{i+1, j}-F_{i, j}\right|+\left|F_{i, j+1}-F_{i, j}\right|
∇Fij=∣Fi+1,j−Fi,j∣+∣Fi,j+1−Fi,j∣
β
,
λ
\beta,\lambda
β,λ:权衡因子
∣ F − X ∣ |F-X| ∣F−X∣:逼近项,确保原始图像和被评估的图像之间有一定的保真度
适应函数的目的是平滑噪声,同时保持与原始图像的保真项。
STEP 4:新个体
使用轮盘赌算法选择一对初始个体(parent)。
交叉:从四种交叉操作中随机选择一种。
(i) Single point
从以下两个操作中随机选取一个:
- One point column: 随机选取一列,列的两边分别是parent两个个体对应的像素。

- One point row: 与One point column类似的。

(ii) Two point
从以下两个操作中随机选取一个:
- Two point column:随机选取两列,列之间与列之外的像素分别用parent的两个个体的像素填充。

- Two-point row:与 Two point column类似的。

(iii) Cross grid
随机选取一行和一列,将图像分为四块,两个对角部分分别用parent的两个个体对应的像素填充。

(iv) Pixel-by-pixel random
逐像素随机从parent的两个个体中选取像素。
变异:对每一个新个体以
N
m
N_m
Nm的概率进行变异,随机应用四种变异操作中的一个。
(i) NAFSM.
(ii) DAMF.
(iii) AWMF
(iv) Random:保持
X
(
i
,
j
)
X(i,j)
X(i,j)的非噪声点不变的前提下,逐像素乘一个[0.8,1.2]的随机数。
直到新的种群个体数达到 N c ⋅ N p N_c\cdot N_p Nc⋅Np。
STEP 5:评估新种群
计算新种群的适应值
STEP 6:
根据适应值对种群(初始种群和新种群)进行排序,得到
N
p
N_p
Np个最佳个体,并把它们作为下次迭代的初始种群。
STEP7:结束
步骤4到6重复执行
i
t
e
r
m
a
x
iter_{max}
itermax次,最后一代中的最佳个体就是去噪的结果图
# -*- coding:utf-8 -*-
'''
@paper:Effective hybrid genetic algorithm for removing salt and pepper noise
'''
import numpy as np
from awmf import Awmf
from damf import Damf
from nafsmf import Nafsmf
# Effective hybrid genetic algorithm
class Ehga(object):
def __init__(self, pop_num, crossover_num, mutation_rate, lambda_, beta, iter_max):
self.dam_filter = Damf()
self.awm_filter = Awmf(h=1, w_max=39)
self.nafsm_filter = Nafsmf(h=1, s_max=3, T1=10, T2=30)
self.pop_num = pop_num
self.iter_max = iter_max
self.crossover_num = crossover_num
self.mutation_rate = mutation_rate
self.m = None # 灰度图大小(m,n)
self.n = None
self.lambda_ = lambda_ # balancing parameters of fidelity term
self.beta = beta # balancing parameters of total variation regularising term
def init_population(self, img_noise):
X_damf = self.dam_filter.process_image(img_noise)
X_awmf = self.awm_filter.process_image(img_noise)
X_nafsm = self.nafsm_filter.process_image(img_noise)
init_pop = np.zeros((self.pop_num, self.m, self.n))
init_three = np.stack((X_damf, X_awmf, X_nafsm))
init_pop[0:3] = init_three.copy()
for n in range(3, self.pop_num):
index = np.random.choice(3, 2, replace=False)
X_pair = init_three[index]
new_individual = np.zeros((self.m, self.n))
for i in range(self.m):
for j in range(self.n):
new_individual[i, j] = X_pair[np.random.randint(2)][i, j]
init_pop[n] = new_individual
return init_pop
def cal_fit_value(self, X, F):
'''
:param X: 噪声图像
:param F: 滤波处理后的图像
:return: 适应值
'''
fitness = []
for F_i in F:
fidelity_term = self.lambda_ * np.sum(np.abs(F_i - X))
regularising_term = 0
for i in range(self.m - 1):
for j in range(self.n - 1):
nabla_F = np.abs(F_i[i + 1, j] - F_i[i, j]) + np.abs(F_i[i, j + 1] - F_i[i, j])
regularising_term += np.sqrt(1 + self.beta ** 2 * nabla_F ** 2)
fitness_i = fidelity_term + regularising_term
fitness.append(fitness_i)
return fitness
def selection(self, pop, fit_value):
'''从种群中选择适应值最大的两个个体'''
# 求适应值最小值,所以求负值
fit_value = -np.array(fit_value)
# 计算适应值之和
total_fit = np.sum(fit_value)
# 使概率总和为1
p_fit_value = fit_value / total_fit
# 概率求和排序
p_fit_value = np.cumsum(p_fit_value)
random_p = np.sort(np.random.random(2))
# 轮盘赌选择法
fit_index = 0
new_index = 0
parent = np.zeros((2, self.m, self.n))
while new_index < 2:
# 如果这个概率大于随机出来的那个概率,就选这个
if p_fit_value[fit_index] > random_p[new_index]:
parent[new_index] = pop[fit_index]
new_index = new_index + 1
else:
fit_index = fit_index + 1
return parent
def crossover(self, parent):
'''
crossover_type:
0:One point column
1:One point row
2:Two point column
3:Two point row
4:Cross grid
5:Pixel-by-pixel random
'''
crossover_type = np.random.randint(6)
if crossover_type == 0:
n = np.random.randint(self.n)
new_individual = parent[0].copy()
new_individual[:, n:] = parent[1][:, n:]
elif crossover_type == 1:
m = np.random.randint(self.m)
new_individual = parent[0].copy()
new_individual[m:, :] = parent[1][m:, :]
elif crossover_type == 2:
n = np.sort(np.random.randint(0, self.n, 2))
new_individual = parent[0].copy()
new_individual[:, 0:n[0]] = parent[1][:, 0:n[0]]
new_individual[:, n[1]:] = parent[1][:, n[1]:]
elif crossover_type == 3:
m = np.sort(np.random.randint(0, self.m, 2))
new_individual = parent[0].copy()
new_individual[0:m[0], :] = parent[1][0:m[0], :]
new_individual[m[1]:, :] = parent[1][m[1]:, :]
elif crossover_type == 4:
n = np.random.randint(self.n)
m = np.random.randint(self.m)
new_individual = parent[0].copy()
new_individual[0:m, 0:n] = parent[1][0:m, 0:n]
new_individual[m:, n:] = parent[1][m:, n:]
else:
new_individual = np.zeros((self.m, self.n))
for i in range(self.m):
for j in range(self.n):
if np.random.random() <= 0.5:
new_individual[i, j] = parent[0][i, j]
else:
new_individual[i, j] = parent[1][i, j]
return new_individual
def mutation(self, new_individual, img_noise):
'''
mutation_type:
0:NAFSM
1:DAMF
2:AWMF
3:Random
'''
if np.random.random(1) < self.mutation_rate:
mutation_type = np.random.randint(4)
if mutation_type == 0:
new_individual = self.nafsm_filter.process_image(img_noise)
elif mutation_type == 1:
new_individual = self.dam_filter.process_image(img_noise)
elif mutation_type == 2:
new_individual = self.awm_filter.process_image(img_noise)
else:
tem = np.random.random(size=(self.m, self.n)) * 0.4 + 0.8
new_individual = new_individual * tem
mask = (img_noise != 0) | (img_noise != 255)
new_individual[mask] = img_noise[mask]
return new_individual
def process_image(self, img_noise):
self.m, self.n = np.shape(img_noise)
pop = self.init_population(img_noise)
for iter in range(self.iter_max):
print('iter:', iter)
fit_value = self.cal_fit_value(img_noise, pop)
pop_new = np.zeros((self.pop_num + self.crossover_num, self.m, self.n))
pop_new[:self.pop_num] = pop
for i in range(self.crossover_num):
print(i)
parent = self.selection(pop, fit_value)
new_individual = self.crossover(parent)
new_individual = self.mutation(new_individual, img_noise)
pop_new[self.pop_num + i] = new_individual
fit_value = self.cal_fit_value(img_noise, pop_new)
sort_fit_value = np.argsort(fit_value)
pop_new = pop_new[sort_fit_value[0:self.pop_num]]
return pop_new[0].astype(int)

| Noise level | Noise image | DAMF | NAFSM | AWMF | EHGA |
|---|---|---|---|---|---|
| 90% |  |  |  |  |  |
| 95% |  | 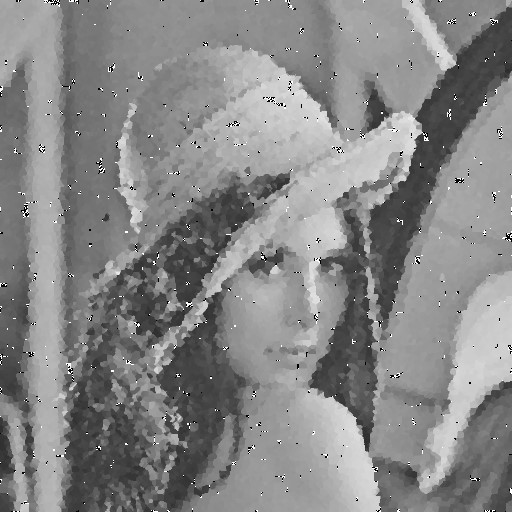 | 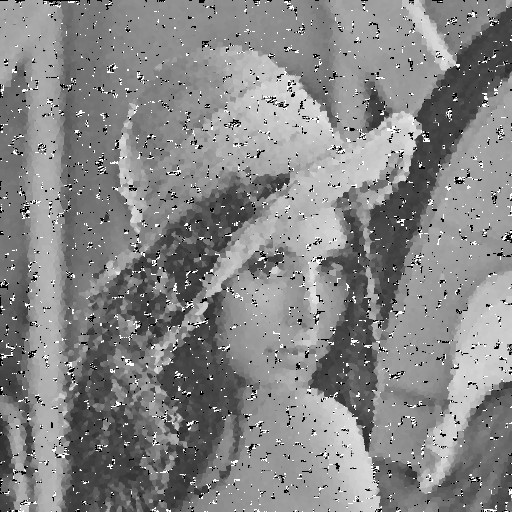 |  |  |
| 99% |  | 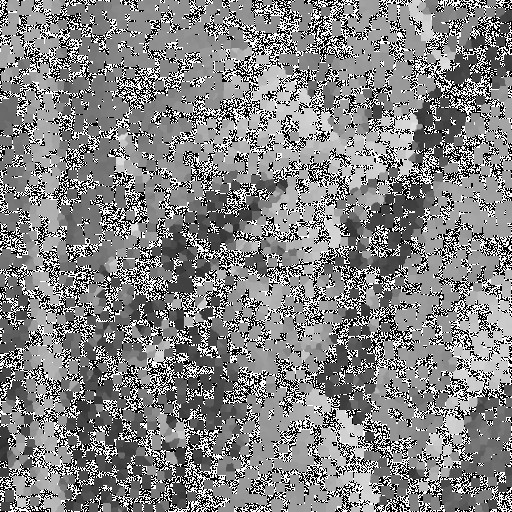 |  |  |  |























 1130
1130











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










