miRNA测序数据生信分析——第二讲,数据库下载整理
miRNA测序数据生信分析——第二讲,数据库下载整理
有六个Rfam、miRBase、miRTarBase、miRDB、TargetScan和TargetMiner
重点是前4个。
这一部分:在对数据库整理时,涉及到三个软件(infernal、blast、seqkit)、2个脚本的撰写以及对NCBI的Refseq和GENE数据库的使用
1. Rfam数据库
1.1 Rfam数据库——简单概述
一个非编码RNA的整合数据库,可用来识别鉴定各种类型的ncRNA。提供的是ncRNA在基因组上的基因序列,而非转录成熟ncRNA的序列。
官方网站:https://rfam.xfam.org/
指导手册:http://eddylab.org/infernal/Userguide.pdf
下载Rfam数据库文件地址:https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/
最新版:2022-11-02
Rfam数据库中ncRNA类型分三大类:Cis-reg和Gene和Intron。这三大类又有子类。

1.2 Rfam数据库——下载整理:两种情况
Rfam数据库使用的两种情况:
①用于注释基因组上的ncRNA基因序列
②用于注释ncRNA/sRNA测序中的tRNA和rRNA序列(miRNA、lncRNA转录会加工成mature miRNA,因此不能注释)
两种情况分析时准备的Rfam数据库文件和整理方法是不同的,这里分别阐述。
1.2.1 用于注释基因组上的ncRNA基因序列
需要的Rfam数据库文件:Rfam.clanin、Rfam.cm和family.txt
需要的软件:infernal
#软件infernal
#官网:http://eddylab.org/infernal/,下载最新安装包
#安装
cd /home/zhaohuiyao/Biosoft/ncRNA_soft
wget http://eddylab.org/infernal/infernal-1.1.4-linux-intel-gcc.tar.gz
tar -zxvf ./infernal-1.1.4-linux-intel-gcc.tar.gz
#已是编译好的版本。可执行文件位置:/home/zhaohuiyao/Biosoft/ncRNA_soft/infernal-1.1.4-linux-intel-gcc/binaries/
cd /home/zhaohuiyao/Database/Rfam
wget https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.clanin
wget https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.cm.gz
wget https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/database_files/family.txt.gz
gunzip ./Rfam.cm.gz
gunzip ./family.txt.gz
#建立数据库索引
/home/zhaohuiyao/Biosoft/ncRNA_soft/infernal-1.1.4-linux-intel-gcc/binaries/cmpress /home/zhaohuiyao/Database/Rfam/Rfam.cm
1.2.2 用于注释ncRNA/sRNA测序中的tRNA和rRNA序列
需要的Rfam数据库文件:Rfam.fa、Rfam.full_region和family.txt
需要的软件:blast
需要自己写一个脚本:Deal_Rfam_full_region.py
cd /home/zhaohuiyao/Database/Rfam
wget https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/fasta_files/Rfam.fa.gz
wget https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.full_region.gz
wget https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/database_files/family.txt.gz
gunzip ./Rfam.fa.gz
gunzip ./Rfam.full_region.gz
gunzip ./family.txt.gz
#特别注意:Rfam.fa(保存ncRNA序列)和Rfam.full_region(ncRNA属于哪个RF家族),两者信息数量不对等,需要手动修正。
#最终拿到两者信息完美匹配的Rfam.fa和Rfam.full_region
python3 ./Deal_Rfam_full_region.py -i ./Rfam.fa -I ./Rfam.full_region -o ./

#结果文件Rfam.full_region.tmp和Rfam.fa.tmp替换原来的文件
mv Rfam.full_region.tmp Rfam.full_region
mv Rfam.fa.tmp Rfam.fa #最终的Rfam.fa长度从19~10656bp
/home/zhaohuiyao/Biosoft/general/seqkit stat -j 16 -T ./Rfam.fa

#建立数据库索引
/home/zhaohuiyao/Biosoft/general/ncbi-blast-2.10.0+/bin/makeblastdb -in ./Rfam.fa -dbtype nucl -out ./Rfam
2. miRBase数据库
2.1 miRBase数据库——简单概述
一个提供包括miRNA序列数据、注释、预测基因靶标等信息的全方位数据库,是存储miRNA信息最主要的公共数据库之一。
官方网址:https://www.mirbase.org/
下载地址:https://www.mirbase.org/ftp/CURRENT/
最新版:2018-12-01
2.2 miRBase数据库——下载整理
cd /home/zhaohuiyao/Database/miRBase
wget https://www.mirbase.org/ftp/CURRENT/organisms.txt.gz
wget https://www.mirbase.org/ftp/CURRENT/mature.fa.gz
gunzip ./organisms.txt.gz
grep -v "#" ./organisms.txt | wc -l #285个物种
gunzip ./mature.fa.gz
3. miRTarBase数据库
3.1 miRTarBase数据库——简单概述
一个保存miRNA靶基因信息的数据库,其中存在的MITs是经过实验验证的,包括reporter assay, western blot, microarray and next-generation sequencing experiments等实验方法。因此可信度高。最新版v9.0,时间2021-09,依据miRBase v22。
官方网址:https://mirtarbase.cuhk.edu.cn/~miRTarBase/miRTarBase_2022/php/index.php
包括物种:37种。有单独物种的文件(例:hsa_MTI.xlsx),也有全部物种的文件miRTarBase_MTI.xlsx。
3.2 miRTarBase数据库——下载整理
对miRTarBase_MTI.xlsx进行提取,仅保留三列。miRNA,Target Gene,Target Gene (Entrez ID)。并删除重复项。(重复原因,不同的实验方法对同一靶向关系验证),拿到最终文件miRTarBase_MTI.txt
这个处理可以在Windows下用excel完成,①直接删列;②将miRNA与Target Gene两列合并(=A1&“_”&B1),利用合并后的字符串进行去重复。
但是没有想到,文件里面有迷惑的地方。令人不解

miRNA的名称与物种之间不一致,这种情况,我就忽略了
cd /home/zhaohuiyao/Database/miRTarBase
#文件miRTarBase_MTI.txt
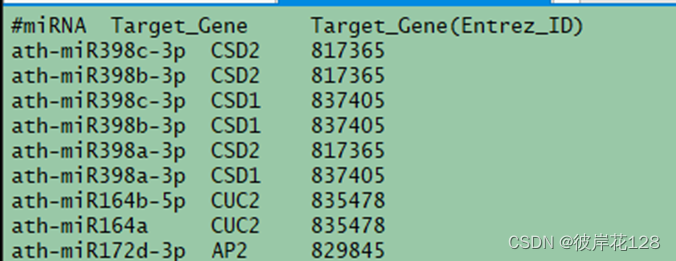
grep -v “#” ./miRTarBase_MTI.txt | awk ‘{split($0,arr,“-”);print arr[1]}’ | sort | uniq > miRTarBase.organism
wc -l miRTarBase.organism #26个物种
4. miRDB数据库
4.1 miRDB数据库——简单概述
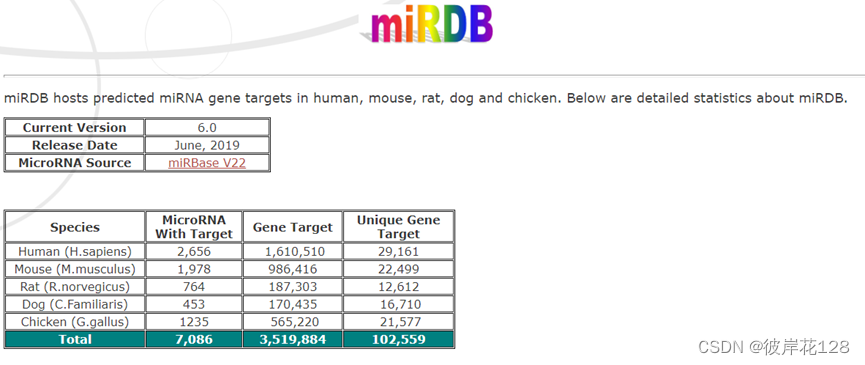
一个保存miRNA靶基因信息的数据库。最新版v6.0,时间2019-06,依据miRBase v22。
六大模块:Target Search、Target Expression、Target Ontology、Target Mining、Custom Prediction、FuncMir Collection
官方网址:https://mirdb.org/
仅针对5个物种的靶基因信息进行分析。(人类,小鼠,褐家鼠,家狗,原鸡)
4.2 miRDB数据库——下载整理
cd /home/zhaohuiyao/Database/miRDB
wget https://mirdb.org/download/miRDB_v6.0_prediction_result.txt.gz
gunzip ./miRDB_v6.0_prediction_result.txt.gz
grep -v “#” ./miRDB_v6.0_prediction_result.txt | awk ‘{split($0,arr,“-”);print arr[1]}’ | sort | uniq > miRDB.organism #5个物种
出现问题:
举例如下
以cfa-miR-1185为例,在miRDB官网进行检索,点击Target Search,检索靶标基因,结果如下
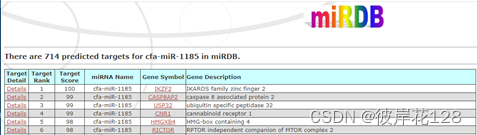
#有714个靶基因,且有靶基因的Gene Symbol
但是呀,万万没有想到!!!在下载的数据库文件miRDB_v6.0_prediction_result.txt文件中进行检索,结果有2287个
grep -c “cfa-miR-1185” ./miRDB_v6.0_prediction_result.txt 
#并且检索结果中是靶mRNA的检索号,而不是Gene Symbol
因此,需要我们自己提供Gene Symbol和靶mRNA检索号的对应关系,对miRDB_v6.0_prediction_result.txt进行整理。这个整理有点复杂
#第一步,下载Gene Entrez ID和mRNA检索号的对应关系文件。依据NCBI的RefSeq数据库
cd /home/zhaohuiyao/Database/RefSeq
wget https://ftp.ncbi.nlm.nih.gov/refseq/release/release-catalog/release218.accession2geneid.gz
gunzip ./release218.accession2geneid.gz
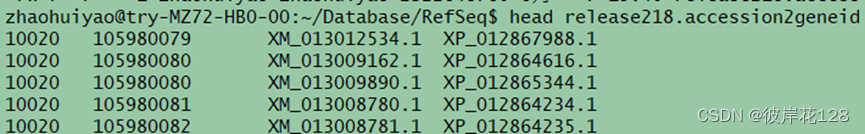
#该文件由四列组成,分别是Taxonomic ID、Entrez GeneID、Transcript accession.version 、Protein accession.version。若第三列转录本检索号由NR或XR开头,表示非编码基因,则第四列的值为na
#第二步,下载Entrez GeneID和Gene Symbol的对应关系文件。依据NCBI的GENE数据库
#可以下载NCBI上所有基因的文件,不分物种
https://ftp.ncbi.nlm.nih.gov/gene/DATA/GENE_INFO/All_Data.gene_info.gz
#但是不划算,我只需要分别下载5个物种的基因文件
#这里以Homo sapiens(hsa)为例
cd /home/zhaohuiyao/Database/miRDB
wget https://ftp.ncbi.nlm.nih.gov/gene/DATA/GENE_INFO/Mammalia/Homo_sapiens.gene_info.gz
gunzip ./Homo_sapiens.gene_info.gz
#第三步,依据第一步和第二步下载的文件,对miRDB_v6.0_prediction_result.txt进行整理,拿到独属于指定物种的关系文件
python3 ./Deal_miRDB.py -i ./miRDB_v6.0_prediction_result.txt -db1 …/RefSeq/release218.accession2geneid -db2 ./Homo_sapiens.gene_info -s hsa -o ./

#结果文件miRDB_v6.0_prediction_result.txt.hsa(3351016)和miRDB_v6.0_prediction_result.txt.hsa.Error(0+24725)
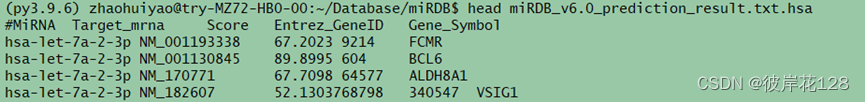

#第四步,分在再完成物种Mus_musculus(mmu)、物种Rattus norvegicus(rno)、物种Canis familiaris(cfa)和物种Gallus gallus(gga)的整理工作
#第五步,将5个物种的结果进行整合。
cat miRDB_v6.0_prediction_result.txt.hsa miRDB_v6.0_prediction_result.txt.mmu miRDB_v6.0_prediction_result.txt.rno miRDB_v6.0_prediction_result.txt.cfa miRDB_v6.0_prediction_result.txt.gga > miRDB_v6.0_prediction_result.txt.final
5. TargetScan数据库
5.1 TargetScan数据库——简单概述
一个保存miRNA靶基因信息的数据库。最新版v7.2,时间2018-03。
官网:https://www.targetscan.org/vert_80/
没有一个完整的数据库,只有不同物种的各自数据库,若想本地进行检索,需要分别下载。
物种有:Human、Mouse、Rat、chimpanzee、Rhesus、Cow、Dog、Opossum、Chicken、Frog、Worm、Fly、Fish等。(但只有Human、Mouse、Worm、Fly、Fish能做本地检索,下载靶标信息文件)
#选择物种和靶基因的名称

6. TargetMiner数据库
6.1 TargetMiner数据库——简单概述
版本2012年,没有更新,且只有人类,不常使用
官网:https://www.isical.ac.in/






















 2901
2901











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








