SRA数据下载(通过EBI-ENA数据库,使用ASpera)
SRA数据下载(通过EBI-ENA数据库,使用ASpera)
这是本人的第一篇文章,欢迎浏览提问,我会在最快时间内答复,谢谢大家。
本文主要是针对新手下载SRA数据过程,我本人已亲自实践有效。按照本文下载SRA数据,需要提前安装Linux系统(常用CentOs或Ubuntu)。需要一定的Linux系统的操作命令认识以及生信知识(包括NCBI数据库检索等)。
需要注意:登录NCBI或EBI-ENA数据库时可能会比较卡或容易失败,那么你可能需要外网登录。
1. SRA简识
1.1 SRA数据
一种储存高通量测序数据格式。高通量测序:即NGS和TGS,通常数据产出在几十到几百MB不等
1.2 各种检索号
PRJNA:study_acession
SRP:secondary_study_accession(DRP,ERP)
SAMN:sample_accession
SRS:secondary_sample_accession(DRS,ERS)
SRX:experiment_accession
SRR:run_accession(DRR,ERR)
SRA:submission_accession
如果你想要更多了解SRA数据,请自行查找其他资料。
2. 举例——SRX5327410数据集
以SRX5327410数据集为例,进行后续的数据检索与下载。
2.1 NCBI数据库——观察SRX5327410数据集
打开NCBI,输入检索号SRX5327410,选择数据库:SRA
该数据集包括39个SRR,是由Pacbio使用Sequel I测序仪进行的三代测序(小分子实时测序,SMRT),文库类型是CCS-13.5kb。
物种:Homo sapiens。

2.2 EBI-ENA数据库——确定需要下载数据集的下载地址
因为EBI-ENA数据库可以直接提供fastq格式测序文件,省去了sra文件转fastq文件的时间
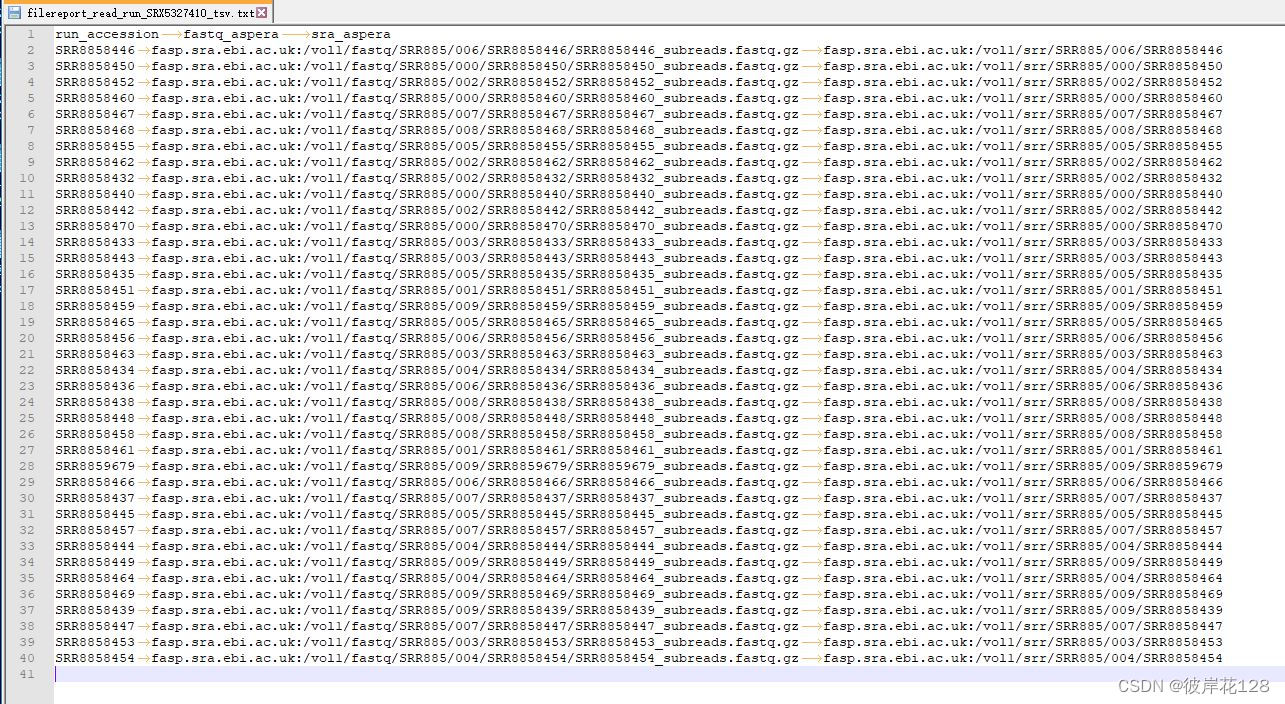
EBI-ENA数据库,检索SRX5327410,点击View 获得结果,点击Show Column Selection,选择run_accession,fastq_aspera,sra_aspera这三项。点击Hide Column Selection
获得结果,点击Show Column Selection,选择run_accession,fastq_aspera,sra_aspera这三项。点击Hide Column Selection
点击Download report后的TSV,得到一个文件filereport_read_run_SRX5327410_tsv.txt
2.3 使用ASpera或Aspera_cli下载sra数据
2.3.1 安装ASpera或Aspera_cli
查看博文——Aspera和Aspera_cli软件的安装和使用
2.3.1 批量下载fastq数据
cd /home/zhaohuiyao/Database/SRX5327410/
#上传文件filereport_read_run_SRX5327410_tsv.txt
awk '{if(NR>1){split($2,arr,":"); print arr[2]}}' filereport_read_run_SRX5327410_tsv.txt > SRX5327410_download_fq.txt
#如果想下载sra数据
awk '{if(NR>1){split($3,arr,":"); print arr[2]}}' filereport_read_run_SRX5327410_tsv.txt > SRX5327410_download_sra.txt
#开始下载
conda activate aspera-cli
ascp -i /home/zhaohuiyao/miniconda3/pkgs/aspera-cli-3.9.6-h5e1937b_0/etc/asperaweb_id_dsa.openssh -l 200M -k 1 -T --mode recv --host fasp.sra.ebi.ac.uk --user fasp --file-list SRX5327410_download_fq.txt ./
conda deactivate
#另一种方式
conda activate aspera-cli
cat SRX5327410_download_fq.txt | while read line; do ascp -i /home/zhaohuiyao/miniconda3/pkgs/aspera-cli-3.9.6-h5e1937b_0/etc/asperaweb_id_dsa.openssh -l 200M -k 1 -T fasp@fasp.sra.ebi.ac.uk:$line ./; done
conda deactivate
2.4 下载某一个SRR检索号的文件(以SRR8858432为例)
EBI-ENA数据库官网:http://ftp.sra.ebi.ac.uk/,依据序列号进入vol1/srr/SRR885/002/SRR8858432
conda activate aspera-cli
ascp -i /home/zhaohuiyao/miniconda3/pkgs/aspera-cli-3.9.6-h5e1937b_0/etc/asperaweb_id_dsa.openssh -l 200M -k 1 -T fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR885/002/SRR8858432/SRR8858432_subreads.fastq.gz ./
conda deactivate
2.5 至此所需的高通量测序数据已下载完成
但如果你下载的是sra数据,不管是从NCBI还是EBI-ENA上,那么你需要将其转为fq文件。需要软件sratoolkit
NCBI下载的是.sra后缀文件,EBI-ENA数据库下载的sra文件没有后缀
2.5.1 sratoolkit的安装
Github官网:https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit,选择与自己的Linux相匹配的安装包(sratoolkit.3.0.7-ubuntu64.tar.gz)
cd /home/zhaohuiyao/Biosoft
#上传安装包sratoolkit.3.0.7-ubuntu64.tar.gz
tar -xzvf sratoolkit.3.0.7-ubuntu64.tar.gz
cd sratoolkit.3.0.7-ubuntu64/
cd bin/
#可执行文件位置:/home/zhaohuiyao/Biosoft/sratoolkit.3.0.7-ubuntu64/bin
2.5.2 sra文件转fq文件,主要用到两个命令
cd /home/zhaohuiyao/Database/SRX5327410/
/home/zhaohuiyao/Biosoft/sratoolkit.3.0.7-ubuntu64/bin/fastq-dump --gzip SRR8858433
#或者
/home/zhaohuiyao/Biosoft/sratoolkit.3.0.7-ubuntu64/bin/fasterq-dump -e 24 -p SRR8858433
#-e:线程数
#-p:显示过程

#批量转sra文件
cd /home/zhaohuiyao/Database/SRX5327410/
cuf -f 1 filereport_read_run_SRX5327410_tsv.txt > SRR_Acc_List.txt
cat SRR_Acc_List.txt | while read line; do /home/zhaohuiyao/Biosoft/sratoolkit.3.0.7-ubuntu64/bin/fasterq-dump -e 24 -p $line -O ./; done







 本文详细介绍了如何通过EBI-ENA数据库和ASpera工具下载SRA数据,包括从NCBI检索SRX5327410数据集,获取fastq文件,以及使用sratoolkit转换sra文件为fastq格式。涉及到的关键步骤包括EBI-ENA数据库的使用、ASpera的安装与配置、以及sra文件的转换。
本文详细介绍了如何通过EBI-ENA数据库和ASpera工具下载SRA数据,包括从NCBI检索SRX5327410数据集,获取fastq文件,以及使用sratoolkit转换sra文件为fastq格式。涉及到的关键步骤包括EBI-ENA数据库的使用、ASpera的安装与配置、以及sra文件的转换。
















 4278
4278

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








