「之前画过的热图:」
本期图片
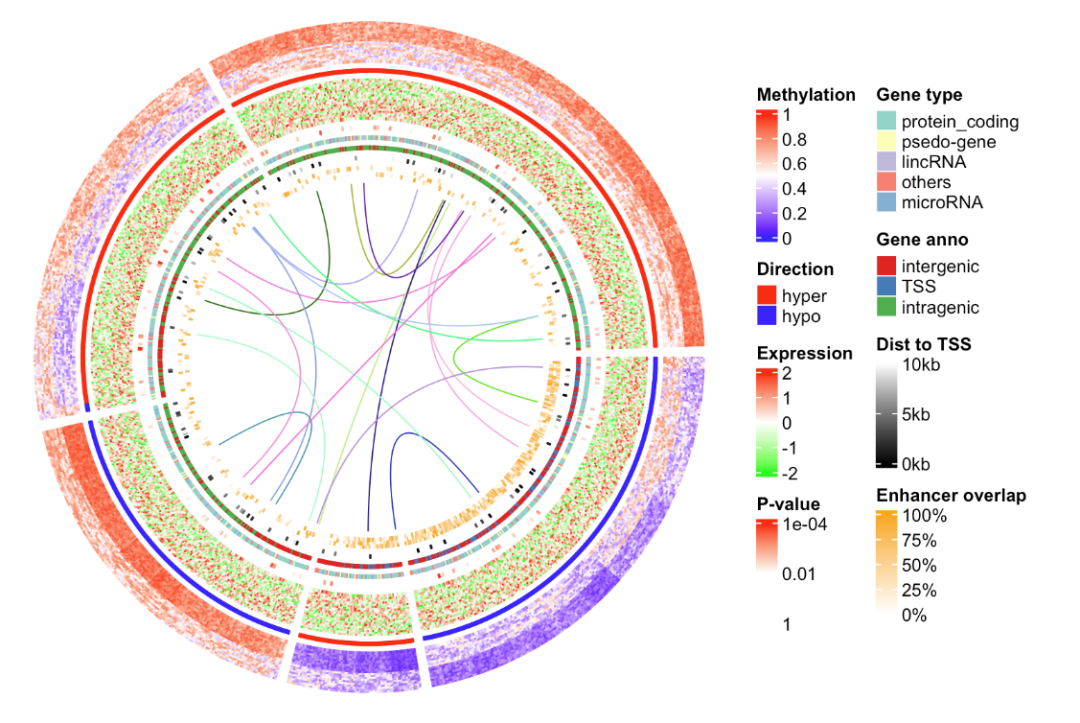
❝将上一期的热图掰弯,并随机给differetially methylated regions (DMRs)加个几个相互作用。
❞
示例数据和代码领取
点赞、在看 本文,分享至朋友圈集赞20个并保留30分钟,截图发至微信mzbj0002领取。
「木舟笔记2022年度VIP可免费领取」。
木舟笔记2022年度VIP企划
「权益:」
「2022」年度木舟笔记所有推文示例数据及代码(「在VIP群里实时更新」)。
data+code 木舟笔记「科研交流群」。
「半价」购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
「收费:」
「99¥/人」。可添加微信:mzbj0002 转账,或直接在文末打赏。

绘制
示例数据
rm(list = ls())
# devtools::install_github("jokergoo/Complexheatmap")
library(ComplexHeatmap)
library(circlize)
library(RColorBrewer)
# 载入示例数据
res_list = readRDS("meth.rds")
str(res_list)
type = res_list$type
mat_meth = res_list$mat_meth
mat_expr = res_list$mat_expr
direction = res_list$direction
cor_pvalue = res_list$cor_pvalue
gene_type = res_list$gene_type
anno_gene = res_list$anno_gene
dist = res_list$dist
anno_enhancer = res_list$anno_enhancer
source("random_matrices.R")绘制
set.seed(123)
km = kmeans(mat_meth, centers = 5)$cluster
# 一圈一圈轨迹叠加
col_meth = colorRamp2(c(0, 0.5, 1), c("blue", "white", "red"))
circos.heatmap(mat_meth, split = km, col = col_meth, track.height = 0.12)
col_direction = c("hyper" = "red", "hypo" = "blue")
circos.heatmap(direction, col = col_direction, track.height = 0.01)
col_expr = colorRamp2(c(-2, 0, 2), c("green", "white", "red"))
circos.heatmap(mat_expr, col = col_expr, track.height = 0.12)
col_pvalue = colorRamp2(c(0, 2, 4), c("white", "white", "red"))
circos.heatmap(cor_pvalue, col = col_pvalue, track.height = 0.01)
col_gene_type = structure(brewer.pal(length(unique(gene_type)), "Set3"), names = unique(gene_type))
circos.heatmap(gene_type, col = col_gene_type, track.height = 0.01)
col_anno_gene = structure(brewer.pal(length(unique(anno_gene)), "Set1"), names = unique(anno_gene))
circos.heatmap(anno_gene, col = col_anno_gene, track.height = 0.01)
col_dist = colorRamp2(c(0, 10000), c("black", "white"))
circos.heatmap(dist, col = col_dist, track.height = 0.01)
col_enhancer = colorRamp2(c(0, 1), c("white", "orange"))
circos.heatmap(anno_enhancer, col = col_enhancer, track.height = 0.03)
circos.clear()
df_link = data.frame(
from_index = sample(nrow(mat_meth), 20),
to_index = sample(nrow(mat_meth), 20)
)
circlize_plot = function() {
circos.heatmap(mat_meth, split = km, col = col_meth, track.height = 0.12)
circos.heatmap(direction, col = col_direction, track.height = 0.01)
circos.heatmap(mat_expr, col = col_expr, track.height = 0.12)
circos.heatmap(cor_pvalue, col = col_pvalue, track.height = 0.01)
circos.heatmap(gene_type, col = col_gene_type, track.height = 0.01)
circos.heatmap(anno_gene, col = col_anno_gene, track.height = 0.01)
circos.heatmap(dist, col = col_dist, track.height = 0.01)
circos.heatmap(anno_enhancer, col = col_enhancer, track.height = 0.03)
for(i in seq_len(nrow(df_link))) {
circos.heatmap.link(df_link$from_index[i],
df_link$to_index[i],
col = rand_color(1))
}
circos.clear()
}
lgd_meth = Legend(title = "Methylation", col_fun = col_meth)
lgd_direction = Legend(title = "Direction", at = names(col_direction),
legend_gp = gpar(fill = col_direction))
lgd_expr = Legend(title = "Expression", col_fun = col_expr)
lgd_pvalue = Legend(title = "P-value", col_fun = col_pvalue, at = c(0, 2, 4),
labels = c(1, 0.01, 0.0001))
lgd_gene_type = Legend(title = "Gene type", at = names(col_gene_type),
legend_gp = gpar(fill = col_gene_type))
lgd_anno_gene = Legend(title = "Gene anno", at = names(col_anno_gene),
legend_gp = gpar(fill = col_anno_gene))
lgd_dist = Legend(title = "Dist to TSS", col_fun = col_dist,
at = c(0, 5000, 10000), labels = c("0kb", "5kb", "10kb"))
lgd_enhancer = Legend(title = "Enhancer overlap", col_fun = col_enhancer,
at = c(0, 0.25, 0.5, 0.75, 1), labels = c("0%", "25%", "50%", "75%", "100%"))
library(gridBase)
plot.new()
circle_size = unit(1, "snpc") # snpc unit gives you a square region
pushViewport(viewport(x = 0, y = 0.5, width = circle_size, height = circle_size,
just = c("left", "center")))
par(omi = gridOMI(), new = TRUE)
circlize_plot()
upViewport()
h = dev.size()[2]
lgd_list = packLegend(lgd_meth, lgd_direction, lgd_expr, lgd_pvalue, lgd_gene_type,
lgd_anno_gene, lgd_dist, lgd_enhancer, max_height = unit(0.9*h, "inch"))
draw(lgd_list, x = circle_size, just = "left")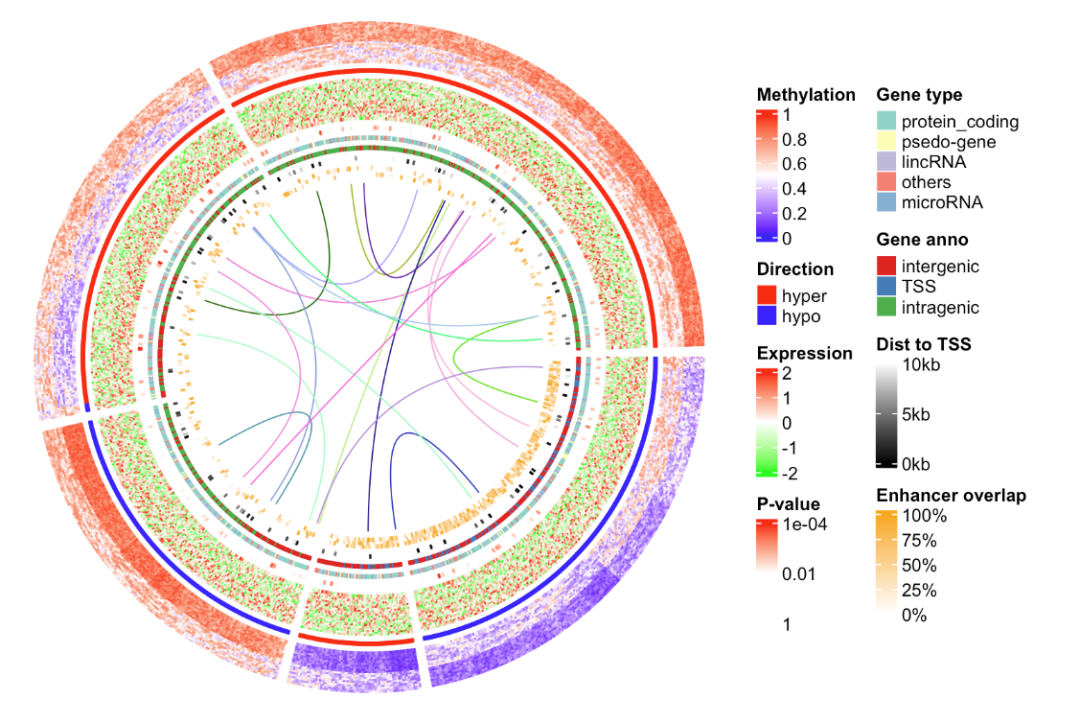
参考
Supplementary S3. Correlations between methylation, expression and other genomic features (jokergoo.github.io)
Chapter 6 The circos.heatmap() function | Circular Visualization in R (jokergoo.github.io)
往期





















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








