💥💥💞💞欢迎来到本博客❤️❤️💥💥
🏆博主优势:🌞🌞🌞博客内容尽量做到思维缜密,逻辑清晰,为了方便读者。
⛳️座右铭:行百里者,半于九十。
📋📋📋本文目录如下:🎁🎁🎁
目录
⛳️赠与读者
👨💻做科研,涉及到一个深在的思想系统,需要科研者逻辑缜密,踏实认真,但是不能只是努力,很多时候借力比努力更重要,然后还要有仰望星空的创新点和启发点。当哲学课上老师问你什么是科学,什么是电的时候,不要觉得这些问题搞笑。哲学是科学之母,哲学就是追究终极问题,寻找那些不言自明只有小孩子会问的但是你却回答不出来的问题。建议读者按目录次序逐一浏览,免得骤然跌入幽暗的迷宫找不到来时的路,它不足为你揭示全部问题的答案,但若能让人胸中升起一朵朵疑云,也未尝不会酿成晚霞斑斓的别一番景致,万一它居然给你带来了一场精神世界的苦雨,那就借机洗刷一下原来存放在那儿的“躺平”上的尘埃吧。
或许,雨过云收,神驰的天地更清朗.......🔎🔎🔎
💥1 概述
文献来源:

摘要 :聚类是使数据在各种应用中变得有意义的重要领域,如处理卫星图像、从财务数据中提取信息,甚至处理社会科学中的数据。本文提出了一种新的聚类方法,称为高斯密度距离(GDD)聚类算法,该算法基于样本空间的距离和密度特性。该方法的创新之处在于,在没有任何先验信息和参数的情况下找到最佳可能的聚类。算法的另一个新颖之处在于,当在二维数据上执行时,它形成的聚类非常接近人类的聚类感知。GDD与当今最受欢迎的聚类算法有一些相似之处;然而,它利用高斯核和距离根据数据密度和形状形成聚类。由于GDD在运行前不需要任何特殊参数,因此产生的聚类在不同运行时不会改变。在研究过程中,设计了一个实验框架来分析提出的聚类算法及其评估,这基于对某些特征数据集的聚类性能。该算法使用多个合成数据集进行了广泛测试,并在论文中展示了一些选定的结果。由其他知名聚类算法产生的比较研究结果也在文中进行了讨论。
从图像处理到网络信息处理,所有未分类的数据都需要被划分成有意义的部分。尽管应用领域广泛,但理解什么是聚类以及我们如何定义正确的簇是聚类任务的重要组成部分。当聚类过程中使用单一的评估目标时,判断得出的分类是好还是坏会变得模糊(Fisher, 1987)。传统的聚类算法通常通过观察数据集的空间属性来寻找有意义的片段。在这个过程的初始阶段,用户需要输入一些参数,如数据集中的起始点、预期的簇数量或形成一个簇所需的样本数量等。这些初始参数对于某些问题可能是有帮助的,其中结果簇的数量是预设的或事先已知的。然而,这些参数显著影响最终的簇,因此恰当地猜测它们需要领域的专业知识。
目前有许多常用的聚类分析方法(Grira, Crucianu, & Boujemaa, 2004; Jain, Murty, Flynn, 1999; Xu & Wunsch, 2005)。根据它们产生结果的方式,这些方法可以分为两大类:划分聚类和层次聚类。划分聚类方法基于数据集的距离或密度标准创建平面聚类。基于距离的方法优化了基于模式间距离的全局准则。一些流行的基于距离的聚类方法包括K-means(Hartigan & Wong, 1979)、CLARA(Wei, Lee, & Hsu, 2000)和CLARANS(Ng & Han, 2002)。另一方面,基于密度的划分聚类方法优化了基于模式密度分布的局部准则,DBSCAN和DenClue属于此类聚类方法(Ester, Kriegel, Sander, & Xu, 1996; Hinneburg, Aggarwal, & Keim, 1998)。层次聚类方法可进一步分为凝聚型和分裂型两大类,分别对应自下而上和自上而下的分析方法(Rani & Rohil, 2013)。关于层次聚类方法的进一步研究可见于Murtagh和Contreras(2011);Zhao, Karypis, 和 Fayyad(2005)。上述所有方法都需要某种形式的参数,如要搜索的簇数或开始过程的初始点。
本文提出了一种新的聚类方法,称为高斯距离分布(GDD),在聚类过程中不需要任何用户交互。尽管文献中有许多无需参数的聚类研究,但大多数都使用基于图的层次聚类方法(Anders & Sester, 2000; Böhm 等, 2007, 2010; He 等, 2009; Ngomo, 2010)或为K-means、DBSCAN和DENCLUE聚类算法提供参数估计方法(Esmaelnejad, Habibi, & Yeganeh, 2010; Gan & Li, 2003; Pei 等, 2008; Pelleg & Moore, 2000)。我们的聚类算法的新颖之处在于:它是非参数的,簇的数量及其边界是在过程中动态确定的,而不是像参数估计研究那样在初始计算时确定。因此,不同运行产生的簇不会改变。GDD聚类的目标是在没有任何先验信息和监督的情况下找到自然簇,这些簇能够像人类感知一样被自然分离。该方法适用于不同样本密度和尺度的自然簇,如形状集合(Jain, Law, 2005)等。
在研究过程中,GDD算法通过使用知名的数据集进行了广泛的测试,这些数据集以形状集著称,如Jain、基于路径、螺旋等(Chang & Yeung, 2008)。数据集在相关部分进行了简要说明,并讨论了选择它们的原因。选择二维形状集是为了更好地观察和简化计算性能测试。提出的算法可以通过使用适当的方程计算欧几里得距离和高斯密度应用于任意维度的数据集。然而,随着维度数量的增加,维度灾难变得更加明显,这在更高维度的聚类中增加了复杂度和挑战(Steinbach, Ertz, & Kumar, 2003)。
论文其余部分的大致内容安排如下:第二部分总结了相关的文献。第三部分详细介绍了我们的算法、所需的数据结构和计算流程。第四部分解释了实验设置并介绍了性能评估指标。第五部分展示了实验结果并就结果进行了讨论。
文献中提出了许多聚类算法,它们被应用于不同的场景中。标准差、方差、密度、分布和欧几里得距离为待聚类的数据提供了重要线索。因此,一般来说,每个聚类算法对于相同的输入数据,无论是在簇成员身份还是产生的簇数量上,都会产生不同的划分。即使使用相同的聚类算法,通过提供不同的参数值、探索不同的模式表示或特征空间,也可能得到交替的聚类结果。本节首先详细介绍一些已知聚类算法的信息及其局限性,然后阐述文献中的非参数聚类算法,探讨它们与所提算法之间的相似性和差异。
2.1. K-均值聚类 在众多聚类算法中,K-均值是最为人熟知的算法之一,它利用距离来寻找聚类,并需要将簇的数量k作为输入参数(Hartigan & Wong, 1979; Macqueen, 1967)。该算法通过从输入数据集中随机选择k个样本作为初始质心开始进行划分,然后迭代地检查输入空间中所有样本的距离来形成簇。与其他聚类算法相比,K-均值不需要太多参数,并且通过一些启发式方法,其计算效率较高。
K-均值的局限性包括:1) 对初始参数有严格依赖。最终的簇划分可能会基于最初选择的参数而变化。2) 无法识别具有任意形状的簇。K-均值的簇模型基于球形簇,这些簇以一种使得均值向簇中心收敛的方式可分离。3) 假设簇应具有相似的大小,以至于分配到最近的簇质心即为正确的分配。
有许多K-均值变体算法试图消除这些局限性或复杂性问题。K-中位数就是其中之一,它在K-均值的基础上使用中位数而非均值(Bradley et al., 1997; Jain & Dubes, 1988)。类似地,K-核心使用算法中的核心点代替均值以最小化距离之和(Kaufman & Rousseeuw, 1987)。还有一种名为模糊C-均值的算法,它为每个数据点找到属于簇质心的模糊归属度来确定哪些点属于哪个簇(Bezdek, 1981)。为了选择更好的初始质心,提出了K-均值++算法(Arthur & Vassilvitskii, 2007)。KD树(Kanungo等人,2002)、核心集(Frahling & Sohler, 2006)和三角不等式(Elkan, 2003)的研究提出了一些修改以加速K-均值聚类过程的执行。逃离局部最小值是该领域内的另一项重要研究(Hartigan & Wong, 1979)。
2.2. DBSCAN聚类 DBSCAN利用密度可达距离来聚类邻近点(Ester等人,1996)。如果一个数据点在半径ε内可达,且相连的数据点超过指定的最少点数(minPts),则该区域被视为一个簇。DBSCAN算法需要两个参数(ε和minPts)来形成簇。也有一些针对DBSCAN的参数估计研究。然而,ε的选择对性能影响很大,如果选择过小,数据空间的大部分区域将不会被聚类;另一方面,如果ε选得过大,簇将会合并。DBSCAN算法使用距离几何上计算簇。如果在使用之前将数据空间映射到R*-树结构,可以降低DBSCAN算法的复杂度(Arlia & Coppola, 2001)。
2.3. OPTICS聚类 OPTICS作为DBSCAN的一种变体,不使用固定的范围参数ε来进行聚类,而是利用密度信息找到相应的范围参数(Ankerst等人,1999)。因此,OPTICS在寻找具有不同密度的嵌套簇时更为强大。此外,动态方法DM-DBSCAN作为文献中OPTICS的补充,通过动态方法解决全局范围参数ε的值,改进了不同密度的聚类(Elbatta & Ashour, 2013)。OPTICS算法与DBSCAN有相似的起源,但OPTICS能够发现不同密度的簇。
2.4. GMM-EM聚类
GMM-EM将数据集中的每个点定义为一个高斯函数,用于定义聚类(Shental、Bar-Hillel、Hertz和Weinshall,2003年)。高斯值添加到聚类系统中的责任因子定义了在形成聚类时强关联于另一个点。在GMM-EM中,使用期望最大化(EM)来找到聚类(Dempster、Laird和Rubin,1977年)。寻找最优GMM是一个NP问题,优化是一个复杂的过程。当数据散布成高斯分布时,期望最大化使用高斯混合模型是有效的,然而使用该算法对于不同形状和样本分组的数据进行聚类是困难的。GMM-EM方法试图将作为输入参数的高斯数量最佳地拟合到表示数据集样本密度的空间。如果高斯正确拟合,那么聚类过程结束。通过使用期望最大化找到的高斯参数。然而,这种方法耗时且在数据形状是非高斯时可能无法正确聚类。
2.5. 谱聚类
谱聚类使用相似矩阵的特征值来划分数据进行聚类,尤其在图像分析方面表现良好。在谱聚类中有几种变体算法稍有不同。它们易于实现,并且可以通过标准线性代数方法有效解决。例如,图割算法通过使用路径将图分割成段(Hagen、Kahng,1992年,Shi、Malik,2000年)。创建切分路径和最小化切分成本是图割方法的主要关注点。另一种谱聚类算法变体使用K-Means方法来找到聚类。在谱聚类中,输入参数或相似性图的更改会极大地影响聚类的结果(Ng、Jordan和Weiss,2001年)。
2.6. 其他无参数聚类方法
文献中的无参数聚类研究之一是关于空间数据库及其应用的(Anders & Sester,2000年)。在这项工作中,采用了基于层次图的聚类方法,通过使用中位数和方差的统计计算来定义聚类。尽管在统计信息的使用上与GDD存在相似性用于定义聚类,但是由于它使用了基于图的方法,因此形成的聚类在根本上是不同的。
另一种基于图的无参数聚类研究灵感来源于生物现象:蛋白质相互作用(PPI)网络(Ngomo & Axel-Cyrille,2010年)。在这种方法中,从PPI获得了一个相似性矩阵,然后搜索最小切割路径来获取聚类。这项研究与图割算法非常类似,只是形成相似性矩阵的方式不同。
另一种基于图的无参数聚类研究被称为PaCK:“可扩展的无参数K-部分图聚类”(He等,2009年)。PaCK试图将聚类问题构建为一个压缩问题。其目标是基于二进制连接矩阵中的模式来查找无参数方式下,它也使用最小描述长度(MDL)原则来设计标准,并试图贪心地最小化这个标准。基本上,它使用k-部分图对数据进行聚类;使用图和压缩技术进行聚类使其与我们提出的方法完全不同。
另一种无参数聚类研究,不基于图的,使用了MDL、似然和数据压缩方法(Böhm等,2010年)。在这项研究中,也使用了高斯密度函数,然而,一般的聚类过程采用了类似于哈夫曼编码的熵公式。这种方法与GMM-EM聚类有一些相似之处,然而,GDD为每个样本数据的包含使用了叠加的高斯密度值,而不是用于聚类区域估计。
另一种无参数聚类研究称为RIC:无参数噪声鲁棒聚类,也使用MDL确定数据集中自然聚类(Böhm等,2007年)。RIC聚类技术试图在存在噪声的情况下估计聚类的相关结构。RIC还使用熵计算对数据进行编码和压缩,就像Böhm等人(2010年)所做的那样。RIC主要关注噪声对聚类的影响,而GDD在其方法中并未明确考虑噪声。
📚2 运行结果
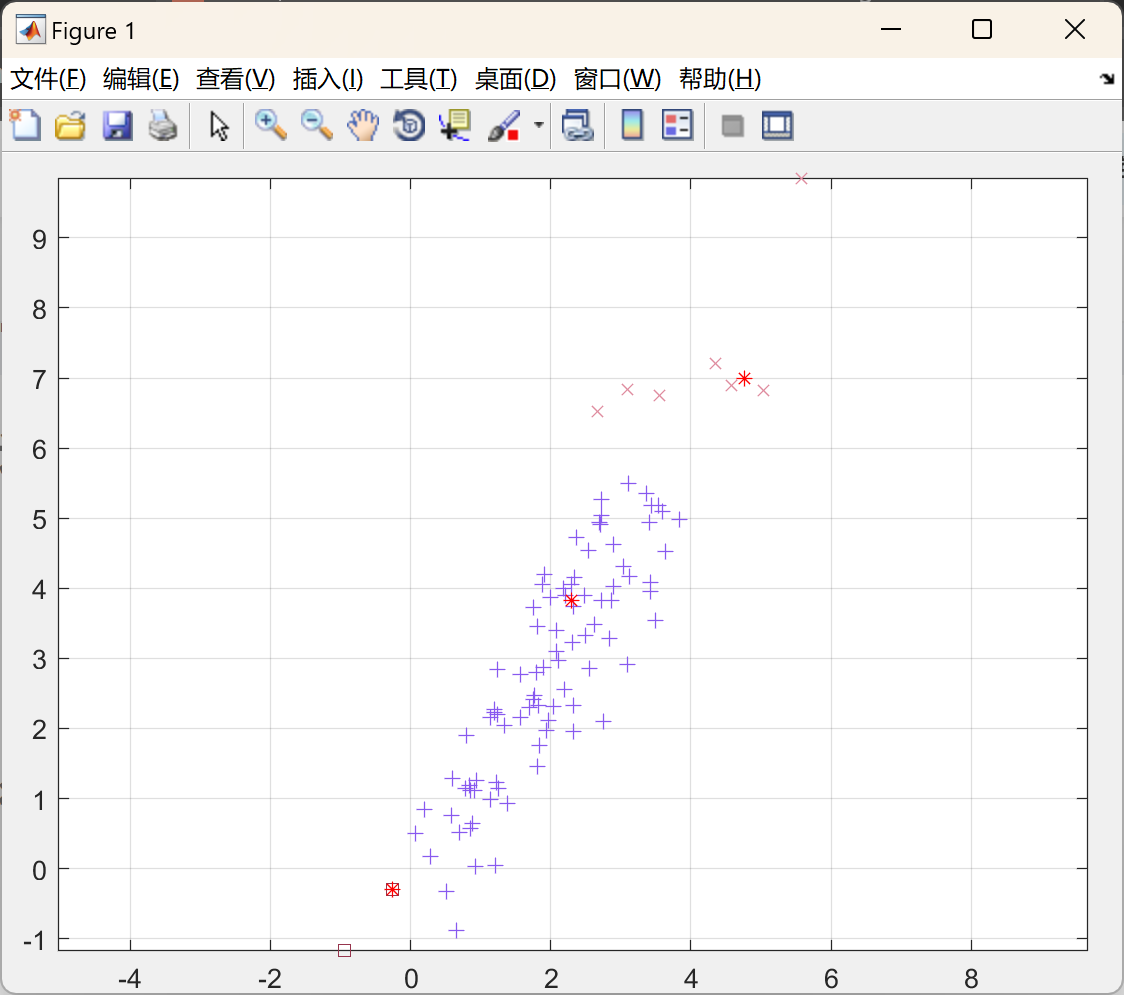

部分代码:
%% GDD Algorithm
function [CLUSTERS,centroids]=func_GDD(DataSet)
% tic
% CLUSTERS=[];
% centroids=[];
%DataSet=[y x];
%% FILTERING-----------------------------------------------------------------
X_CPU=DataSet;
N=size(X_CPU,1); %Number of Data Samples
%DUPLICATE REMOVAL
% %ALL POINTS IN X Needed to be unique (No Duplicate Points)
% [n,m,p]=size(X_CPU);
% a=reshape(X_CPU,n,[],1);
% b=reshape(a(:),n*m,[])';
% % c=unique(b,'rows','stable')';
% c=unique(b,'rows')';
% % c=unique(b)';
% X_CPU=reshape(c,n,m,[]);
A = table(X_CPU);
[C, indexA, indexC]=unique(A);
X_CPU=table2array(C);
% Return if N=2 as 2 cluster
if(size(X_CPU,1)==2 || size(X_CPU,1)<= size(DataSet,1)/100)
for i=1:size(X_CPU,1)
CLUSTERS{i}=[i];
centroids(i)=i;
end
%Re-order Cluster Indices According to Original DuplicateDataSet if Any
for i=1:size(CLUSTERS,2)
ClustMemCount=size(CLUSTERS{i},2);
for j=1:ClustMemCount
OrgIndices=find(indexC(:)==CLUSTERS{i}(j));
CLUSTERS{i}(j)=[];
for k=1:size(OrgIndices,1)
CLUSTERS{i}(end+1)=OrgIndices(k);
end
end
🎉3 参考文献
文章中一些内容引自网络,会注明出处或引用为参考文献,难免有未尽之处,如有不妥,请随时联系删除。

🌈4 Matlab代码、文章
资料获取,更多粉丝福利,MATLAB|Simulink|Python资源获取
























 2863
2863











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










