1、引言
周集中教授团队多年在群落确定性和随机性过程深耕,于2017年总结出来下述图表中的计算路径,广受生态领域研究者的推崇。本文基于该计算路径,梳理了整理了邓晔老师组的代码,通过一个实例演示了计算过程和结果。
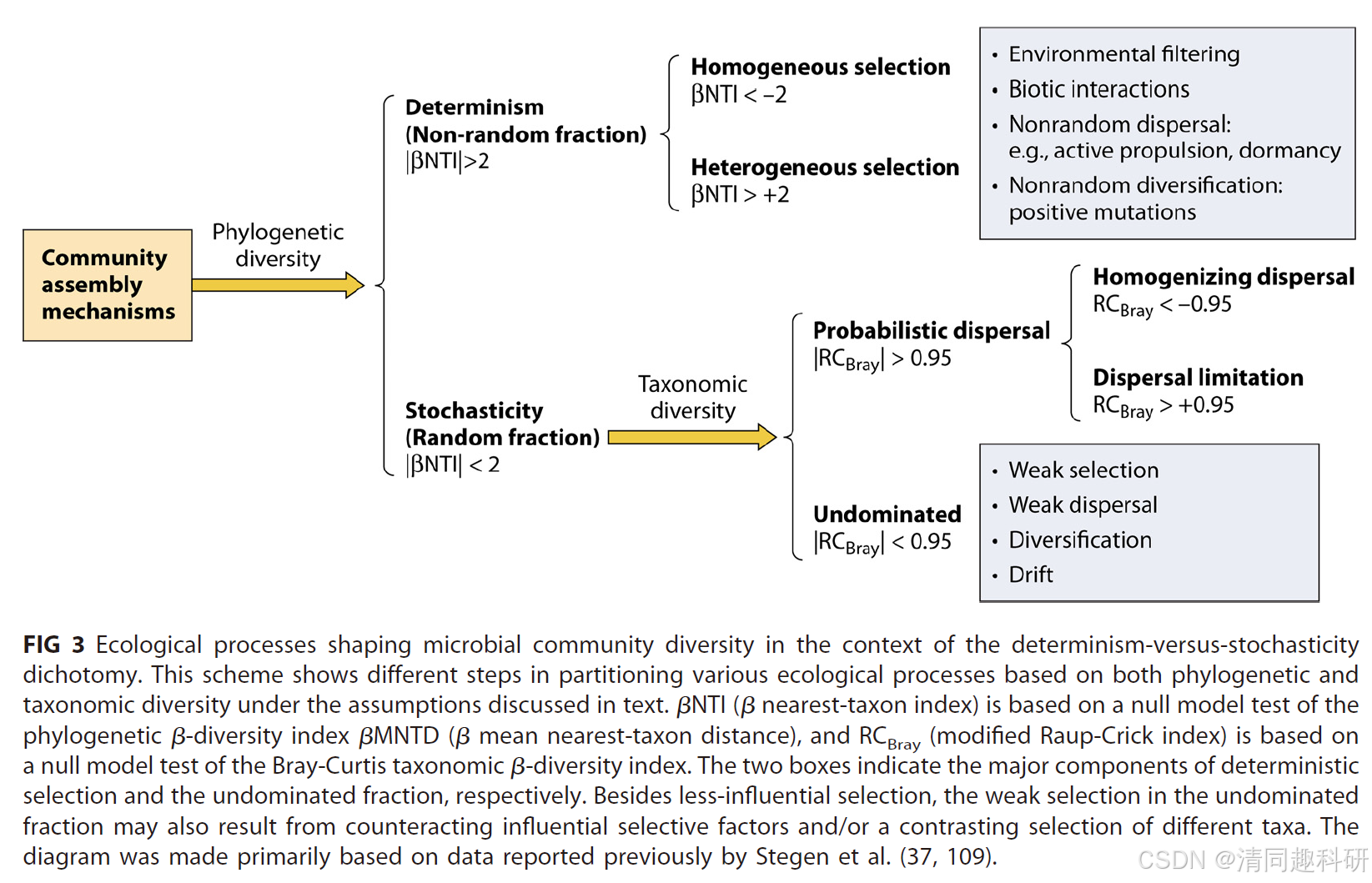
图片来源:Jizhong Zhou and Daliang Ning, Microbiology and Molecular Biology Reviews
2、示例数据和R代码
2.1、文件准备
准备下述三个文件
(1)OTU_table.txt是OTU(ASV)表,是OUT序列数量信息

(2)tree.tre是数文件,是OTU表对应的系统发育信息

2.2、计算βNTI数据表
#设置工作目录和清除环境
setwd("E:R/QPEN") #设置工作目录
rm(list = ls(all=T)) #清空当前工作空间所有对象
#加载必要的包
library(picante)
library(ape) # picante 和 ape 库用于处理生态数据和系统发育数据
library(parallel) #用于并行化计算,加速代码执行
# 读取OTU表格和系统发育树数据
otu <- read.table(file = "OTU_table_H.txt", sep = "\t", header = TRUE, row.names = 1)
tree <- read.tree("tree.tre", tree.names = NULL)
rand <- 1000 # 随机化次数
# 匹配物种丰度和系统发育树
matched <- match.phylo.comm(tree, t(otu))
comm <- matched$comm # 匹配后的物种丰度数据
phylo <- matched$phy # 匹配后的系统发育树
# 计算系统发育树的Cophenetic距离
phydist <- cophenetic(phylo)
weighted = TRUE # 是否按物种丰度加权
# 计算实际观察到的bMNTD
bmntd.obs <- as.matrix(comdistnt(comm, phydist, abundance.weighted = weighted, exclude.conspecifics = FALSE))
# 定义随机化函数
bMNTD.randomization <- function(i, dis, com, weighted, exclude.consp) {
library(picante)
dist.rand <- dis
rand.name <- sample(colnames(dis))
colnames(dist.rand) <- rand.name
rownames(dist.rand) <- rand.name
bMNTD.rand <- as.matrix(comdistnt(com, dist.rand, abundance.weighted = weighted, exclude.conspecifics = exclude.consp))
bMNTD.rand
}
# 并行化计算随机化bMNTD
c1 <- parallel::makeCluster(2, type = "PSOCK")
bmntd.random <- parallel::parLapply(c1, 1:rand, bMNTD.randomization,
dis = phydist, com = comm, weighted = weighted, exclude.consp = FALSE)
parallel::stopCluster(c1)
# 将随机化结果转换为数组
bmntd.random.array <- array(unlist(bmntd.random), dim = c(nrow(bmntd.random[[1]]), ncol(bmntd.random[[1]]), length(bmntd.random)))
# 计算bNTI指数
bNTI <- (bmntd.obs - apply(bmntd.random.array, c(1, 2), mean)) / (apply(bmntd.random.array, c(1, 2), sd))
diag(bNTI) <- 0 # 将对角线上的值设为0
bNTI[is.na(bNTI)] <- 0 # 将NA值设为0
bNTI[is.na(bmntd.obs)] <- NA # 将NA值设为NA
# 将bNTI指数写入CSV文件
write.csv(bNTI, paste("bNTI", weighted, "weighted.csv", sep = ".")) # 根据weighted参数命名
输出结果

2.3、RCbray表格计算
# 读取原始数据
comm.orig <- read.table(file = "OTU_table.txt", sep = "\t", header = TRUE, row.names = 1) # 读取OTU表格数据
comm.trans <- t(comm.orig) # 转置数据,使得每行表示一个样本,每列表示一个物种或OTU
# 设置分析方法和计算距离
method <- "bray"
if (method == "jaccard") {
binary <- TRUE
} else {
binary <- FALSE
}
comm <- comm.trans[, colSums(comm.trans) > 0] # 剔除掉所有OTU均为0的列
dt.obs <- as.matrix(vegdist(comm, method = method, binary = binary)) # 计算实际群落数据的距离矩阵
# 随机化过程
dt.null.list <- list()
rand <- 500
for (i in 1:rand) {
comm.rand <- comm
comm.rand[] <- 0
prob.species <- colSums(comm > 0)
prob.abundance <- colSums(comm)
species <- rowSums(comm > 0)
samps <- rowSums(comm)
for (j in 1:nrow(comm)) {
id.species <- sample(1:ncol(comm), species[j], replace = FALSE, prob = prob.species)
count <- sample(id.species, (samps[j] - species[j]), replace = TRUE, prob = prob.abundance[id.species])
table <- table(count)
comm.rand[j, as.numeric(names(table))] <- as.vector(table)
comm.rand[j, id.species] <- comm.rand[j, id.species] + 1
}
dt.null.list[[i]] <- as.matrix(vegdist(comm.rand, method = method, binary = binary))
}
# 处理随机化结果并计算Raup-Crick指数
dt.null.array <- array(unlist(dt.null.list), dim = c(nrow(dt.null.list[[1]]), ncol(dt.null.list[[1]]), length(dt.null.list)))
comp <- function(x, c) {
(x < c) + 0.5 * (x == c)
}
alpha <- matrix(rowSums(apply(dt.null.array, 3, comp, c = dt.obs)), nrow = nrow(dt.obs)) / rand
rc <- (alpha - 0.5) * 2
rownames(rc) <- rownames(dt.obs)
colnames(rc) <- colnames(dt.obs)
# 将结果写入CSV文件
write.csv(rc, paste("RaupCrick.", method, ".", rand, ".csv", sep = ""))
输出结果

2.4、群落装配过程确定和汇总
# 读取bNTI和RC数据
bNTI <- read.csv("bNTI.TRUE.weighted.csv", row.names = 1)
RC <- read.csv("RaupCrick.bray.500.csv", row.names = 1)
# 提取样本名称并匹配
samp.nti <- names(bNTI) # bNTI数据的样本名称
samp.rc <- names(RC) # RC数据的样本名称
matched <- match(samp.nti, samp.rc) # 匹配样本名称
RC <- RC[matched, matched] # 根据匹配结果筛选RC数据的子集
# 将bNTI和RC数据转换为向量形式
bNTI.matrix <- as.matrix(bNTI) # 将bNTI转换为矩阵
rowname <- rownames(bNTI.matrix) # 提取行名
colname <- colnames(bNTI.matrix) # 提取列名
rown <- row(bNTI.matrix) # 提取行索引
coln <- col(bNTI.matrix) # 提取列索引
bNTI.v <- as.vector(as.dist(bNTI.matrix)) # 将bNTI矩阵转换为向量
RC.v <- as.vector(as.dist(as.matrix(RC))) # 将RC矩阵转换为向量
rown.v <- as.vector(as.dist(rown))
coln.v <- as.vector(as.dist(coln))
# 构建结果数据框
result <- data.frame(name1 <- rowname[rown.v],name2 <- colname[coln.v],
bNTI <- bNTI.v,RC <- RC.v)
colnames(result) <- c("name1","name2","bNTI","RC")
# 定义生态过程判断函数
eco.pro <- function(res, sig.nti, sig.rc) {
nti <- res[1] # 提取bNTI值
rc <- res[2] # 提取RC值
if (nti > sig.nti) {
process <- "Heterogeneous selection" # 异质选择过程
} else if (nti < (-sig.nti)) {
process <- "Homogeneous selection" # 同质选择过程
} else if (abs(nti) < sig.nti && rc < (-sig.rc)) {
process <- "Homogenizing dispersal" # 同化扩散过程
} else if (abs(nti) < sig.nti && rc > sig.rc) {
process <- "Dispersal limitation" # 扩散限制过程
} else {
process <- "Undominated" # 未支配过程
}
process
}
# 应用生态过程判断函数到每一行
result$EcologicalProcess <- apply(result[, c("bNTI", "RC")], 1, eco.pro, sig.nti = 2, sig.rc = 0.95)
# 写入结果到CSV文件
write.csv(result, "Ecological_process_bNTI_RC.csv", row.names = FALSE)
输出结果
D列:βNTI值
E列:RCbray值
F列:生态装配进程

该表会有不同组之间的生态过程,我们只选择组内生态装配过程,即B组至A组对比相关行删除。
可用orgin,R等工具进一步绘制柱状图和箱线图。
2.5、输出图片



3、参考文献
[1] Zhou J, Ning D. 2017. Stochastic community assembly: does it matter in microbial ecology? Microbiol Mol Biol Rev 81:e00002-17.(背景知识)
[2] Zhaojing Zhang, Ye Deng, et al. Deterministic Assembly and Diversity Gradient Altered the Biofilm Community Performances of Bioreactors.Environmental Science & Technology 2019 53 (3), 1315-1324代码来源)
[3] mem.rcees.ac.cn/index.php/download/(Deng Lab,原代码)
4、相关信息
!!!有需要的小伙伴可以去评论区自取今天的测试代码和实例数据。
📌示例代码中提供了数据和代码,已经小编测试,可直接运行。
⚠️本账号会不时更新代码,代码失效可联系小编~
以上就是利用零模型全部内容。


























 658
658

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








