写在前面
在R语言中,缺失值(NA,Not Available)是数据分析中常见的问题(尤其在处理大型数据集时)。在数据处理过程中,直接删除或认为填写缺失值都是错误的行为,我们需要利用R语言科学的"填写"缺失值处理。R提供了多种方法来检测、处理和替换缺失值,以确保分析结果的准确性和完整性。
1) 需要用到的包
VIM、mice missForest
2) 需要用到的数据集
VIM包中的哺乳动物sleep数据集。该数据集包含62种哺乳动物的睡眠类变量、生态学变量和体质类变量间的关系。睡眠类变量为因变量,另外两个为自变量。睡眠累变量包含做梦时长(Dream)、不做梦时长(NonD)和时长之和(Sleep);体质类变量包括体重(BodyWgt)、脑重(BrainWgt)、寿命(Span)、妊娠期(Gest);生态学变量包括物种被捕食的程度(Pred)、睡眠时的暴露程度(Exp)和面临的总危险度(Danger).
17.1 处理缺失值的步骤
1)识别缺失数据;
2)检查导致数据缺失的原因:缺失数据可以分为3类,完全随机缺失(MCAR),随机缺失(MAR)和非随机缺失(NMAR)。
3)删除包含缺失值的观测值或合理插补。
17.2 识别缺失值
R中使用NA代表缺失值;NaN代表不可能值;Inf和-Inf分别代表正无穷和负无穷值。
17.2.1 is.na()函数
可以识别NA和NaN,返回TRUE;若不是缺失值则返回FALSE。
17.2.2 complete.cases()函数
识别完整数据,返回TRUE;识别到NA和NaN,返回FALSE;
!complete.cases()函数是用于识别缺失值的,返回的结果与complete.cases()函数相反。
17.2.3 举例
y <- c(NA,NaN,Inf,-Inf,5)
is.na(y)## [1] TRUE TRUE FALSE FALSE FALSEcomplete.cases(y)## [1] FALSE FALSE TRUE TRUE TRUE!complete.cases(y)## [1] TRUE TRUE FALSE FALSE FALSElibrary(VIM)## Warning: 程辑包'VIM'是用R版本4.3.2 来建造的## 载入需要的程辑包:colorspace## VIM is ready to use.## Suggestions and bug-reports can be submitted at: https://github.com/statistikat/VIM/issues##
## 载入程辑包:'VIM'## The following objects are masked _by_ '.GlobalEnv':
##
## diabetes, testdata, wine## The following object is masked from 'package:rattle':
##
## wine## The following object is masked from 'package:datasets':
##
## sleepdata(sleep,package = "VIM")
is.na(sleep)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## [1,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [2,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [3,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [4,] FALSE FALSE TRUE TRUE FALSE TRUE FALSE FALSE FALSE FALSE
## [5,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [6,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [7,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [8,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [9,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [10,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [11,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [12,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [13,] FALSE FALSE FALSE FALSE FALSE TRUE TRUE FALSE FALSE FALSE
## [14,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [15,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [16,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [17,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [18,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [19,] FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE
## [20,] FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE
## [21,] FALSE FALSE TRUE FALSE TRUE FALSE FALSE FALSE FALSE FALSE
## [22,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [23,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [24,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [25,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [26,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [27,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [28,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [29,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [30,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [31,] FALSE FALSE TRUE TRUE TRUE FALSE FALSE FALSE FALSE FALSE
## [32,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [33,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [34,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [35,] FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE
## [36,] FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE
## [37,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [38,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [39,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [40,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [41,] FALSE FALSE TRUE FALSE TRUE FALSE FALSE FALSE FALSE FALSE
## [42,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [43,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [44,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [45,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [46,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [47,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [48,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [49,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [50,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [51,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [52,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [53,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [54,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [55,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
## [56,] FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE
## [57,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [58,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [59,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [60,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [61,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [62,] FALSE FALSE TRUE TRUE TRUE FALSE FALSE FALSE FALSE FALSEsleep[complete.cases(sleep),]#列出没有缺失值的行## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## 2 1.000 6.60 6.3 2.0 8.3 4.5 42.0 3 1 3
## 5 2547.000 4603.00 2.1 1.8 3.9 69.0 624.0 3 5 4
## 6 10.550 179.50 9.1 0.7 9.8 27.0 180.0 4 4 4
## 7 0.023 0.30 15.8 3.9 19.7 19.0 35.0 1 1 1
## 8 160.000 169.00 5.2 1.0 6.2 30.4 392.0 4 5 4
## 9 3.300 25.60 10.9 3.6 14.5 28.0 63.0 1 2 1
## 10 52.160 440.00 8.3 1.4 9.7 50.0 230.0 1 1 1
## 11 0.425 6.40 11.0 1.5 12.5 7.0 112.0 5 4 4
## 12 465.000 423.00 3.2 0.7 3.9 30.0 281.0 5 5 5
## 15 0.075 1.20 6.3 2.1 8.4 3.5 42.0 1 1 1
## 16 3.000 25.00 8.6 0.0 8.6 50.0 28.0 2 2 2
## 17 0.785 3.50 6.6 4.1 10.7 6.0 42.0 2 2 2
## 18 0.200 5.00 9.5 1.2 10.7 10.4 120.0 2 2 2
## 22 27.660 115.00 3.3 0.5 3.8 20.0 148.0 5 5 5
## 23 0.120 1.00 11.0 3.4 14.4 3.9 16.0 3 1 2
## 25 85.000 325.00 4.7 1.5 6.2 41.0 310.0 1 3 1
## 27 0.101 4.00 10.4 3.4 13.8 9.0 28.0 5 1 3
## 28 1.040 5.50 7.4 0.8 8.2 7.6 68.0 5 3 4
## 29 521.000 655.00 2.1 0.8 2.9 46.0 336.0 5 5 5
## 32 0.005 0.14 7.7 1.4 9.1 2.6 21.5 5 2 4
## 33 0.010 0.25 17.9 2.0 19.9 24.0 50.0 1 1 1
## 34 62.000 1320.00 6.1 1.9 8.0 100.0 267.0 1 1 1
## 37 0.023 0.40 11.9 1.3 13.2 3.2 19.0 4 1 3
## 38 0.048 0.33 10.8 2.0 12.8 2.0 30.0 4 1 3
## 39 1.700 6.30 13.8 5.6 19.4 5.0 12.0 2 1 1
## 40 3.500 10.80 14.3 3.1 17.4 6.5 120.0 2 1 1
## 42 0.480 15.50 15.2 1.8 17.0 12.0 140.0 2 2 2
## 43 10.000 115.00 10.0 0.9 10.9 20.2 170.0 4 4 4
## 44 1.620 11.40 11.9 1.8 13.7 13.0 17.0 2 1 2
## 45 192.000 180.00 6.5 1.9 8.4 27.0 115.0 4 4 4
## 46 2.500 12.10 7.5 0.9 8.4 18.0 31.0 5 5 5
## 48 0.280 1.90 10.6 2.6 13.2 4.7 21.0 3 1 3
## 49 4.235 50.40 7.4 2.4 9.8 9.8 52.0 1 1 1
## 50 6.800 179.00 8.4 1.2 9.6 29.0 164.0 2 3 2
## 51 0.750 12.30 5.7 0.9 6.6 7.0 225.0 2 2 2
## 52 3.600 21.00 4.9 0.5 5.4 6.0 225.0 3 2 3
## 54 55.500 175.00 3.2 0.6 3.8 20.0 151.0 5 5 5
## 57 0.900 2.60 11.0 2.3 13.3 4.5 60.0 2 1 2
## 58 2.000 12.30 4.9 0.5 5.4 7.5 200.0 3 1 3
## 59 0.104 2.50 13.2 2.6 15.8 2.3 46.0 3 2 2
## 60 4.190 58.00 9.7 0.6 10.3 24.0 210.0 4 3 4
## 61 3.500 3.90 12.8 6.6 19.4 3.0 14.0 2 1 1sum(is.na(sleep$Dream))#计算Dream缺失值有几个## [1] 12mean(is.na(sleep$Dream))#计算 sleep$Dream 列中缺失值的比例## [1] 0.1935484注意:这里的mean()不是用来求均值,而是计算一个逻辑向量中 TRUE 的比例。
17.3 缺失值的可视化
17.3.1 mice包中的md.pattern()函数
该函数生成一个矩阵或数据框形式展示缺失值模式的表格,和用该表格绘制成的图形。
-
17.3.1.1 语法
library(mice)
md.pattern(x,plot=,rotate.names=)x:一个含有缺失值的数据框;
plot:默认为TRUE,作图;
rotate.names:变量名称在图中的排列方式,是水平放置还是垂直放置。默认值为 ‘rotate.names = FALSE’,即不旋转变量名称,水平放置。当为TRUE时,则为垂直放置。
-
17.3.1.2 举例
library(mice)## Warning: 程辑包'mice'是用R版本4.3.2 来建造的##
## 载入程辑包:'mice'## The following objects are masked from 'package:flexclust':
##
## bwplot, densityplot## The following object is masked from 'package:stats':
##
## filter## The following objects are masked from 'package:base':
##
## cbind, rbinddata(sleep,package = "VIM")
md.pattern(sleep,plot=TRUE,rotate.names=TRUE)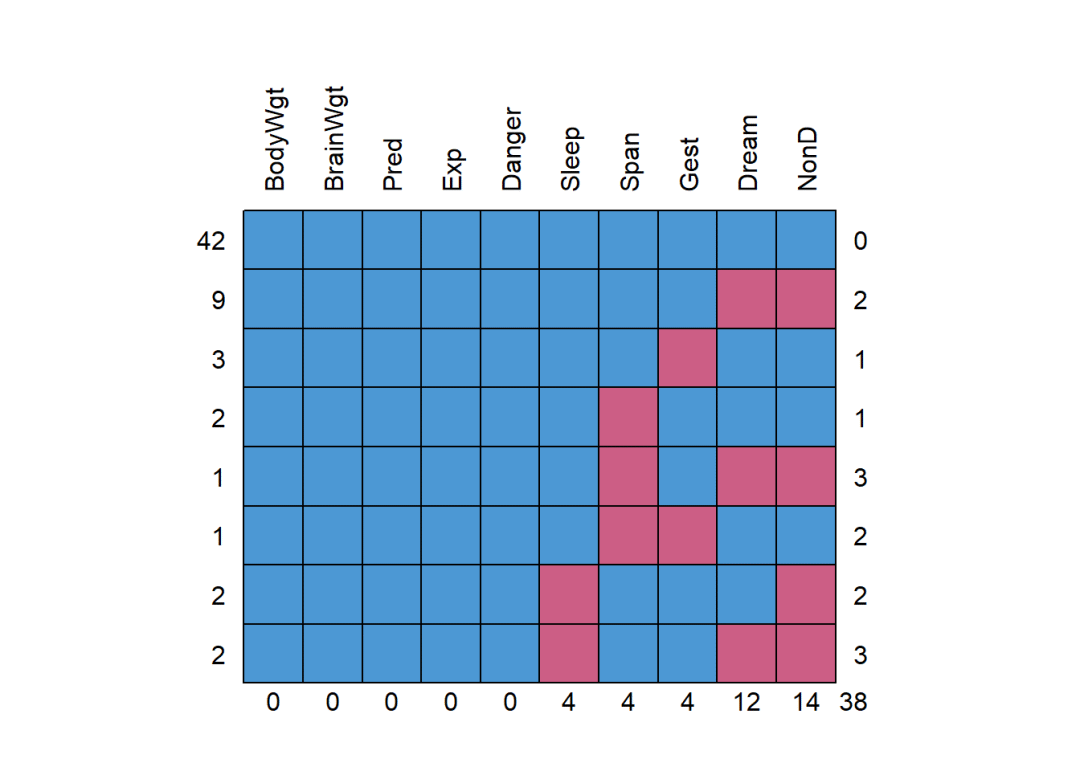
## BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD
## 42 1 1 1 1 1 1 1 1 1 1 0
## 9 1 1 1 1 1 1 1 1 0 0 2
## 3 1 1 1 1 1 1 1 0 1 1 1
## 2 1 1 1 1 1 1 0 1 1 1 1
## 1 1 1 1 1 1 1 0 1 0 0 3
## 1 1 1 1 1 1 1 0 0 1 1 2
## 2 1 1 1 1 1 0 1 1 1 0 2
## 2 1 1 1 1 1 0 1 1 0 0 3
## 0 0 0 0 0 4 4 4 12 14 38结果解释:
1.列表中的0表示有缺失值,1表示没有缺失值。第1列表示各缺失值的观测值个数,最后1列表示各模式中有缺失值的个数。从第一行可以看出共42个观测值没有缺失值。最后一行为每个变量中缺失值的数目。
2.格子图片中,蓝色的为非缺失值,红色的为缺失值。左侧数据相当于列表中的第一列,为观测值的数目;右侧相当于列表的最后一列,为缺失值的个数。下面为各变量中缺失值的总数。
17.3.2 VIM包中的aggr()函数
-
17.3.2.1 语法
library(VIM)
aggr(x,prop=,numbers=)x:数据框;
prop:一个逻辑值,指示是否使用缺失/插补值和组合的比例,而不是总数。prop=TRUE,返回的是指数形式,prop=FALSE时返回的是数字形式;
numbers:一个逻辑值,指示不同组合的比例或频率是否应该用数字表示。当=FALSE(默认)时,则删去数值型标签。
-
17.3.2.2 举例
library(VIM)
aggr(sleep,prop=FALSE,numbers=TRUE)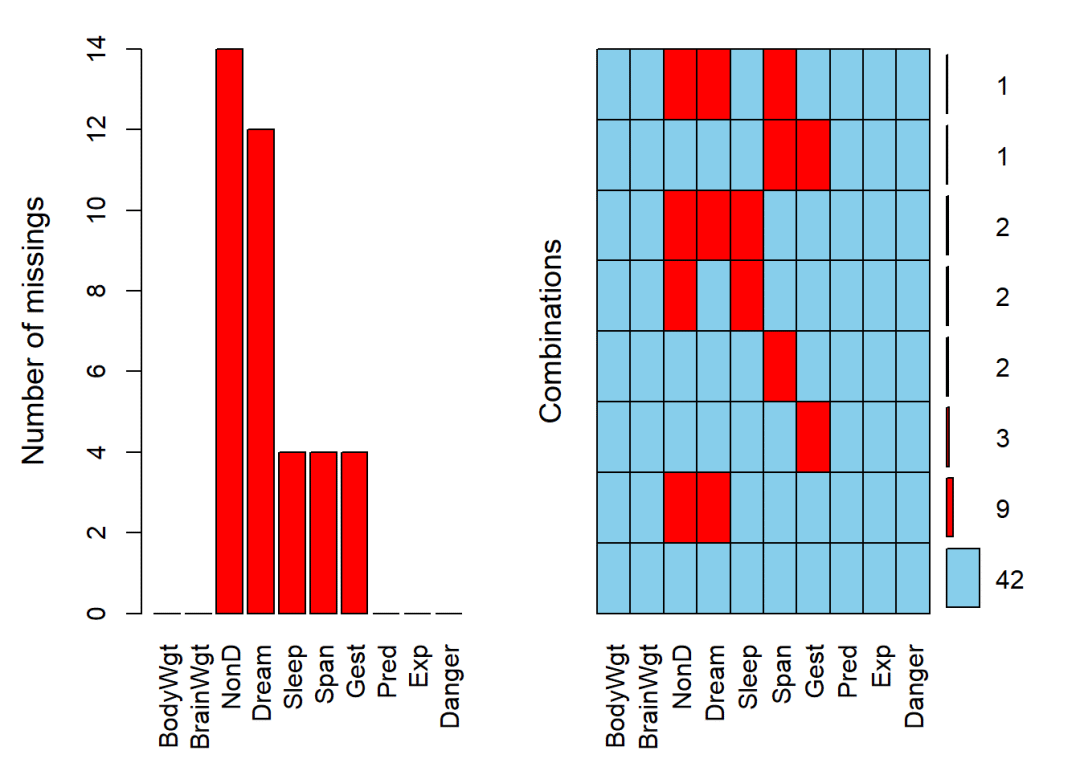
左图中红色柱为含缺失值的变量中缺失值的数目,右图中蓝色为非缺失值,红色为缺失值,右侧数字为有这些缺失值的观测值数量。
17.4 删除缺失值
主要包括行删除和成对删除,其中成对删除用的很少,且会导致数据的一些扭曲,故不推荐。
17.4.1 行删除(na.omit()函数)
newdata <- na.omit(sleep)
aggr(sleep)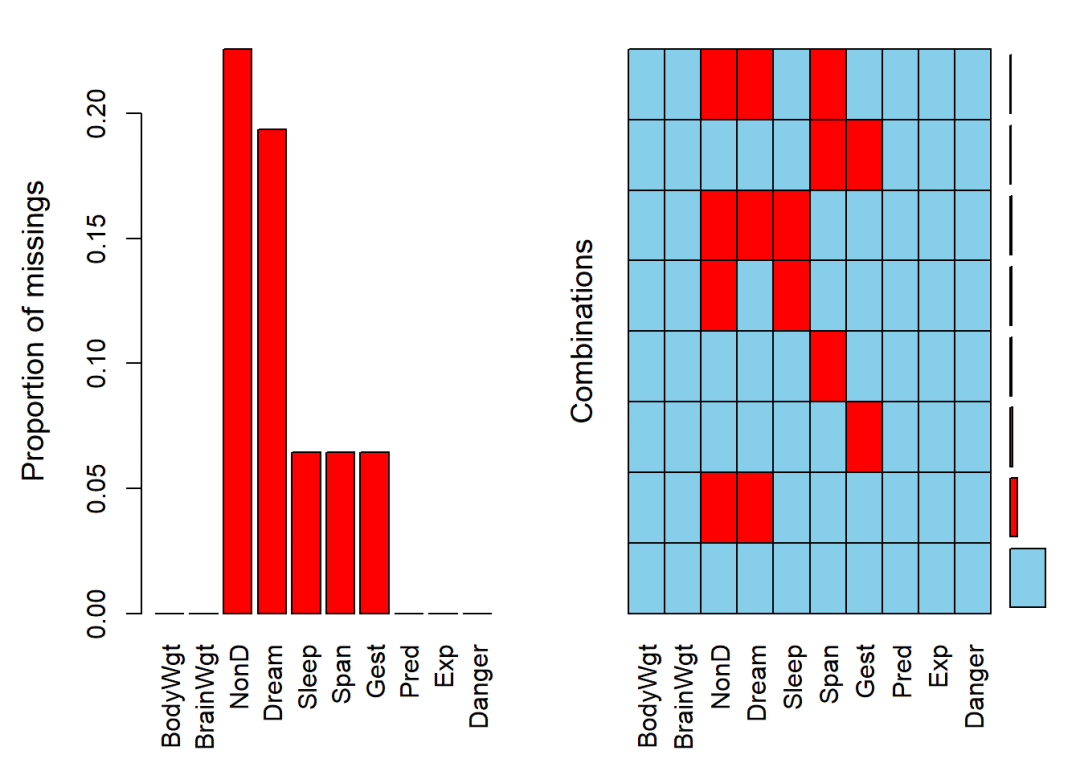
aggr(newdata)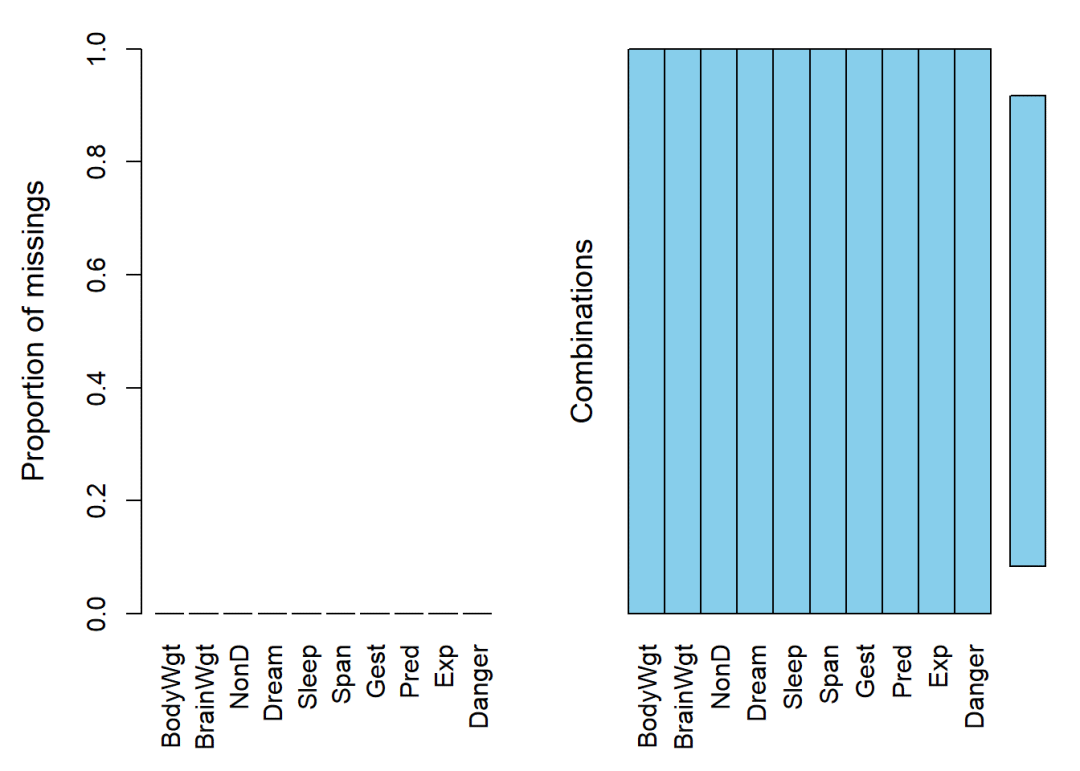
summary(sleep)## BodyWgt BrainWgt NonD Dream
## Min. : 0.005 Min. : 0.14 Min. : 2.100 Min. :0.000
## 1st Qu.: 0.600 1st Qu.: 4.25 1st Qu.: 6.250 1st Qu.:0.900
## Median : 3.342 Median : 17.25 Median : 8.350 Median :1.800
## Mean : 198.790 Mean : 283.13 Mean : 8.673 Mean :1.972
## 3rd Qu.: 48.202 3rd Qu.: 166.00 3rd Qu.:11.000 3rd Qu.:2.550
## Max. :6654.000 Max. :5712.00 Max. :17.900 Max. :6.600
## NA's :14 NA's :12
## Sleep Span Gest Pred
## Min. : 2.60 Min. : 2.000 Min. : 12.00 Min. :1.000
## 1st Qu.: 8.05 1st Qu.: 6.625 1st Qu.: 35.75 1st Qu.:2.000
## Median :10.45 Median : 15.100 Median : 79.00 Median :3.000
## Mean :10.53 Mean : 19.878 Mean :142.35 Mean :2.871
## 3rd Qu.:13.20 3rd Qu.: 27.750 3rd Qu.:207.50 3rd Qu.:4.000
## Max. :19.90 Max. :100.000 Max. :645.00 Max. :5.000
## NA's :4 NA's :4 NA's :4
## Exp Danger
## Min. :1.000 Min. :1.000
## 1st Qu.:1.000 1st Qu.:1.000
## Median :2.000 Median :2.000
## Mean :2.419 Mean :2.613
## 3rd Qu.:4.000 3rd Qu.:4.000
## Max. :5.000 Max. :5.000
##summary(newdata)## BodyWgt BrainWgt NonD Dream
## Min. : 0.0050 Min. : 0.14 Min. : 2.100 Min. :0.000
## 1st Qu.: 0.3162 1st Qu.: 3.60 1st Qu.: 6.150 1st Qu.:0.900
## Median : 2.2500 Median : 12.20 Median : 8.500 Median :1.650
## Mean : 100.8139 Mean : 218.68 Mean : 8.743 Mean :1.900
## 3rd Qu.: 10.4125 3rd Qu.: 155.50 3rd Qu.:11.000 3rd Qu.:2.375
## Max. :2547.0000 Max. :4603.00 Max. :17.900 Max. :6.600
## Sleep Span Gest Pred
## Min. : 2.90 Min. : 2.00 Min. : 12.0 Min. :1.000
## 1st Qu.: 8.05 1st Qu.: 5.25 1st Qu.: 32.0 1st Qu.:2.000
## Median : 9.80 Median : 11.20 Median : 90.0 Median :3.000
## Mean :10.64 Mean : 19.37 Mean :129.9 Mean :2.952
## 3rd Qu.:13.60 3rd Qu.: 27.00 3rd Qu.:195.0 3rd Qu.:4.000
## Max. :19.90 Max. :100.00 Max. :624.0 Max. :5.000
## Exp Danger
## Min. :1.000 Min. :1.00
## 1st Qu.:1.000 1st Qu.:1.25
## Median :2.000 Median :2.50
## Mean :2.357 Mean :2.69
## 3rd Qu.:3.750 3rd Qu.:4.00
## Max. :5.000 Max. :5.00aggr和summary的结果均显示,sleep中存在NA,而newdata中不存在NA。
我们再看一下共删除了多少行观测值:
nrow(newdata)## [1] 42nrow(sleep)## [1] 62这里看到,行删除后只剩42行,而原来有62行,共删除了20行观测值。有些缺失值遍布到观测值的数目较多,使用这一方法可能会导致删除的观测值过多而对结果产生一定误差。那么也可以使用插补的方法来对数据缺失值进行预处理。
17.5 单一插补
17.5.1 简单插补(simple imputation)
即用某个值(均值、中位数或众数)来替换变量中的缺失值。针对连续型变量,比较推荐使用中位数;针对分类型变量一般用众数进行简单插补。
-
17.5.1.1 语法
zoo包中的na.aggregate()函数
library(zoo)
na.aggregate(object, by = 1, ..., FUN = mean,
na.rm = FALSE, maxgap = Inf)object:进行插补的对象,一般是某数据集中的某一变量,例如sleep$NonD
FUN:mean,均值插补;median,中位数插补;mode,众数插补
na.rm:逻辑值。是否应该删除剩余的缺失值?,默认为FALSE
-
17.5.1.2 举例
library(zoo)
summary(sleep$NonD)## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 2.100 6.250 8.350 8.673 11.000 17.900 14sleep$NonD <- na.aggregate(sleep$NonD, by = 1, FUN = median,
na.rm = FALSE, maxgap = Inf)
summary(sleep$NonD)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 2.10 6.80 8.35 8.60 10.55 17.90可以看到,按中位数插补后,中位数和最小值和最大值都没有变化,均值稍微变了一点但变化不大。
17.5.2 k邻近插补
对于具有一个或多个缺失值的观测值,找到与其最相似但具有值(数值完整)的观测值,然后利用这些观测值进行插补。
该方法适用于观测值小于1000的小-中型数据集,针对更大的数据集可以使用missForest插补。
-
17.5.2.1 语法
使用VIM包中的kNN()函数,详细用法见VIM::kNN;在k邻近插补中,连续缺失值的汇总值时k个(一般是5个)最邻近值的中位数,类别型缺失值则使用众数。
library(VIM)
kNN(data)-
17.5.2.2 举例
library(VIM)
head(sleep)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## 1 6654.000 5712.0 8.35 NA 3.3 38.6 645 3 5 3
## 2 1.000 6.6 6.30 2.0 8.3 4.5 42 3 1 3
## 3 3.385 44.5 8.35 NA 12.5 14.0 60 1 1 1
## 4 0.920 5.7 8.35 NA 16.5 NA 25 5 2 3
## 5 2547.000 4603.0 2.10 1.8 3.9 69.0 624 3 5 4
## 6 10.550 179.5 9.10 0.7 9.8 27.0 180 4 4 4sleep_imp <- kNN(sleep)
head(sleep_imp)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger BodyWgt_imp
## 1 6654.000 5712.0 8.35 0.8 3.3 38.6 645 3 5 3 FALSE
## 2 1.000 6.6 6.30 2.0 8.3 4.5 42 3 1 3 FALSE
## 3 3.385 44.5 8.35 2.3 12.5 14.0 60 1 1 1 FALSE
## 4 0.920 5.7 8.35 2.4 16.5 3.2 25 5 2 3 FALSE
## 5 2547.000 4603.0 2.10 1.8 3.9 69.0 624 3 5 4 FALSE
## 6 10.550 179.5 9.10 0.7 9.8 27.0 180 4 4 4 FALSE
## BrainWgt_imp NonD_imp Dream_imp Sleep_imp Span_imp Gest_imp Pred_imp Exp_imp
## 1 FALSE FALSE TRUE FALSE FALSE FALSE FALSE FALSE
## 2 FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## 3 FALSE FALSE TRUE FALSE FALSE FALSE FALSE FALSE
## 4 FALSE FALSE TRUE FALSE TRUE FALSE FALSE FALSE
## 5 FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## 6 FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## Danger_imp
## 1 FALSE
## 2 FALSE
## 3 FALSE
## 4 FALSE
## 5 FALSE
## 6 FALSE结果显示,sleep_imp数据框中新添加了10个变量,即原变量_imp,这是kNN()函数默认添加的,可以使用imp_var=FALSE来阻止这10个变量的添加。
library(VIM)
sleep_imp1 <- kNN(sleep,imp_var = FALSE)
head(sleep_imp1)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## 1 6654.000 5712.0 8.35 0.8 3.3 38.6 645 3 5 3
## 2 1.000 6.6 6.30 2.0 8.3 4.5 42 3 1 3
## 3 3.385 44.5 8.35 2.3 12.5 14.0 60 1 1 1
## 4 0.920 5.7 8.35 2.4 16.5 3.2 25 5 2 3
## 5 2547.000 4603.0 2.10 1.8 3.9 69.0 624 3 5 4
## 6 10.550 179.5 9.10 0.7 9.8 27.0 180 4 4 417.5.3 missForest 插补
主要针对大型数据集(观测值大于500-1000),使用随机森林法来插补缺失值。
-
17.5.3.1 语法
missForest包中的missForest()函数,详细用法见missForest::missForest
library(missForest)
set.seed()
missForest(xmis)$ximpxmis:一个包含缺失值的数据矩阵。列对应于变量,行对应于观测值。
这里使用set.seed()函数是为了使结果具有重复性。
-
17.5.3.2 举例
library(missForest)## Warning: 程辑包'missForest'是用R版本4.3.2 来建造的##
## 载入程辑包:'missForest'## The following object is masked from 'package:VIM':
##
## nrmseset.seed(1234)
sleep_missForest <- missForest(sleep)$ximp
head(sleep)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## 1 6654.000 5712.0 8.35 NA 3.3 38.6 645 3 5 3
## 2 1.000 6.6 6.30 2.0 8.3 4.5 42 3 1 3
## 3 3.385 44.5 8.35 NA 12.5 14.0 60 1 1 1
## 4 0.920 5.7 8.35 NA 16.5 NA 25 5 2 3
## 5 2547.000 4603.0 2.10 1.8 3.9 69.0 624 3 5 4
## 6 10.550 179.5 9.10 0.7 9.8 27.0 180 4 4 4head(sleep_missForest)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## 1 6654.000 5712.0 8.35 1.350 3.3 38.600 645 3 5 3
## 2 1.000 6.6 6.30 2.000 8.3 4.500 42 3 1 3
## 3 3.385 44.5 8.35 2.333 12.5 14.000 60 1 1 1
## 4 0.920 5.7 8.35 2.524 16.5 6.934 25 5 2 3
## 5 2547.000 4603.0 2.10 1.800 3.9 69.000 624 3 5 4
## 6 10.550 179.5 9.10 0.700 9.8 27.000 180 4 4 4$ximp 是从插补结果中提取出填补后的数据矩阵。
17.6 多重插补(multiple imputation, MI)
是一种基于重复模拟的处理缺失值的方法。当面对复杂的缺失值问题时,MI最常用。MI是从一个包含缺失值的数据集中生成一组(3-10个)对应的完整的数据集,这些数据集为模拟数据集,缺失数据将用蒙特卡罗法填充。R中可以使用mice、Amelia和mi包执行,这里介绍mice包的用法。
17.6.1 语法
1.插补
library(mice)
imp <- mice(data,m)
fit <- with(imp,analysis)
pooled <- pool(fit)
summary(pooled)data:一个包含缺失值的矩阵或数据库;
imp:一个包含m个插补数据集(模拟数据集)的列表和对象,同时含有完整插补过程的信息。默认m=5,也可以设置为其他值,m为模拟数据集的数目;
analysis:一个表达式对象,用来设定应用于m个插补数据集的统计分析方法,包括线性回归模型的lm()函数,广义线性模型的glm()函数、广义可加模型的gam();
fit:一个包含m个单独统计分析结果的列表对象;
pooled:一个包含m个统计分析平均结果的列表对象。
注意:如果仅用lm()函数无法在多重插补的基础上建立回归模型,所以需要借助with和pool函数。
2.查看
插补的结果保存在imp$imp中,可以查看其中任一变量的插补结果;也可以查看完整的插补数据集:
Hide
imp$imp$变量
complete(imp,action = n)1.通过提取imp对象的子成分,可以观测到实际的插补值;
2.complete()函数用来查看m个插补数据集(模拟数据集)中的任意一个,其中n为1时则查看第一个插补数据集,n为5时则查看第5个插补数据集。n不会大于m。
17.6.2 举例
library(mice)
imp <- mice(sleep,method="rf",seed=1234,m=5)##
## iter imp variable
## 1 1 Dream Sleep Span Gest
## 1 2 Dream Sleep Span Gest
## 1 3 Dream Sleep Span Gest
## 1 4 Dream Sleep Span Gest
## 1 5 Dream Sleep Span Gest
## 2 1 Dream Sleep Span Gest
## 2 2 Dream Sleep Span Gest
## 2 3 Dream Sleep Span Gest
## 2 4 Dream Sleep Span Gest
## 2 5 Dream Sleep Span Gest
## 3 1 Dream Sleep Span Gest
## 3 2 Dream Sleep Span Gest
## 3 3 Dream Sleep Span Gest
## 3 4 Dream Sleep Span Gest
## 3 5 Dream Sleep Span Gest
## 4 1 Dream Sleep Span Gest
## 4 2 Dream Sleep Span Gest
## 4 3 Dream Sleep Span Gest
## 4 4 Dream Sleep Span Gest
## 4 5 Dream Sleep Span Gest
## 5 1 Dream Sleep Span Gest
## 5 2 Dream Sleep Span Gest
## 5 3 Dream Sleep Span Gest
## 5 4 Dream Sleep Span Gest
## 5 5 Dream Sleep Span Gestfit <- with(imp,lm(Dream~Span+Gest))
pooled <- pool(fit)
summary(pooled)## term estimate std.error statistic df p.value
## 1 (Intercept) 2.486829211 0.260066801 9.5622709 42.70262 3.541325e-12
## 2 Span -0.006983738 0.011828155 -0.5904335 54.65253 5.573338e-01
## 3 Gest -0.003299397 0.001527141 -2.1605066 40.91960 3.664093e-02imp$imp$Dream## 1 2 3 4 5
## 1 1.0 1.8 0.6 0.7 1.8
## 3 1.8 2.3 2.0 1.8 2.4
## 4 3.4 3.4 2.6 1.4 1.4
## 14 0.6 0.7 0.8 0.5 0.9
## 24 0.8 1.3 1.2 1.2 1.2
## 26 3.4 4.1 2.4 3.6 1.4
## 30 3.4 3.9 1.4 1.9 1.2
## 31 1.2 0.9 1.4 1.2 0.6
## 47 1.9 2.4 2.4 0.9 2.0
## 53 0.0 0.6 0.6 0.5 0.5
## 55 1.8 0.9 0.8 0.9 1.2
## 62 2.0 0.5 3.1 1.2 0.0complete(imp,action = 2)## BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
## 1 6654.000 5712.00 8.35 1.8 3.3 38.6 645.0 3 5 3
## 2 1.000 6.60 6.30 2.0 8.3 4.5 42.0 3 1 3
## 3 3.385 44.50 8.35 2.3 12.5 14.0 60.0 1 1 1
## 4 0.920 5.70 8.35 3.4 16.5 9.0 25.0 5 2 3
## 5 2547.000 4603.00 2.10 1.8 3.9 69.0 624.0 3 5 4
## 6 10.550 179.50 9.10 0.7 9.8 27.0 180.0 4 4 4
## 7 0.023 0.30 15.80 3.9 19.7 19.0 35.0 1 1 1
## 8 160.000 169.00 5.20 1.0 6.2 30.4 392.0 4 5 4
## 9 3.300 25.60 10.90 3.6 14.5 28.0 63.0 1 2 1
## 10 52.160 440.00 8.30 1.4 9.7 50.0 230.0 1 1 1
## 11 0.425 6.40 11.00 1.5 12.5 7.0 112.0 5 4 4
## 12 465.000 423.00 3.20 0.7 3.9 30.0 281.0 5 5 5
## 13 0.550 2.40 7.60 2.7 10.3 4.7 28.0 2 1 2
## 14 187.100 419.00 8.35 0.7 3.1 40.0 365.0 5 5 5
## 15 0.075 1.20 6.30 2.1 8.4 3.5 42.0 1 1 1
## 16 3.000 25.00 8.60 0.0 8.6 50.0 28.0 2 2 2
## 17 0.785 3.50 6.60 4.1 10.7 6.0 42.0 2 2 2
## 18 0.200 5.00 9.50 1.2 10.7 10.4 120.0 2 2 2
## 19 1.410 17.50 4.80 1.3 6.1 34.0 310.0 1 2 1
## 20 60.000 81.00 12.00 6.1 18.1 7.0 164.0 1 1 1
## 21 529.000 680.00 8.35 0.3 3.9 28.0 400.0 5 5 5
## 22 27.660 115.00 3.30 0.5 3.8 20.0 148.0 5 5 5
## 23 0.120 1.00 11.00 3.4 14.4 3.9 16.0 3 1 2
## 24 207.000 406.00 8.35 1.3 12.0 39.3 252.0 1 4 1
## 25 85.000 325.00 4.70 1.5 6.2 41.0 310.0 1 3 1
## 26 36.330 119.50 8.35 4.1 13.0 16.2 63.0 1 1 1
## 27 0.101 4.00 10.40 3.4 13.8 9.0 28.0 5 1 3
## 28 1.040 5.50 7.40 0.8 8.2 7.6 68.0 5 3 4
## 29 521.000 655.00 2.10 0.8 2.9 46.0 336.0 5 5 5
## 30 100.000 157.00 8.35 3.9 10.8 22.4 100.0 1 1 1
## 31 35.000 56.00 8.35 0.9 3.8 16.3 33.0 3 5 4
## 32 0.005 0.14 7.70 1.4 9.1 2.6 21.5 5 2 4
## 33 0.010 0.25 17.90 2.0 19.9 24.0 50.0 1 1 1
## 34 62.000 1320.00 6.10 1.9 8.0 100.0 267.0 1 1 1
## 35 0.122 3.00 8.20 2.4 10.6 3.9 30.0 2 1 1
## 36 1.350 8.10 8.40 2.8 11.2 4.7 45.0 3 1 3
## 37 0.023 0.40 11.90 1.3 13.2 3.2 19.0 4 1 3
## 38 0.048 0.33 10.80 2.0 12.8 2.0 30.0 4 1 3
## 39 1.700 6.30 13.80 5.6 19.4 5.0 12.0 2 1 1
## 40 3.500 10.80 14.30 3.1 17.4 6.5 120.0 2 1 1
## 41 250.000 490.00 8.35 1.0 10.9 23.6 440.0 5 5 5
## 42 0.480 15.50 15.20 1.8 17.0 12.0 140.0 2 2 2
## 43 10.000 115.00 10.00 0.9 10.9 20.2 170.0 4 4 4
## 44 1.620 11.40 11.90 1.8 13.7 13.0 17.0 2 1 2
## 45 192.000 180.00 6.50 1.9 8.4 27.0 115.0 4 4 4
## 46 2.500 12.10 7.50 0.9 8.4 18.0 31.0 5 5 5
## 47 4.288 39.20 8.35 2.4 12.5 13.7 63.0 2 2 2
## 48 0.280 1.90 10.60 2.6 13.2 4.7 21.0 3 1 3
## 49 4.235 50.40 7.40 2.4 9.8 9.8 52.0 1 1 1
## 50 6.800 179.00 8.40 1.2 9.6 29.0 164.0 2 3 2
## 51 0.750 12.30 5.70 0.9 6.6 7.0 225.0 2 2 2
## 52 3.600 21.00 4.90 0.5 5.4 6.0 225.0 3 2 3
## 53 14.830 98.20 8.35 0.6 2.6 17.0 150.0 5 5 5
## 54 55.500 175.00 3.20 0.6 3.8 20.0 151.0 5 5 5
## 55 1.400 12.50 8.35 0.9 11.0 12.7 90.0 2 2 2
## 56 0.060 1.00 8.10 2.2 10.3 3.5 30.0 3 1 2
## 57 0.900 2.60 11.00 2.3 13.3 4.5 60.0 2 1 2
## 58 2.000 12.30 4.90 0.5 5.4 7.5 200.0 3 1 3
## 59 0.104 2.50 13.20 2.6 15.8 2.3 46.0 3 2 2
## 60 4.190 58.00 9.70 0.6 10.3 24.0 210.0 4 3 4
## 61 3.500 3.90 12.80 6.6 19.4 3.0 14.0 2 1 1
## 62 4.050 17.00 8.35 0.5 5.4 13.0 38.0 3 1 1aggr(complete(imp,action = 2))
第一个命令返回表展示了针对Dream变量的5次插补,第二个命令返回表展示了第2个完整的插补数据集。























 802
802

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








