newCellDataSet(data.matrix(mat_to_cluster), phenoData = pd, featureData = fd,
expressionFamily = negbinomial.size(),
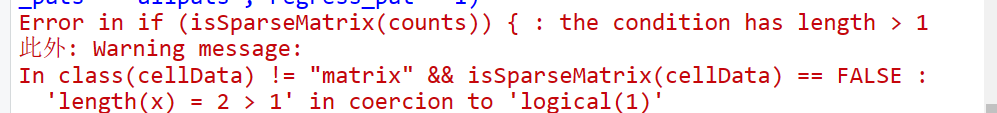
lowerDetectionLimit = 0.1)之前R的版本是4.1是可以正常运行的,但是升级为4.2版本时就报错了,如上图,我尝试了多种方法,最后发现用Seurat的as.sparse函数可以运行,也就是把data.matrix换成as.sparse
newCellDataSet(as.sparse(mat_to_cluster), phenoData = pd, featureData = fd,
expressionFamily = negbinomial.size(),
lowerDetectionLimit = 0.1)但是我发现了聚类的结果不一样,之前是7类

现在是13类

因为版本问题,聚类的结果也不一样,这个暂时没想到怎么办,不过问题不大,我们可以自己设计如何聚类。





















 667
667











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








