实践目的和意义:用ggClusterNet 做网络分析图并计算网络中的拓扑学指数,尤其是鲁棒性数值,来体现网络的稳健性。
#安装ggClusterNet
remotes::install_github("taowenmicro/ggClusterNet", force = T)
#加载所用的包
library(ggClusterNet)
library(phyloseq)
library(tidyverse)
library(igraph)
library(sna)
library(network)
数据的导入:
构建ps的数据格式
ps即phyloseq的数据格式为:
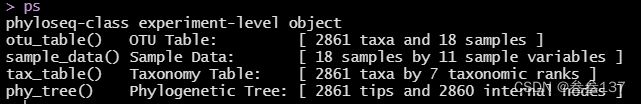
主要的文件为:OTU Table 、Sample Data、Taxonomy Table这三个作为构建ps数据的核心文件。
OTU Table格式为:
OTU ID sample1 sample2 sample3 .......
OUT_1
OUT_2
Sample Data格式为:
第一列为sampleid 第二列为分组
Taxonomy Table
OTU domain phylum class order family genus species
OUT_1
OUT_2
library(phyloseq)
library(ggClusterNet)
library(tidyverse)
library(Biostrings)
otutable <- read.table("./table_filtered.xls", header = T, sep = "\t",row.names = 1)
taxnomoy <- read.table("./tax.txt", header = T,sep = "\t", fill=TRUE, na.strings = "NA", row.names = 1)
metadata <- read.table("./metadata.tsv", header = T,sep = "\t", row.names = 1)
ps <- phyloseq(sample_data(metadata),otu_table(as.matrix(otutable),taxa_are_rows = T),tax_table(as.matrix(taxnomoy)))这样我们就得到了一个基础的数据格式,用于分析数据
分析:
打包输出网络分析内容
根据上述导入的方式,导入数据
ps1 <- phyloseq(otu_table(as.matrix(otu_1),taxa_are_rows = T),sample_data(meta_1),tax_table(as.matrix(taxon)))
############打包输出网络中的结果############
path1 = "./result_micro_R/"
dir.create(path1)
result11 = network.2(ps = ps1,
N = 100, #取前丰度最高的前100
layout_net = "model_Gephi.2", ##布局格式model_Gephi.2这里我用的Gephi的
r.threshold=0.6, #相关性的阈值
p.threshold=0.01, #显著性的阈值
label = TRUE, #是否在节点上标记标签
path = path1, #存放的文件
lab = "Genus", #标签用Genus 这里一定要与taxnomoy中列名一致
zipi = TRUE #是否话zipi图)
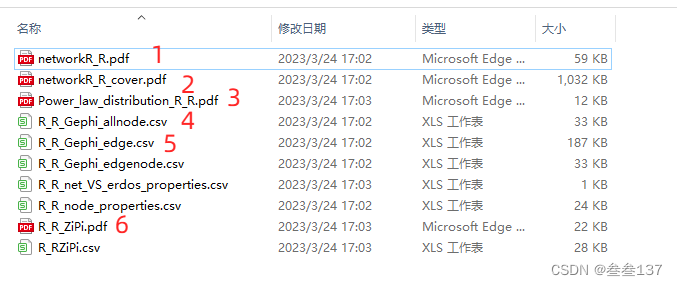
#输出的文件如下:
1、2文件都是网络关系图, 4、5是节点和边文件可以导入到Gephi的文件,可以进行修图。
下图是其中的一个文件
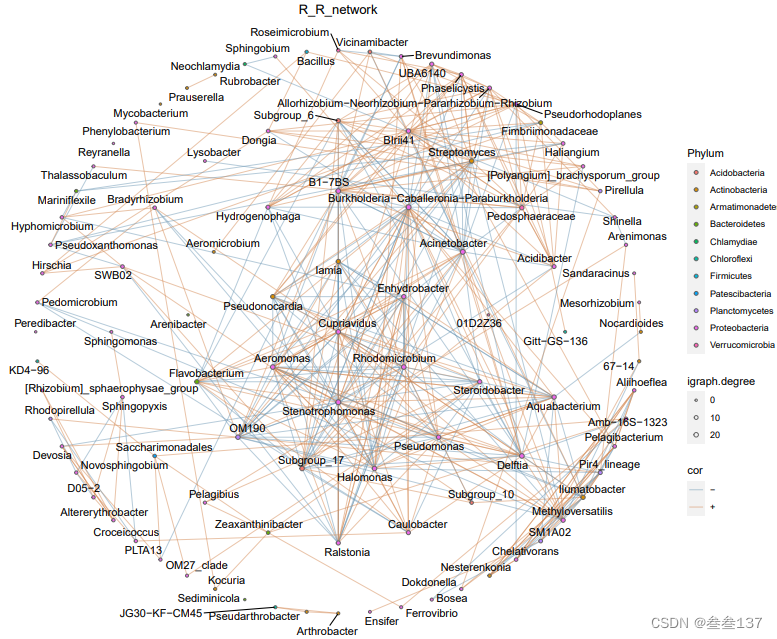
3文件为:度与节点的分布
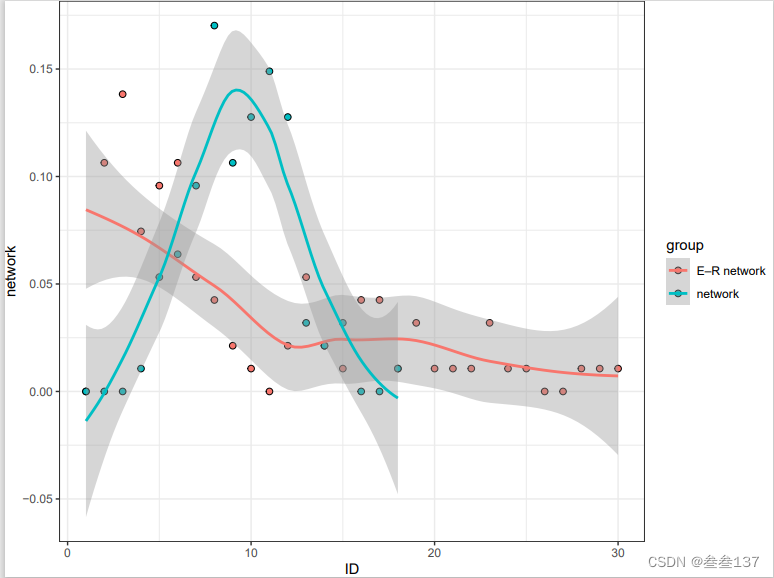
这里注意的是红色的为本此实践的网络,绿的为随机网络。虽然不知道为啥这个图例这样写,但是根据源代码来看,因该是这样的。这个图证明我们的网络是无尺度网络,不是正态分布的随机网络。
6文件是zipi图
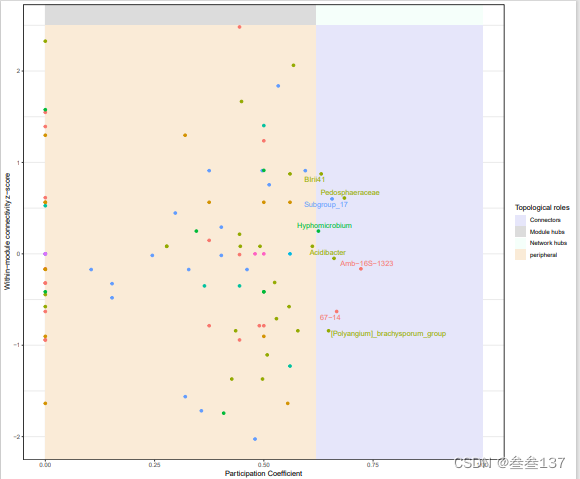
拓扑学指数的计算
########计算相关的############################
result1 <- corMicro(ps=ps1, N=100, method.scale = "TMM", r.threshold = 0.6, p.threshold = 0.01, method = "spearman")
cor1 <- result1[[1]] ####取出想要的结果
result1_4 = nodeEdge(cor = cor1) #####得到节点和边文件
edge1 = result1_4[[1]] #边文件
node1 = result1_4[[2]] #节点文件
igraph1 = igraph::graph_from_data_frame(edge1, directed = FALSE, vertices = node1)
dat1 = net_properties.2(igraph1,n.hub = T) # 可以计算初很多的拓扑学指数(见下表)
网络稳定性和鲁棒性(robustness)
#这里我比较了两组的,可以在metadata中调整分组
otutable <- read.table("./table.xls", header = T, sep = "\t",row.names = 1)
taxnomoy <- read.table("./tax.txt", header = T,sep = "\t", fill=TRUE, na.strings = "NA", row.names = 1)
metadata <- read.table("./metadata.txt", header = T,sep = "\t", row.names = 1)
ps <- phyloseq(sample_data(metadata),otu_table(as.matrix(otutable),taxa_are_rows = T),tax_table(as.matrix(taxnomoy)))
#计算鲁棒性
#result <- corMicro(ps=ps, N=150, method.scale = "TMM", r.threshold = 0.8, p.threshold = 0.05, method = "spearman")
cor <- result[[1]]
png(filename = "robust.png",width = 1000, height = 500 )
p <- Robustness.Random.removal.change1(ps,Top = 200,r.threshold = 0.6, p.threshold = 0.01,method = "spearman")
p
dev.off()这里计算的鲁棒性,函数Robustness.Random.removal 需要你手动改一下,因为他写的有点问题,对于考虑丰度的加权的鲁棒性分析不出来,具体修改的函数笔者附在后边。这里我改成了Robustness.Random.removal.change1函数。
图为:左边为加权的,右边为非加权的(即考不考虑丰度)
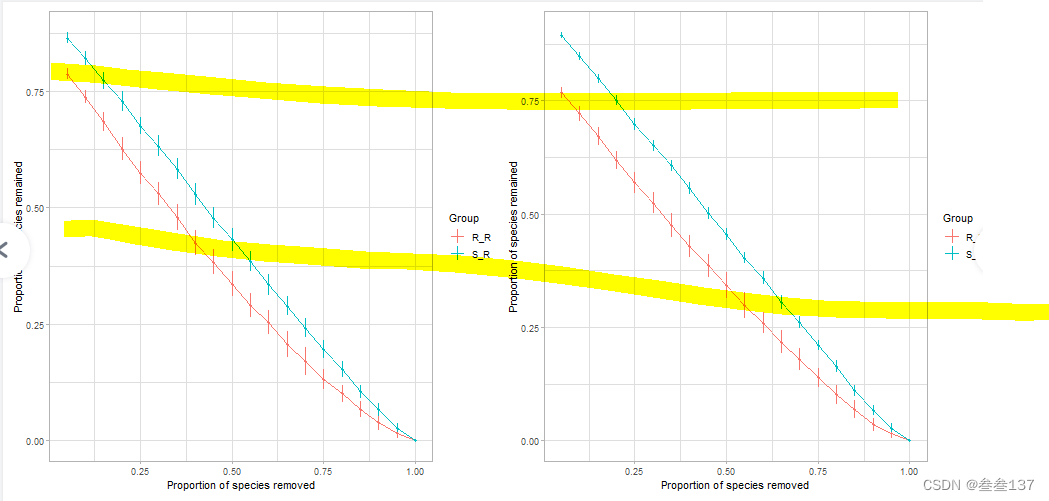
横坐标为随机去除的节点数,纵坐标为留下的节点
表明随机去除相应的节点,保留下来的节点多,表明这些去除的节点对网络的影响越小,进而网络也就越稳定。
ggclustnet还有很多的功能,有待大家们探索~~~~~~~~~~~~





















 2059
2059











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








