
网络分析概述之网络基础简介

本文转载自小白名的生统笔记,原文链接:https://mp.weixin.qq.com/s/Bp-u663WUhFxPfeP7pBh0w
从本节开始介绍网络分析。本篇暂且对网络概念作个初步简介,随后的章节再一一展示怎样分析数据。根据白鱼同学目前比较熟悉的领域,今后的章节主要以“微生物互作网络”、“基因共表达网络”为例作阐述。
注:该“网络”是指数学中分析数据的一种方法,不是“互联网”,也不是“神经网络”之类的。
网络基础概述
何为网络,及其基本结构
在数学中,“网络”(networks)通常被称为“图”(graphs),关于图研究的数学领域称为“图理论”(graph theory)。网络图中的基本元素是节点和边,构建网络图时,将对象称为“节点”(vertices或nodes),并通常将它们绘制为点;将节点之间的连接称为“边”(edges或links),并且通常将它们绘制为点之间的线。如此,得到了网络图。

正式地来说,一个图G=(V,E)是一种包含“节点”集合V与“边”集合E的数学结构,其中E的元素是不同节点的无序组合{u,v},u,v∈V。节点的数量Nv=|V|和边的数量Ne=|E|有时分别被称为图G的“阶数”(order)和“规模”(size)。不失一般性,常简单地使用整数1,…,Nv对节点和边进行标记。
网络可以代表现实世界中的各种系统,例如,可以将因特网描述为网络,其中节点是计算机或其他设备,边是设备之间的物理连接(线路,或者无线);食物链中,节点是物种,边代表了能量的单向流动。
也可以反映假定的关联模型,例如,通过转录组的基因表达水平的相对变化,推测基因间的相互作用关系;微生物物种的互作关系难以像宏观物种那样直接观测,也常根据丰度的协变去推断。
其他示例如社交网络、运输网络、代谢网络、通信网络等等。如下展示一些网络图示例。

(https://mathinsight.org/image/internet_map_jurvetson_2004)
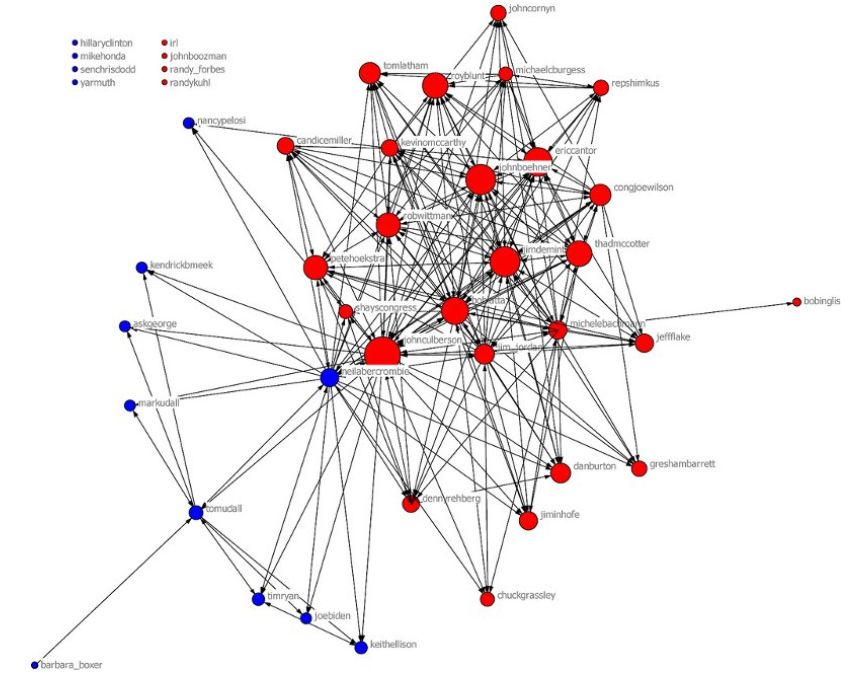
(https://mathinsight.org/image/us_congress_twitterers_porternovelli_2009)
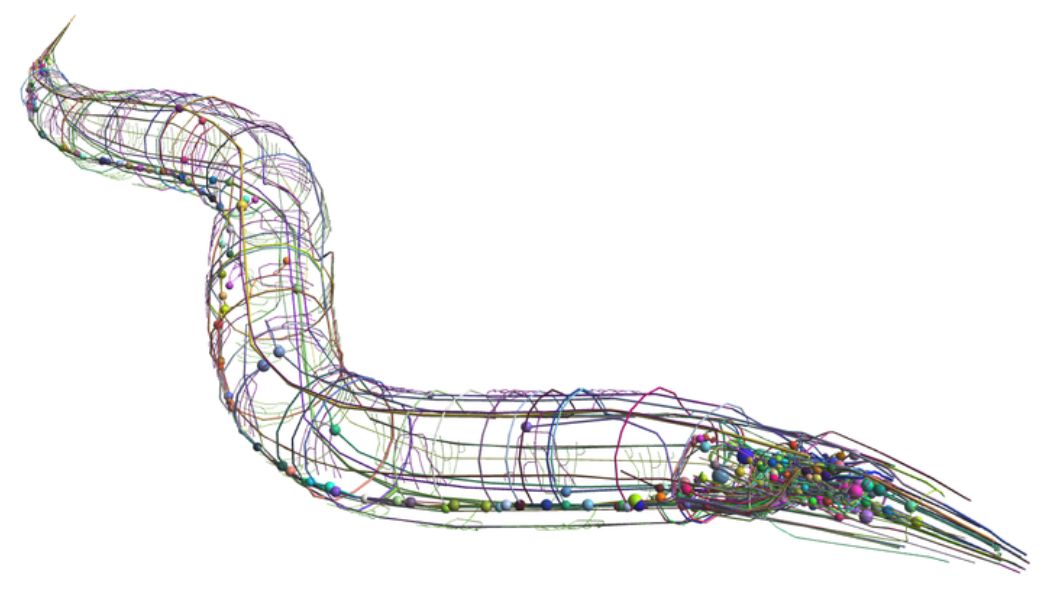
(https://mathinsight.org/image/c_elegans_connectome_anatomy_model)
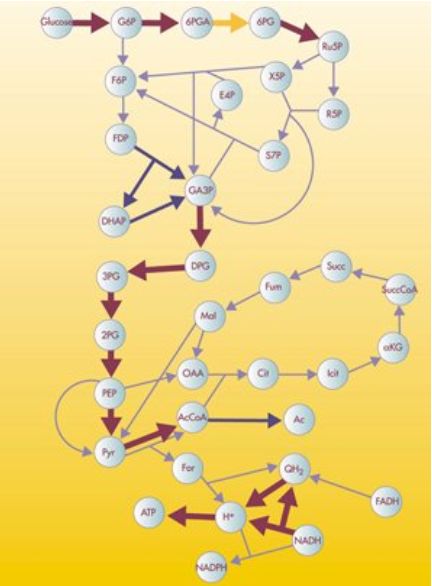
(https://mathinsight.org/image/metabolic_network_model_for_Escherichia_coli)
网络图基本类型,有向图和无向图
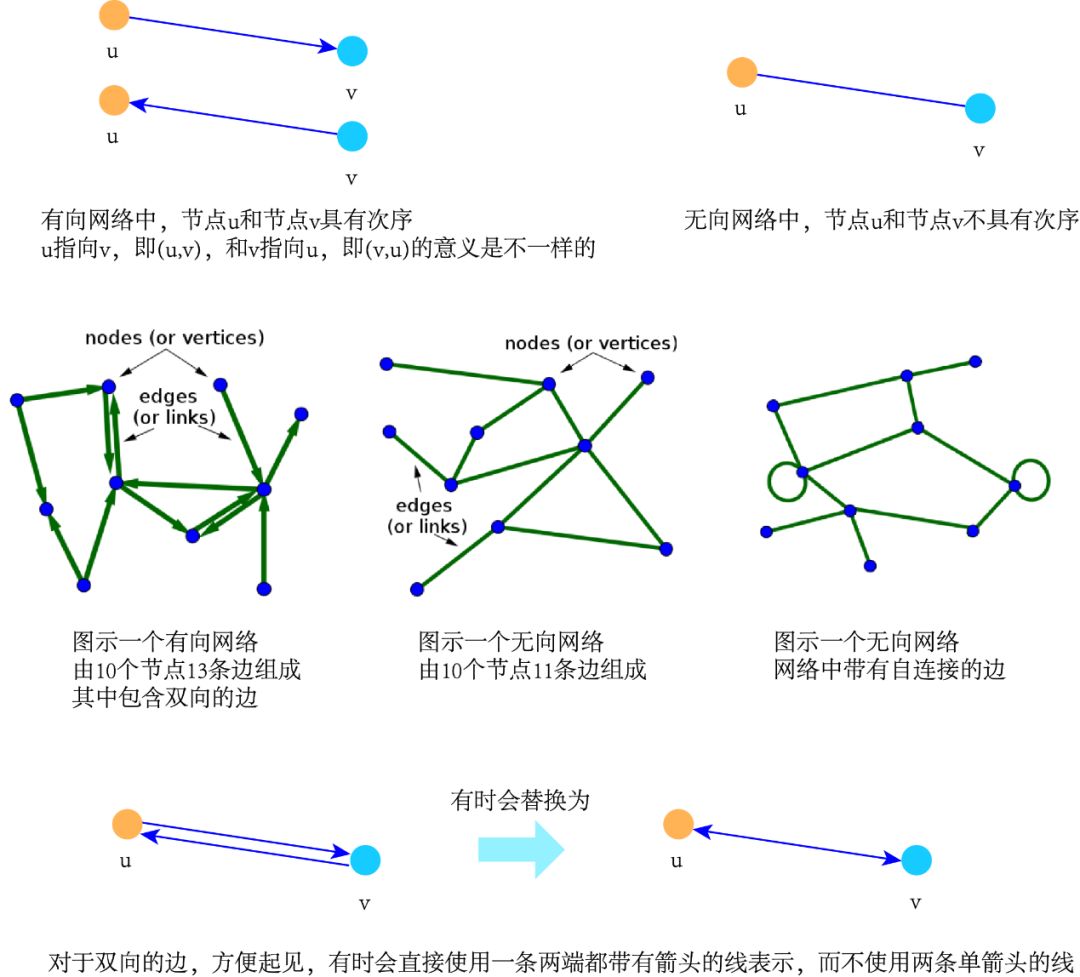
对于网络图G,若集合E中每条边的两个节点都有次序(即对于u,v∈V,(u,v)与(v,u)不同),则图G称为“有向图”(directed graph或digraph)。这些边被称为“有向边”(directed edges)或“弧”(ares),有向边(u,v)的方向规定从左到右,即从起点u指向终点v。需要注意的是,有向图中一对节点间可能有两条有向边,分别以其中一个节点作为起点,另一个作为终点,此时,两条有向边称为是“双向”(mutual)的。类似的,对于网络图G,若每条边的两个节点没有次序,则图G称为“无向图”(undirected graph)。在某些情况下,也可能允许节点具有自连接,这意味着在网络图中出现某节点从自身到自身的边,即“自环”(loops)。
若两个节点间存在多余一条边(称为“多重边”,multi-edges)的情况,或者图中出现“自环”的情况,则图被称为“多重图”(multi-graph)。非多重图称为“简单图”(simple graph),其边称为“正常边”(proper edges)。
图示有向图、无向图,以及双向和自连接类型。
有向网络示例,如KEGG代谢通路,已知的生化反应代谢过程。有向边两侧的节点一个代表底物,一个代表产物。其中很多双向的边,代表了可逆的生化过程。

无向网络示例,如在RNA-seq分析中,基于某种数学模型构建的基因共表达网络,常用无向网络表示。节点代表基因,边代表了某个基因表达量的上调/下调和另外一个基因的表达量显著相关。
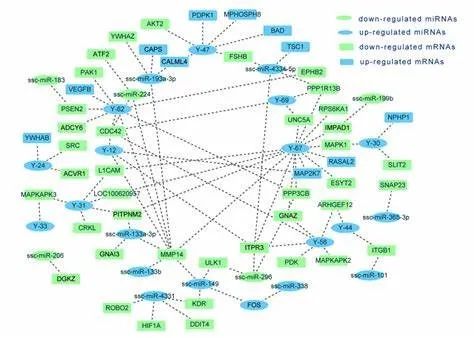
(文献出处:Comparative Anterior Pituitary miRNA and mRNA Expression Profiles of Bama Minipigs and Landrace Pigs Reveal Potential Molecular Network Involved in Animal Postnatal Growth)
节点、边和图的属性和特征
网络图是利用网络表示复杂系统数据的核心,但其它相关的数据也常常需要处理。从网络的视角出发,其它的数据可以被视为“属性”(attributes),即与相应的网络图所关联的值。将一个图与这些属性联系起来的过程,称为图的“修饰”(decorating)。通常图的节点或边(或者两者同时)会具有属性,图作为一个整体也可以有属性。
在上述所展示的示例图中,能看到这些网络的节点以不同的形状、大小、颜色表示,边也被表示为不同类型、粗细、颜色的线条。事实上,这正是展示了节点和边的属性信息。在上图示例的无向网络图中,节点形状表示了RNA类型,颜色表示了RNA表达量上调/下调类型。根据节点、边的属性信息,在绘制网络图时,即可呈现到更丰富的内容。再举一例,如下所示,基于随机矩阵理论构建的大豆根瘤内生细菌共发生网络,按模块划分为圈图,节点代表OTUs,节点颜色代表OTUs所属的门分类,边的颜色代表了OTUs间的正负相关;对于特殊的OTUs(根瘤菌),以特殊形状表示。
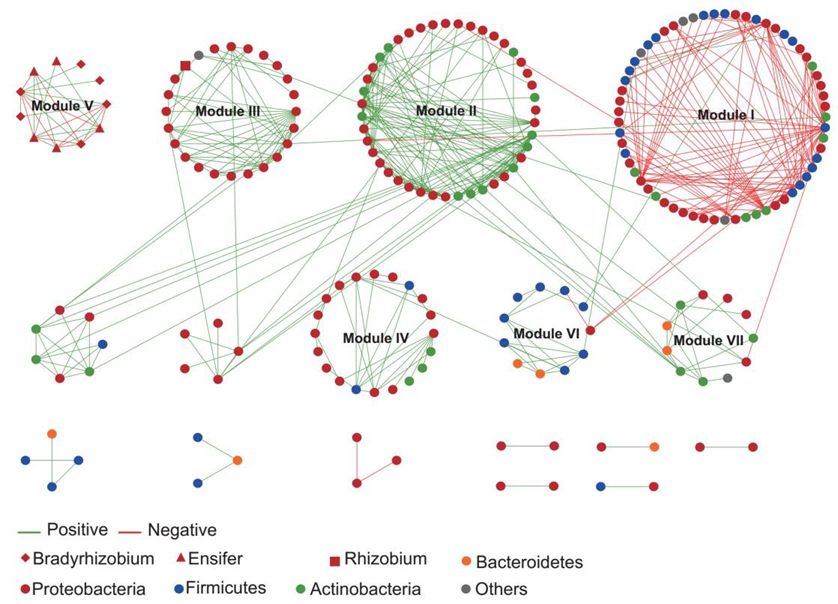
(文献出处:Distinct biogeographic patterns of rhizobia and non-rhizobial endophytes associated with soybean nodules across China)
节点属性
节点属性是以节点作为索引的变量,既可以是离散的也可以是连续的。如在微生物互作网络中,每一个节点代表了一类微生物(如OTU、属、门),或者其它非物种属性,如功能基因、环境变量等,这些节点包含多种各自的属性特征。
如下示例,表格记录了某微生物互作网络中的部分节点属性信息,包括物种信息以及环境因子属性信息。node,网络节点id;phylum,OTU所属的门分类(环境因子记作env);genus,OTU所属的属分类(环境因子记作env);value,OTU的相对丰度,环境因子的测量指标;type,节点属于物种类型(OTU),还是环境因子类型(env)。

在实际的网络分析中,各节点属性往往作为附加内容在后续添加至已构建好的网络中,帮助我们更好地识别网络结构。
边属性
与节点属性类似,边属性是以相邻的节点对(也就是边)作为索引的变量,包括离散和连续两种情况。离散的边属性例子,如在微生物相关性网络中,两个物种之间是正相关(共发生)还是负相关(共排斥)关系。连续的边属性常用来表示某种节点对之间关系的强度,例如在微生物相关性网络中,两个物种之间的相关系数。
边属性在很多分析中可以认为是权重(weighted)。按照惯例,边的权重一般是非负的,且常被缩放至0到1的区间内。如在微生物相关性网络中,可以物种之间相关系数的绝对值定义为边的权重。边具有权重的图称为“含权图”(weighted graph)。
如下示例,表格记录了某微生物互作网络(相关性网络)中的部分边属性信息,包括物种-物种以及物种-环境因子相关性信息。edge,记录了哪两个节点之间存在边连接;type,边类型,该条边是物种-物种互作还是物种-环境因子互作;correlation,相关性网络构建时所得到的物种-物种相关性以及物种-环境因子相关系数,数值表示相关性强弱,符号表示正、负相关;weight,边的权重,这里表示了相关系数的绝对值。

同节点属性,在实际的网络分析中,边属性也往往作为附加内容在后续添加至已构建好的网络中,帮助我们更好地识别网络结构。
节点、边和图的拓扑结构
网络拓扑结构涉及内容较多,篇幅原因就不作详细介绍了,基本概念详见:
节点和边的拓扑结构:http://blog.sciencenet.cn/blog-3406804-1193906.html
网络图的拓扑结构:http://blog.sciencenet.cn/blog-3406804-1193908.html
后续我会再修改完善,同步到公众号上。
通常,在研究一个复杂系统时,感兴趣的问题可以重新表述,转化为某些与对应网络图的结构或特征相关的问题。例如,涉及信息或商品流通的问题通常可以表述为网络图的路径和路径中的流;系统中某个元素的“重要性”可以通过度量网络中相应节点“靠近中心”的程度来表示;寻找系统中的“社团”以及类似的未指明的“团体”,则常作为图分割问题进行分析;等等。
在这一方面,还需多查阅资料,多阅读文献,明确自己关注的问题,选择合适的网络分析手段,以对科学问题作出合理的解释。
网络图的表示及处理
网络分析的基本数据格式及分析工具简介。
网络数据文件
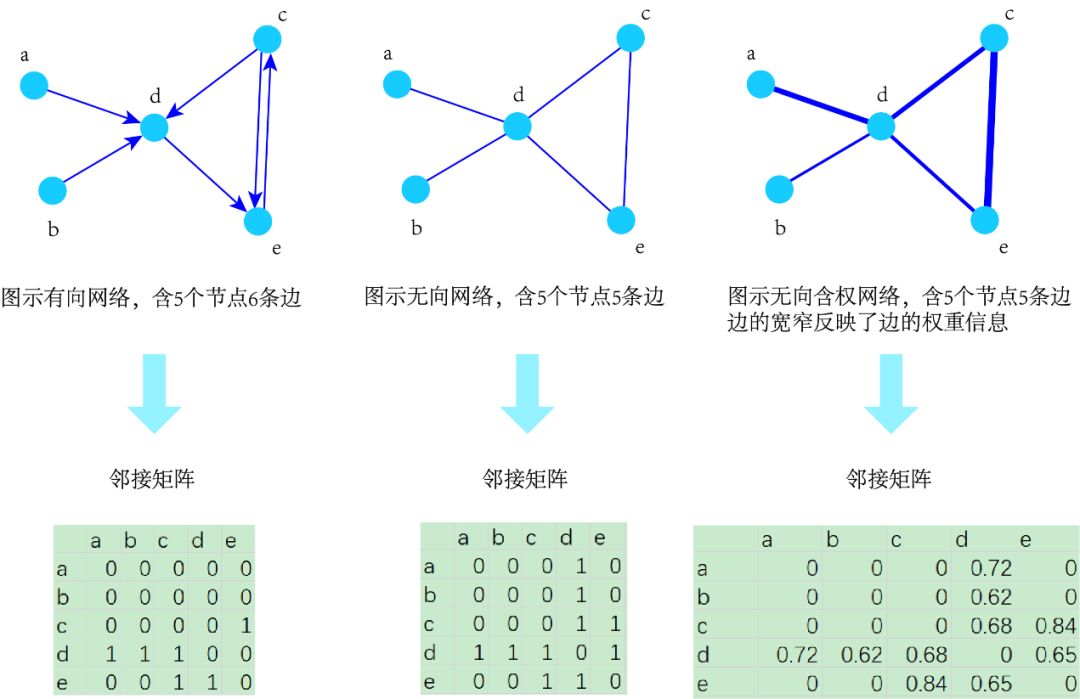
现实中,并不能经常手动地输入一个图,因为大多数现实网络的节点和边数量即是不是几万个(或者几百万个),至少也有几十个。而构建网络图所需要的信息通常存储在数据文件里。从最基本的角度说,存在三种表示图的格式:邻接列表、边列表及邻接矩阵。
邻接列表
图G的“邻接列表”(adjacency list)是一个大小为Nv,按照V中节点的顺序排列的序列。序列的每个元素都是一个列表,若存在i为起点j为终点的边,则第i个列表是所有的终点节点j的集合。
这是igraph常用的一种表示方法,该数据结构不便展示,还请大家自行了解。如使用R包igraph,str()查看网络图。
边列表
“边列表”(edge list)是一个简单的两列列表,给出了所有的边连接的节点对。在边列表中,记录边的形式是由节点u指向节点v(source -> target)。若为有向网络,u指向v和v指向u的意义是不同的;若为无向网络,则u指向v和v指向u是等同的。
边列表中有时会含有额外的内容,作为记录边的属性信息,如权重等。因此,边列表也经常作为边属性文件使用。
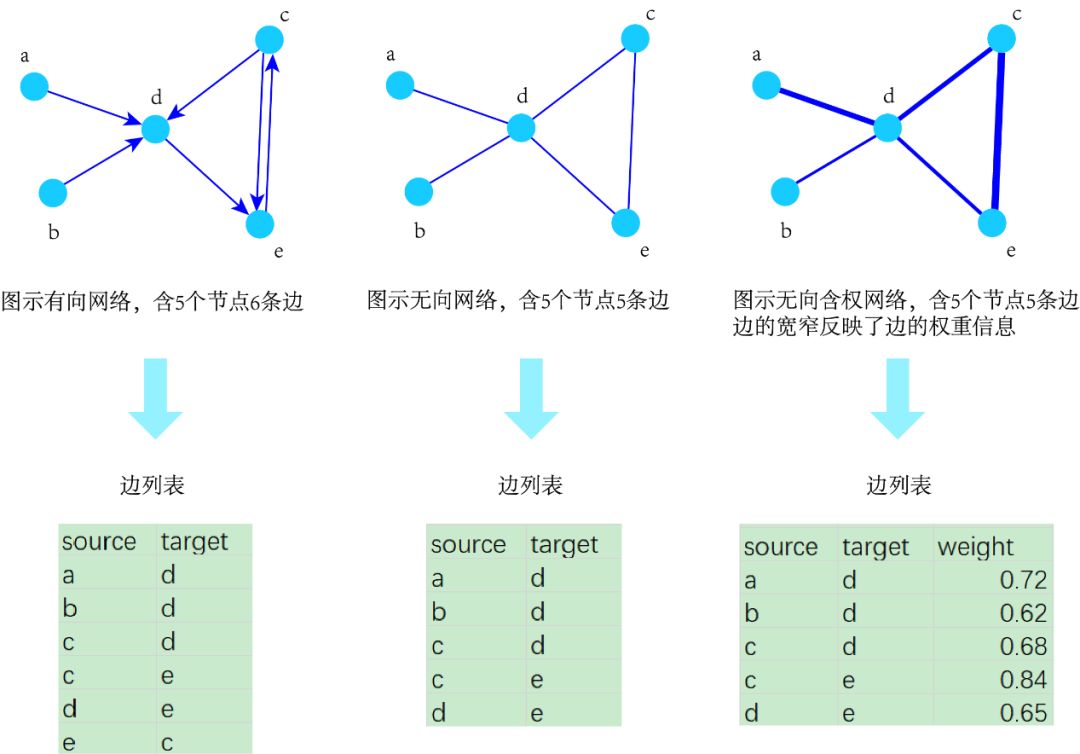
邻接矩阵
图也可以用矩阵的形式存储。对于图G=(V,E),一个大小为Nv×Nv的“邻接矩阵”(adjacency matrix)A定义如下:

若矩阵A行列标号分别为(i,j)的元素非零,则图G中对应存在一条由i指向j的边:若该元素为零,则不存在边。对于无向图,矩阵A是对称矩阵。
对于非零元素,可以直接使用“1”表示,表示两个节点间存在连接;更具体些,使用具体的数值,代表了边的权重(含权网络)。两个节点间不存在边,一定要标识为0。
其它
除此之外,经常还能见到一些特殊的文件类型(它们常带特殊后缀名,以便于识别,如graphml、gml等),这些一般都是方便特定软件编辑的样式。可以通过特定的网络分析工具打开它们,非常方便地执行后续的分析、编辑、可视化等。同样地,若有需要,它们也可以转化为边列表、邻接矩阵等样式。
网络分析及可视化的常用工具
在得到网络图后,需要对网络结构作深入统计分析,以用于解释科学现象,并实现较好的可视化效果作为呈现。以下列举了常用的几种工具。
R,例如igraph(https://igraph.org/)。igraph是一个经典的网络分析R包,它包含一些列数据类型和函数,可以(相对)直接地实现图的算法或者进行算法模型的快速开发,还能够对大型网络进行快速的处理。igraph支持读写等多种网络图格式,如tsv、csv、graphml、gml、lgl、dot、pajek等。除了igraph,R中还有很多网络分析包可供选择,例如network3D绘制交互式网络图等,更多的就不再举例了。使用R进行网路分析有很多优点,如可编程的操作方式、灵活多变、丰富的图形文件格式、大数据下非常节约时间等。
Python,同样是编程的操作方式。作为数据分析的常用工具(特别是在大数据处理中),Python中同样存在很多进行网络分析的模块,如networkx(https://pypi.org/project/networkx/)等。
Graphviz(http://www.graphviz.org/)是一个经典的图布局和绘制工具。它包含一系列独立的命令行程序,可以通过Regraphviz扩展包使用。
Pajek(http://vlado.fmf.uni-lj.si/pub/networks/pajek/)是最早出现的网络分析与可视化工具之一。在windows操作系统下,它有一个单独的程序,并支持互动地绘图和编辑。Pajek有自己的文件格式,可以通过igraph的write.graph和read.graph函数处理(但有一些限制),因此在R和Pajek之间交换数据通常比较简单。
Cytoscape(https://cytoscape.org/)是一个多平台的网络分析与可视化工具,使用Java编写,多用于生物网络。Cytoscape是目前最常用的网络分析工具之一,它实现了不少布局算法,并提供复杂的互动绘图方式,开发十分活跃。Cytoscape提供了很多实用的插件,我们即可使用Cytoscape构建网络图(如CoNet),也可作统计运算,以及可视化处理等。除了使用自己的程序构建网络图外,Cytoscape同样支持通过文本文件从外部导入数据表格,在网络图有很多属性时非常方便处理。igraph的write.graph输出的“gml”格式的网络文件可直接读入至Cytoscape编辑。
Gephi(https://gephi.org/)也是一个多平台的网络分析与可视化工具,使用Java编写,有复杂的互动编辑功能,和Cytoscape一起同为目前最常用的网络分析工具之一。Gephi同样提供了很多实用的插件,可以非常方便地对网络图执行统计运算,以及可视化处理等。igraph的write.graph输出的“graphml”格式的网络文件可直接使用Gephi编辑;同样地,Gephi也支持通过文本文件从外部导入数据表格。
除此之外,尚有许多其它的网络分析工具,还请大家自行了解了。
* R包igraph的基本操作
末了先简单展示一下最最基本的操作,包括文件读写、格式转换、数据结构等,后续的章节将主要使用igraph包处理数据,并会再详细介绍更多操作。
示例数据和R代码链接(提取码:mab5):
https://pan.baidu.com/s/1GR7gwPDwtlgNECZF3t2uzQ
##输入数据示例,邻接矩阵
#带权重的邻接矩阵
adjacency_weight <- read.delim('adjacency_weight.txt', row.names = 1, sep = '\t', check.names = FALSE)
adjacency_weight
#不带权重的邻接矩阵
adjacency_unweight <- read.delim('adjacency_unweight.txt', row.names = 1, sep = '\t', check.names = FALSE)
adjacency_unweight
##格式转换示例
#带权重邻接矩阵 -> igraph 的邻接列表,获得含权的无向网络
igraph_weight = graph_from_adjacency_matrix(as.matrix(adjacency_weight), mode = 'undirected', weighted = TRUE, diag = FALSE)
igraph_weight
is.weighted(igraph_weight)
#不带权重的邻接矩阵 -> igraph 的邻接列表,获得非含权的无向网络
igraph_unweight = graph_from_adjacency_matrix(as.matrix(adjacency_unweight), mode = 'undirected', weighted = NULL, diag = FALSE)
igraph_unweight
is.weighted(igraph_unweight)
##网络图绘制
plot(igraph_weight, layout = layout.circle)
plot(igraph_unweight, layout = layout.fruchterman.reingold)
##节点和边操作,以含权网络为例
#节点操作,例如将 OTU 人为地分为两组
otu_num <- nrow(adjacency_weight)
group1 <- round(otu_num/2)
group2 <- otu_num - group1
V(igraph_weight)$color <- c(rep('purple', group1), rep('orange', group2))
plot(igraph_weight, layout = layout.circle)
#边操作,例如边权重代表了 OTU 相关性,根据相关性正负分为两组
weight <- E(igraph_weight)$weight #查看边的权重
color <- rep('', length(weight))
color[which(weight > 0)] <- 'red'
color[which(weight < 0)] <- 'green'
E(igraph_weight)$color <- color
plot(igraph_weight, layout = layout.circle)
##输出节点属性和边属性列表,以含权网络为例
#节点属性列表,输出 OTU 名称,以及上述赋值的颜色
node_list <- data.frame(
id = names(V(igraph_weight)),
color = V(igraph_weight)$color
)
node_list
write.table(node_list, 'node_list.txt', sep = '\t', row.names = FALSE, quote = FALSE)
#边属性列表,输出相关的 OTU 的关系,代表相关系数的边权重,以及上述赋值的颜色
edge <- data.frame(as_edgelist(igraph_weight)) #igraph 的邻接列表转为边列表
edge_list <- data.frame(
source = edge[[1]],
target = edge[[2]],
weight = E(igraph_weight)$weight,
color = E(igraph_weight)$color
)
write.table(edge_list, 'edge_list.txt', sep = '\t', row.names = FALSE, quote = FALSE)
参考资料
Eric D Kolacayk, Gabor Csardi, 网络数据的统计分析:R语言实践(李杨 译). 西安交通大学出版社, 2016.
Math Insight:https://mathinsight.org/index/general
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
文献阅读 热心肠 SemanticScholar Geenmedical
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读























 7万+
7万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








