沉积物微宇宙中磺胺甲恶唑生物强化去除的新见解:去除效率、生态风险和微生物机制
New insights into bioaugmented removal of sulfamethoxazole in sediment microcosms: degradation efficiency, ecological risk and microbial mechanisms

Article,2024-02-29,Microbiome,[IF 15.5]
DOI:10.1186/s40168-023-01741-5
原文链接:https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-023-01741-5
第一作者:Jianfei Chen (陈建妃)
通讯作者:Shuguang Xie (谢曙光)
主要单位:
福建师范大学环境与资源学院 (Fujian Key Laboratory of Pollution Control & Resource Reuse, College of Environmental and Resource Sciences, Fujian Normal University, Fuzhou 350007, China)
北京大学环境科学与工程学院 (State Key Joint Laboratory of Environmental Simulation and Pollution Control, College of Environmental Sciences and Engineering, Peking University, Beijing 100871, China)
2024年2月29日,北京大学环境科学与工程学院谢曙光和福建师范大学环境与资源学院陈建妃等团队在国际著名微生物学期刊Microbiome在线联合发表了题为“New insights into bioaugmented removal of sulfamethoxazole in sediment microcosms: degradation efficiency, ecological risk and microbial mechanisms”的研究文章。该研究从磺胺甲恶唑降解效率、抗生素抗性基因传播风险以及对细菌群落的影响等方面,评估了利用磺胺甲恶唑降解菌进行生物强化处理的可行性,并强调了利用拟节杆菌R1进行生物强化的优势。另外,利用稳定同位素探针技术(SIP)识别了生物强化体系中参加磺胺甲恶唑的原位活性降解菌,揭示了外源降解菌在强化体系中的功能地位和强化策略。研究成果为优化磺胺类抗生素的生物强化处理技术提供重要科学依据。
- 摘要 -
微生物强化降解是去除磺胺甲恶唑的重要手段,但外源降解菌在强化体系中的低存活性限制了其实际应用。为进一步提高和优化生物强化处理性能,本研究采用DNA-SIP和宏基因组分析等方法,探讨了分别接种了两种重要的磺胺甲恶唑降解菌(Pseudomonas sp. M2或Paenarthrobacter sp. R1)的稳定塘沉积物微宇宙的生物强化机制。结果表明,接种菌株M2和菌株R1(尤其是R1)显著提高了磺胺甲恶唑的生物降解率,且其生物降解能力在一定周期内(经过3次重复添加磺胺甲恶唑)是可持续的。另外,外源降解菌的生物强化策略也显著降低了抗生素抗性基因的积累和传播风险。接种菌株M2显著降低了沉积物细菌多样性,并改变了沉积物细菌群落;而接种菌株R1对细菌群落的影响较小且与土著微生物建立了密切的互作关系。DNA-SIP分析结果证明,在两种生物强化体系中,拟节杆菌都被鉴定为主要的磺胺甲恶唑同化细菌。结合基因组信息和纯培养证据,菌株R1通过直接参与磺胺甲恶唑降解来促进其去除,而菌株M2通过参与磺胺甲恶唑降解和刺激土著微生物(尤其是拟节杆菌)对磺胺甲恶唑的降解活性而促进磺胺甲恶唑的去除。通过外源降解菌(尤其是R1)进行生物强化处理是去除磺胺甲恶唑的可行的策略。
- 引言 -
磺胺类抗生素是人工合成的一类广谱性抗生素,在人畜疾病的预防和治疗中广泛应用,是最常用的兽用抗生素之一。医药废水、养殖废水和生活污水是环境中磺胺类抗生素的主要来源,磺胺类抗生素在地表水、沉积物和土壤等环境中均有检出。由于其对环境微生物群落结构和功能以及抗生素抗性基因累积和传播等方面的负面影响,已经引起人们对其环境污染和治理的关注。磺胺类抗生素在环境中的自然衰减比较缓慢,开发促进磺胺类抗生素快速完全降解的策略对污染治理具有重要意义。
微生物降解是环境中磺胺类抗生素去除的主要途径,因此强化微生物降解可能是提高磺胺类抗生素污染修复的有效措施。微生物强化技术(Bioaugmentation)通过向污染场地投加特定功能微生物,从而能够增加环境中特定污染物的去除效果,目前已在土壤修复和活性污泥处理等多个领域成功实施。国内外研究者已经分离培养了大量磺胺类抗生素的降解菌,但这些菌的降解机制的研究主要基于液体培养基试验,关于投加磺胺类抗生素降解菌进行生物强化去除抗生素的研究仍十分有限。外源降解菌在土著微生物群落中存在竞争力弱、存活难、持效性低等问题,这大大限制了其规模化应用。而外源菌和土著微生物的相互作用关系及其促进磺胺类抗生素降解的机制的解析,有助于解决这一问题,但是目前仍缺乏这方面的研究。此外,磺胺类抗生素作为一类具有光谱抑菌性的抗生素,在强化处理过程中可能会引起为微生态变化和抗生素抗性基因及可移动遗传元件的富集,也是需要共同关注的问题。
因此,本研究的总体目标是开发一种利用外源磺胺类抗生素降解菌(拟节杆菌R1和假单胞菌M2)修复沉积物中磺胺类抗生素生物强化的方法,并阐明其相关微生物学机制。主要包括以下几个方面的内容:(1)处理效果以及持续性;(2)抗性基因累积和传播风险;(3)对土著微生物群落结构的影响;(4)增强生物降解的微生物学机制。
- 结果 -
磺胺甲恶唑降解效率
Characteristics of Sulfamethoxazole Degradation
试验设计5个分组,(A)灭菌对照;(B)空白对照(未加磺胺甲恶唑和降解菌);(C)未接种菌对照;(D)假单胞菌M2生物强化试验组;(E)拟节杆菌R1生物强化试验组。试验进行3个批次,分别在第0、8和10天加入10 mg/L磺胺甲恶唑。磺胺甲恶唑浓度监测结果表明,非生物处理对磺胺甲恶唑的去除率贡献不大,未接种的沉积物微生物具有一定的磺胺甲恶唑降解能力,但去除率很低(仅为10%)。假单胞菌M2或拟节杆菌R1的生物强化都加速了磺胺甲恶唑的生物降解,尤其是R1,生物强化效果在3个循环中有效。

图1 生物强化体系中磺胺甲恶唑的降解特性。
细菌群落的时间动态和相互作用网络
Temporal Dynamics and interaction networks of bacterial communities
Alpha多样性分析结果显示,假单胞菌M2的加入显著降低了沉积物微生物群落的多样性,而拟节杆菌R1的加入增加了Observed Features指数。基于Weighted_Unifrac相似性分析的结果表明,生物强化(尤其是接种假单胞菌M2)显著改变了微生物群落结构。其中,Paenarthrobacter(拟节杆菌)、Flavobacterium、Azohydromonas和f_Rhodocyclaceae.g_unclassified等菌属在接种R1的生物强化体系中相对丰度更高,而Pseudomonas(假单胞菌)在接种M2的生物强化体系中相对丰度更高。
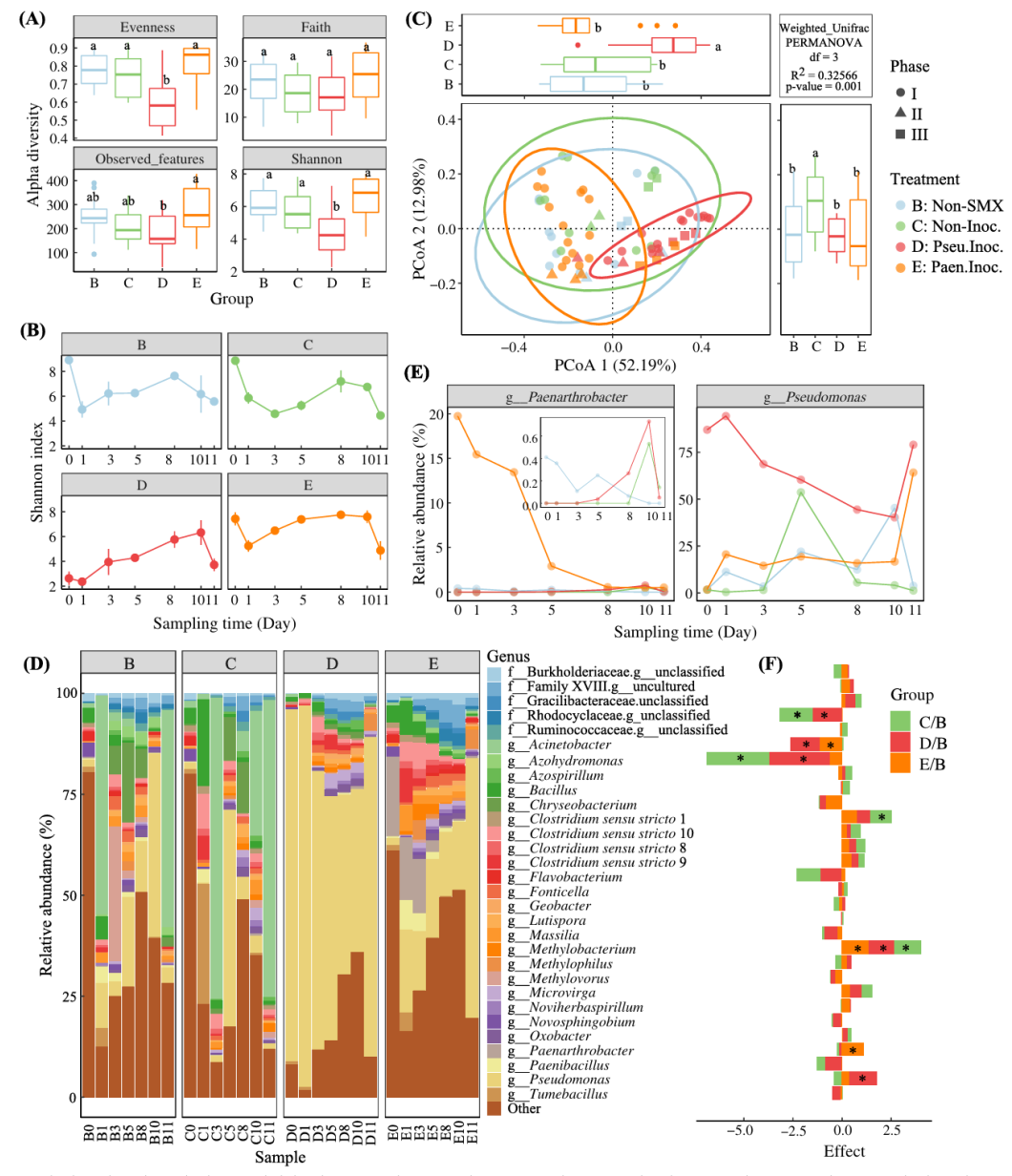
图2
(A)细菌alpha多样性箱线图;
(B)Shannon指数随时间变化图;
(C)基于Weighted_Unifrac距离的PCoA图;
(D)主要菌属的相对丰度图;
(E)Pseudomonas sp. M2和Paenarthrobacter sp. R1的时间动态。
分子生态网络分析结果表明,磺胺甲恶唑的加入降低了网络的复杂性(平均程度降低)、紧密性(平均聚类系数降低、平均路径距离增加)和模块化(模块数减少),但促进了合作关系的发生(正相关比例增加)。接种假单胞菌M2减轻了这种影响,而接种拟节杆菌R1则加剧了这种情况。另外,在未添加磺胺甲恶唑、未接种降解菌、接种假单胞菌M2和接种拟节杆菌R1这4个处理中,分别确定了8、3、4和6个关键类群。除Acinetobacter和f_Planococcaceae. g_unclassified外,不同处理中未出现共同的关键类群。更重要的是,拟节杆菌被识别为接种R1体系中的模块枢纽,与土著微生物之间形成了紧密的相互作用关系。然而,假单胞菌未被确定为关键类群,仅与群落中少数微生物具有相关性。

图3
(A)基于成对Pearson相关性的细菌共现网络图(ρ>0.6);
(B)细菌拓扑分布
功能基因、抗生素抗性基因和可移动遗传元件丰度
Abundance of functional genes, antibiotic resistance genes, and mobile genetic elements
细菌16S rRNA基因在培养1天后迅速增加,之后保持相对稳定。此外,磺胺甲恶唑的加入显著增加了细菌生长。磺胺类抗生素生物降解单加氧酶编码基因(sadA基因)数量随培养时间先增加后减少,且sadA基因拷贝数在不同处理间无显著差异。磺胺类抗生素抗性基因sul1基因,在未添加磺胺甲恶唑和未接种降解菌两个处理中,数量分别呈现缓慢和快速增加趋势,而在接种假单胞菌M2和接种拟节杆菌R1两个处理中,呈现先快速增长随后随着磺胺甲恶唑的降解而降低,在重新加入磺胺甲恶唑之后数量略有增加。另外一种磺胺类抗生素抗性基因,sul2基因,数量也随着培养时间的增加而增加,且其数量在未接种和接种假单胞菌M2处理中更高(p<0.05)。定量PCR的结果表明,磺胺类抗生素抗性基因在不同处理中的大小顺序依次为:未接种菌>接种假单胞菌M2>接种拟节杆菌R1>空白对照。宏基因组分析进一步表明,加入磺胺甲恶唑降解菌进行生物强化可以降低抗生素抗性基因的积累和传播风险。如图4C和4D所示,未接种菌体系中的抗生素抗性基因和可移动遗传元件丰度均明显高于其他处理,分别是空白对照、接种假单胞菌M2和接种拟节杆菌R1处理的4.2和5.3倍、3.9和2.1倍、6.2和2.8倍。此外,值得注意的是,磺胺甲恶唑的污染不仅导致磺胺类抗生素抗性基因的积累,而且促进了四环素、多药、氯霉素和氨基糖苷类抗性基因的丰度,以及整合酶、质粒和转座酶等可移动遗传元件的丰度。
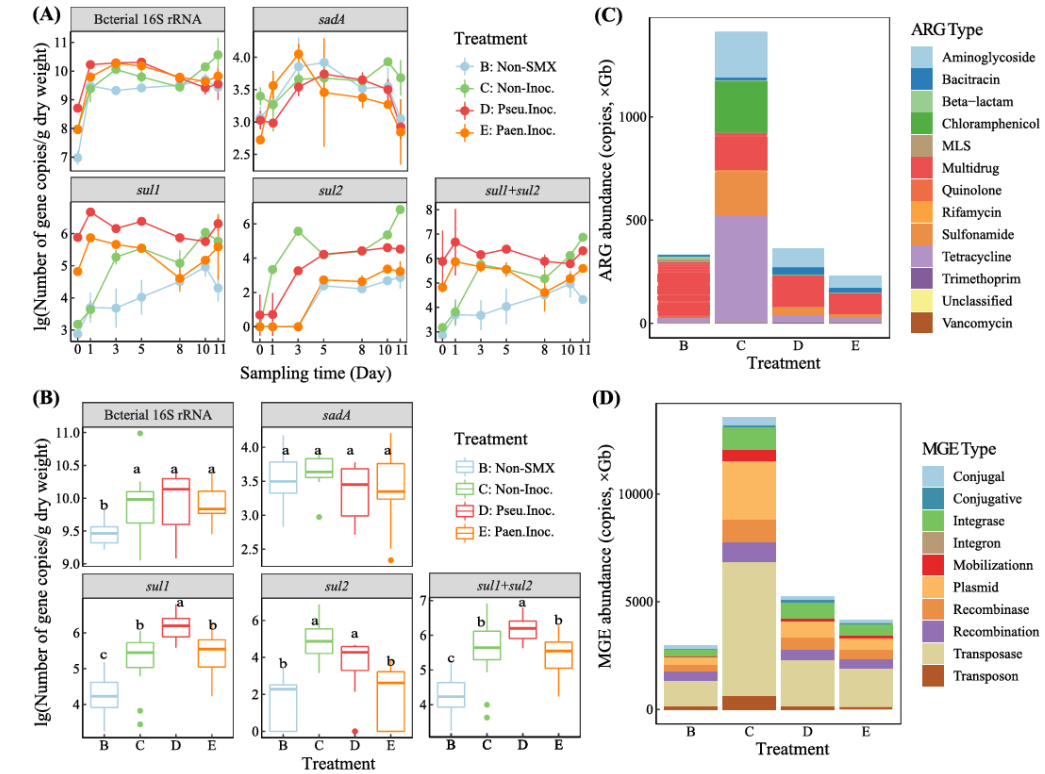
图4
(A)基于qPCR结果的细菌16S rRNA基因、磺胺类抗生素降解基因(sadA基因)和抗性基因(sul1和sul2)的时间动态和(B)箱线图;
(C)基于宏基因组测序的抗生素抗性基因和(D)可移动遗传元件的相对丰度。
基因组分析发现,假单胞菌M2和拟节杆菌R1的基因组上含有sul1基因,这解释了在两菌加入的初始阶段时sul1基因数量快速增加的原因。外源降解菌直接引入抗性基因,是抗生素生物强化应用需要考虑的重要问题。本研究的结果表明,虽然生物强化直接将sul1基因引入到沉积物中,但是最终通过磺胺甲恶唑的快速降解可以显著降低抗性基因的传播风险。
DNA-SIP识别活性磺胺甲恶唑降解菌
Active Sulfamethoxazole Degraders Revealed by DNA-SIP

图5
(A)细菌16S rRNA基因密度梯度分馏特征;
(B)拟节杆菌R1和(C)假单胞菌M2生物强化处理的潜在磺胺甲恶唑降解菌的密度梯度分馏特征。
为进一步揭示外源降解菌促进磺胺甲恶唑去除的微生物机制,本研究采用DNA-SIP方法鉴定了生物强化过程中参与磺胺甲恶唑生物降解的活性微生物。分别有23种和4种细菌被鉴定为假单胞菌M2和拟节杆菌R1生物强化处理体系中的功能性磺胺甲恶唑降解菌。在假单胞菌M2生物强化处理体系中,磺胺甲恶唑降解菌多样性较高,但大多数微生物的平均丰度低于1%,其中,拟节杆菌是优势降解菌。虽然假单胞菌在强化处理体系中丰度很高,但是它没有被13C-磺胺甲恶唑标记,表明假单胞菌M2可能仅通过促进土著微生物对磺胺甲恶唑的代谢来提高磺胺甲恶唑的去除。此外,在拟节杆菌R1生物强化处理体系中,Paenarthrobacter(拟节杆菌)、Methylophilus、Methylobacterium和Caulobacter被鉴定为磺胺甲恶唑的活性降解菌,拟节杆菌也是参与磺胺甲恶唑降解的最重要的功能微生物。因此,拟节杆菌R1主要通过直接参与磺胺甲恶唑的生物降解来促进磺胺甲恶唑的去除。
磺胺甲恶唑同化途径预测
Prediction of sulfamethoxazole assimilation pathway
基于宏基因组分箱分析,得到了91个基因组,包括SIP识别的6种磺胺甲恶唑降解菌(Bin16_Noviherbaspirillum、Bin35_Paenarthrobacter、Bin52_Caulobacter、Bin64_Geobacter、Bin74_Opitutus和Bin88_Methylophilus)。根据磺胺甲恶唑降解菌基因组和我们前期的研究结果,推测了生物强化过程中磺胺甲恶唑的同化途径。首先,磺胺甲恶唑通过sadABC基因编码的黄素依赖性单加氧酶和FMN还原酶转化为1,2,4-三羟基苯。1,2,4-三羟基苯通过hydroxyquinol 1,2-dioxygenase、maleylacetate reductase、3-oxoadipate CoA-transferase、acetyl-CoA acyltransferase和3-oxoadipyl-CoA thiolase等酶转化为琥珀酰辅酶A并最终进入TCA循环。其中,磺胺甲恶唑的初始转化相关功能基因sadABC基因仅在拟节杆菌基因组中检测到,表明其在磺胺甲恶唑初始转化中的重要作用。此外,所有与推测的磺胺甲恶唑同化途径相关的基因都在拟节杆菌的基因组上检测到,这与其在两种生物强化处理体系中被识别为最主要的磺胺甲恶唑降解菌结果一致。Noviherbaspirillum基因组包含除sadABC基因外的所有相关基因,在假单胞菌M2强化处理体系中也发挥了重要作用。此外,其他13C标记微生物主要参与TCA循环步骤(如拟节杆菌R1强化处理体系中的Caulobacter、以及假单胞菌M2强化处理体系中的Geobacter、Methylophilus和Opitutus)。最后但同样重要的是,虽然假单胞菌没有被13C-磺胺甲恶唑标记,也不含有sadABC基因,我们前期基于纯培养的研究已经其可以催化磺胺甲恶唑的对位羟基化,并利用磺胺甲恶唑作为唯一的碳源。因此,在假单胞菌M2强化处理体系中,磺胺甲恶唑的初始转化也可能是由假单胞菌驱动的,其中间产物随后被其他13C标记的微生物(如Paenarthrobacter和Noviherbaspirillum)同化。

图6
(A)SIP预测的磺胺甲恶唑降解菌的基因组bins和参考基因组的系统发育树;
(B)预测的磺胺甲恶唑同化路径
- 结论 -
本研究从磺胺甲恶唑去除效率和抗生素抗性基因传播风险两个方面评估了拟节杆菌R1和假单胞菌M2的生物强化处理效果,并从细菌群落的时间动态、生态网络、原位降解菌和磺胺甲恶唑同化途径等方面探讨了生物强化过程的微生物机制。利用拟节杆菌R1和假单胞菌M2进行生物强化,特别是菌株R1,能够快速去除沉积物微宇宙中的磺胺甲恶唑,并且可以持续至少3个循环。虽然接种外源降解菌直接将sul1基因引入沉积物中,但最终通过磺胺甲恶唑的快速降解可以显著降低抗生素抗性基因的传播风险。由于假单胞菌在细菌群落中的优势地位,菌种M2显著降低了细菌群落的多样性并改变了其结构,与土著微生物的联系并不紧密。与此相反,拟节杆菌R1对土著细菌群落的影响较小,并与各种土著微生物建立了牢固的联系,这有利于其在沉积物中的定殖并发挥功能。DNA-SIP和基因组组装的证据以及我们之前的纯培养实验表明,拟节杆菌R1通过直接参与磺胺甲恶唑降解来增强磺胺甲恶唑的去除,而假单胞菌M2通过直接参与磺胺甲恶唑降解和刺激群落中本地微生物(拟节杆菌)的磺胺甲恶唑降解活性来增强磺胺甲恶唑的去除。总之,本研究表明,利用磺胺甲恶唑降解菌,特别是拟节杆菌R1进行生物强化是一种可行的消减磺胺甲恶唑的策略。
参考文献
Chen, J., Chen, X., Zhu, Y. et al. New insights into bioaugmented removal of sulfamethoxazole in sediment microcosms: degradation efficiency, ecological risk and microbial mechanisms. Microbiome 12, 43 (2024).
- 作者简介 -
第一作者

福建师范大学
陈建妃
副教授
陈建妃,理学博士,副教授,硕士生导师,福建省高层次引进人才(C类);博士毕业于北京大学环境科学与工程学院环境科学专业,2022年8月作为福建省“引进生”加入福建师范大学环境与资源学院;主要研究方向为水环境控制与生物技术,共合作发表SCI论文20余篇,个人H指数15;其中,以第一作者在Microbiome、Water Research、Environmental Microbiology、Environment International等高水平期刊上发表SCI论文10余篇;主持或参与福建省自然科学基金、国家自然科学基金等项目研究。
通讯作者

北京大学
谢曙光
副教授
谢曙光,北京大学环境科学与工程学院副教授、博士生导师。主要从事污染控制、环境生物技术、微生物生态学的研究,作为第一作者或通讯作者已在Microbiome、Environmental Science & Technology、Water Research、Environmental Microbiology、Environment International等SCI期刊发表文章130余篇,H指数39。目前担任国际学术期刊Microbiome、Environmental Microbiome和Frontiers in Bioengineering and Biotechnology的副编辑以及International Biodeterioration & Biodegradation、Toxics、Annals of Microbiology的编委。曾主持国家自然科学基金面上项目、青年科学基金项目、国家重点实验室项目(联合基金、自由课题、开放基金)和省部级重点实验室开放基金,并参与国家重点研发计划政府间国际科技创新合作项目、国家自然科学基金重大研究计划重点支持项目,国家“十一五”和“十二五”水专项、环保部公益项目等多个国家或省部级项目、以及深圳市科技计划项目(技术攻关重点项目)。
宏基因组推荐
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
猜你喜欢
iMeta高引文章 fastp 复杂热图 ggtree 绘图imageGP 网络iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读







 福建师范大学和北京大学的研究团队研究了磺胺甲恶唑在沉积物微宇宙中的生物强化去除,发现接种菌株R1显著提高去除效率,降低了抗生素抗性基因传播风险,并揭示了微生物群落的响应机制。研究强调了拟节杆菌R1在生物强化策略中的优势,为优化磺胺类抗生素处理提供了科学依据。
福建师范大学和北京大学的研究团队研究了磺胺甲恶唑在沉积物微宇宙中的生物强化去除,发现接种菌株R1显著提高去除效率,降低了抗生素抗性基因传播风险,并揭示了微生物群落的响应机制。研究强调了拟节杆菌R1在生物强化策略中的优势,为优化磺胺类抗生素处理提供了科学依据。
















 7943
7943

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








