点击蓝字 关注我们
棉花中高效的CRISPR/Mb2Cas12a基因组编辑系统

iMeta主页:http://www.imeta.science
方法论文
● 原文链接DOI: https://doi.org/10.1002/imt2.209
● 2024年6月4日,华中农业大学金双侠和西南大学张勇团队在iMeta在线联合发表了题为 “Robust CRISPR/Mb2Cas12a genome editing tools in cotton plants” 的文章。
● 本研究旨在在棉花中建立CRISPR/Mb2Cas12a基因编辑系统,进一步评估不同的crRNA表达单元的效率,并最终评估应用多基因编辑的可行性。
● 第一作者:惠凤娇
● 通讯作者:金双侠 (jsx@mail.hzau.edu.cn)、张献龙 (xlzhang@mail.hzau.edu.cn)、张勇 (zhangyong916@swu.edu.cn)
● 合作作者:唐旭、李波、Muna Alariqi、许忠平、孟庆营、胡咏雪、王冠英
● 主要单位:湖北洪山实验室、作物遗传改良全国重点实验室、华中农业大学植物科学技术学院;资源植物保护与种质创新重庆市重点实验室、西部(重庆)科学城种质创制大科学中心、西南大学生命科学学院;新疆农业科学院核技术与生物技术研究所。
亮 点

● CRISPR/Mb2Cas12a系统是棉花中高效、精准的基因编辑工具;
● Mb2Cas12a在22℃至32℃的不同温度下都表现出优异的编辑能力;
● Mb2Cas12a可对棉花基因组中VTTV PAM位点进行更宽松的编辑;
● 基于CRISPR/Mb2Cas12a开发了棉花中的多基因编辑系统,一个载体能够对8个基因位点进行有效编辑。
视频解读
Bilibili:https://www.bilibili.com/video/BV1L4421X71W/
Youtube:https://youtu.be/qiZxmp0u7mA
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
CRISPR/Cas9系统以无与伦比的简便性、高效性和精确性实现靶向突变,迅速发展成为生物技术领域的里程碑。CRISPR/Cas12系统以独有的特点,迅速发展为一种前景广阔的基因编辑工具。最近,CRISPR/Mb2Cas12a被描述为能在植物中进行有效的编辑。重要的是,Mb2Cas12a不仅能识别TTTV(V = A、C、G)PAM,还能识别更宽松的VTTV PAM,从而扩大在全基因组的编辑范围。但是目前这一系统尚未在棉花中进行测试。
陆地棉(Gossypium hirsutum)是一种异源四倍体植物,基因组庞大而复杂,约2.5Gb,大多数基因存在多拷贝。虽然植物中已经开发了多种CRISPR/Cas12a系统,但用于棉花基因编辑的Cas12蛋白仍然很少。目前,在棉花的基因组编辑研究中使用的是混合双启动子系统,其他类型的Cas蛋白和sgRNA/crRNA表达系统能否提高棉花的编辑效率还有待研究。此外,利用Cas9和Cas12a已经在植物中开发出了多基因编辑系统,但在棉花中实现多基因位点编辑的有效性仍有待验证。鉴于此,本研究旨在在棉花中建立CRISPR/Mb2Cas12a基因编辑系统,进一步评估不同的crRNA表达单元的效率,并最终评估应用多基因编辑的可行性。
结 果
CRISPR/Mb2Cas12a在棉花中实现高效的靶向突变
首先,我们构建了一个Mb2Cas12a表达载体,称为Strategy 1。在Strategy 1中,Mb2Cas12a和crRNA表达盒分别由Pol III启动子(pOsUbi2)和Pol II启动子(pGhU6.7)驱动。crRNA与tRNA间隔排列(图 1A)。我们选择了棉花中的视觉报告基因GhPGF和GhCLA1作为目标基因。通过农杆菌介导的遗传转化获得基因编辑再生植株。高通量测序分析显示,T0植株中GhPGF-crRNA1靶标位点的编辑效率高达94.62%(图 1B)。Mb2Cas12a系统的编辑窗口主要在PAM位点下游13到26 nt之间。在所有的编辑编辑事件中都只检测到了DNA片段的缺失,缺失大小主要集中在7到18 bp之间(图 1C)。这些发现与我们之前在棉花中使用LbCas12a的验证结果是一致的,表明Cas12a家族的蛋白中存在着相似的编辑模式。我们观察到叶片和茎中棉酚腺体的密度明显下降,导致大量植株明显的无腺体(图 1D)。同时,GhCLA1-crRNA2靶标编辑的植株叶片表现出部分漂白,编辑效率高达71.34%(图 1B,1D)。值得注意的是,由于GhCLA1编辑位点的纯合突变体无法在土壤中存活,因此所有测试的T0植株都表现出嵌合体表型。
接下来,我们在棉花中评估了另外两种表达系统,Strategy 2和Strategy 3。在Strategy 2中,Mb2Cas12a和crRNA单元分别使用pOsUbi2表达。crRNA表达盒由锤头(HH)核糖核酸酶和肝炎病毒(HDV)核糖核酸酶处理。在Strategy 3中,Mb2Cas12a和crRNA单元同时由一个pOsUbi2驱动,命名为单转录本单元(STU)系统(图 1A)。我们选择了与Strategy 1相同的四个靶标DNA序列来进行编辑效率的探索。与Strategy 1相似,Strategy 2和Strategy 3在GhPGF-crRNA1位点的编辑效率分别高达94.33%和91.95%(图 1B),同时表现出与Strategy 1相似的编辑模式和明显的表型(图 1D)。在GhCLA1-crRNA2目标位点也检测到了明显的编辑事件,编辑效率分别为47.81%和78.18%,另外两个靶位点也实现了高效编辑(图 1B)。
我们的研究表明,CRISPR/Mb2Cas12a是一种高效率的棉花基因编辑工具。它可以成功地与三种不同的crRNA阵列结合使用:tRNA-crRNA-tRNA、HH-crRNA-HDV和STU。此外,我们的研究结果表明,在含有单个crRNA的表达盒中无论使用Pol II还是Pol III启动子,编辑效率基本保持一致。
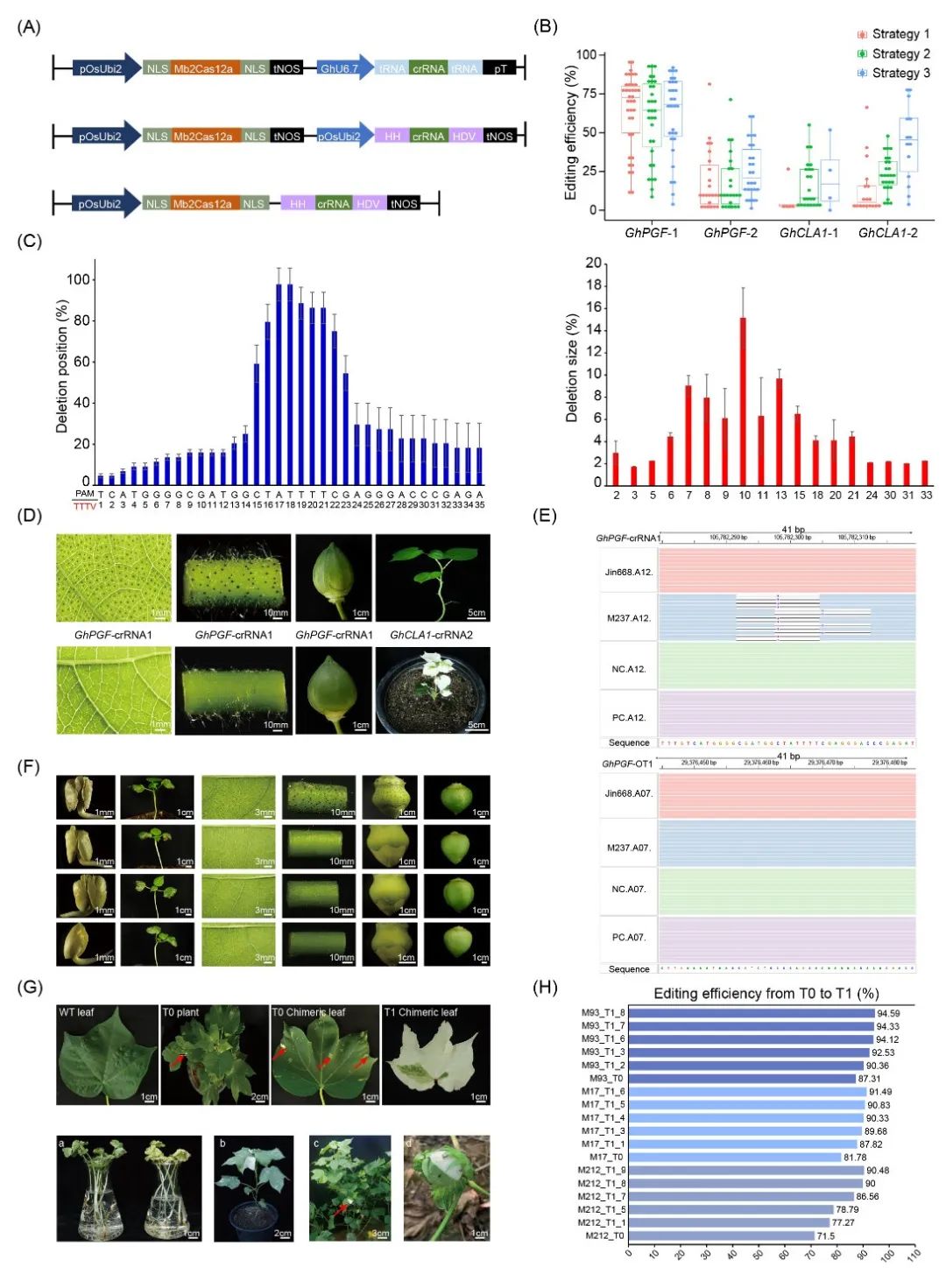
图1. 棉花中开发的高效CRISPR/Mb2Cas12a系统
(A)CRISPR/Mb2Cas12a双元载体T-DNA区域示意图。pOsUbi2:水稻泛素启动子;NLS:核定位信号;pT:polyT;GhU6.7:棉花U6.7启动子;HH:锤头核酶;HDV:肝炎病毒核酶;tNOS:NOS终止子。(B)使用不同载体编辑GhPGF和GhCLA1靶标位点的编辑效率。所有数据均来自二代高通量测序(NGS)。每个点代表一个独立样本的编辑效率。(C)Mb2Cas12a诱导的GhPGF-crRNA1靶标位点DNA缺失位置和大小的频率。PAM序列用红色标出,crRNA用黑色标出。(D)GhPGF和GhCLA1靶标突变的T0棉花植株表型。黑点代表棉花植株上的棉酚腺体。GhCLA1-crRNA2靶标位点T0敲除植株表现出叶片上散布白色区域的嵌合体表型。(E)通过全基因组测序技术评估潜在的脱靶编辑。通过Integrative Genomics Viewer(IGV)可视化靶标位点GhPGF-crRNA1和非靶标位点GhPGF-OT1的序列比对。(F)GhPGF-crRNA1位点T1株系的表型特征。黑点表示T1种子、幼苗、叶片、叶柄、花蕾、棉铃和WT对照样本表面的棉酚腺体。(G)GhCLA1-crRNA2位点突变的T1棉花植株在不同环境下出现漂白现象。(H)利用NGS测定T1株系中GhPGF-crRNA1靶标位点的编辑效率。
Mb2Cas12a在棉花中表现靶向特异性和遗传稳定性
为了评估Mb2Cas12a的特异性,Sanger测序和全基因组重测序分析表明,在预测的所有脱靶位点都没有检测到脱靶突变(图 1E)。此外,植株中编辑位点的基因型和表型忠实地从T0遗传到T1株系,并且在T1中观察到了一些新的编辑事件,这是因为在一些T1株系植株中仍存在Mb2Cas12a蛋白,从而在现有编辑位点之外产生了一些新的编辑现象(图 1F-H)。由于T-DNA插入位点与靶标突变位点的不连锁,在后代中会发生T-DNA序列与靶标位点分离,我们成功获得了两株无转基因成分(T-DNA插入片段)的无腺体T1株系(pgf-1和pgf-2),这也是基因编辑技术相对于传统转基因策略的重要优势之一。
Mb2Cas12a在棉花基因编辑中表现出广泛的温度适应性
为了研究Mb2Cas12a在不同温度条件下的编辑活性,我们选择了12株编辑GhPGF-crRNA1位点的T0植株分别在22℃、25℃、28℃ 和32℃条件下处理7天(图 2A)。我们发现Mb2Cas12a活性受温度变化的影响较小,在22℃条件下检测到相当高的编辑活性。例如,M71(由Strategy 1再生的植株)在22℃、25℃、28℃和32℃下的编辑效率分别为90.26%、94.62%、91.18%和92.29%,表明棉花在四种温度下没有显著差异(图 2B)。对这12株编辑植株的突变模式分析表明,Mb2Cas12的缺失位置和大小不受温度变化的影响(图 2C)。我们还对针对其他三个位点的T1株系进行了相同的温度处理,也没有观察到显著差异(图 S9)。
先前的研究表明,Cas12家族的核酸酶对温度敏感。不同植物物种的编辑效率也存在差异,主要原因可能是大多数植物组织培养过程所需的温度较低(25℃至30℃),降低了Cas12核酸酶的活性。然而,我们的研究结果表明,Mb2Cas12a对不同温度的适应性极强,即使在较低温度条件下也能表现出显著的编辑能力。因此,Mb2Cas12a有希望成为一种友好的基因组编辑技术,适用于一些偏向于低温生长条件的植物物种。
Mb2Cas12a扩展了棉花基因组编辑的范围
为了评估Mb2Cas12a的靶向范围,我们在棉花中进行了全面的全基因组PAM分析。结果显示,SpCas9识别的NGG PAM位点仅覆盖了Jin668(棉花遗传转化中最广泛使用的基因型)参考基因组的21.52%。相比之下,LbCas12a识别的TTTV PAM位点覆盖了全基因组的23.21%,而具有靶向TTV PAM位点特性的Mb2Cas12a可以覆盖59.59%的棉花基因组,与SpCas9相比增加了2.8倍,与LbCas12a相比增加了2.6倍(图 2D)。在此基础上,我们采用Strategy 1构建了靶向GhPGF的表达载体,在ATTV PAM位点实现了~15.50%的编辑效率,产生4-bp的片段缺失(图 2E)。值得注意的是,在以前的报道中,ATTV PAM位点不能被野生型LbCas12a或AsCas12a核酸酶所编辑。我们的结果表明Mb2Cas12a能够有效地编辑更为宽松的ATTV PAM,从而进一步提高其在植物基因编辑中的适用范围。
CRISPR/Mb2Cas12a介导的棉花多基因编辑
为了评估Mb2Cas12a在棉花植株中的多重基因组编辑活性,我们设计了8个crRNAs靶向5个内源基因(GhPGF、GhCLA1、GhFAD、GhTAC1、GhMYB25-like)(图 2F)。在测试的79个样品中,8个目标基因位点的突变频率从1.45%到86.96%不等。与之前描述的GhPGF-crRNA1目标位点的单基因编辑相比,在多基因编辑系统中,crRNA1目标位点的编辑效率为39.7%。此外,我们还发现中间crRNA单元(loci 3-4)的突变效率相对较低,从1.82%到17.93%不等。相比之下,crRNA单元两端的突变效率较高,crRNA1、crRNA7和crRNA8的突变效率分别为39.7%、71.47%和58.62%(图 2G)。
这些结果表明,在使用Mb2Cas12a的多基因编辑系统中,靶标的编辑效率可能受其crRNA特异性位置的影响。这一观察结果可能归因于Pol III启动子在转录tRNA和其他小RNA时具有适用性,而对长转录本可能效率不高。考虑到本研究中是从愈伤组织/再生植株中获得数据,建议对更多的样本进行进一步测试,以便进行更全面的分析。
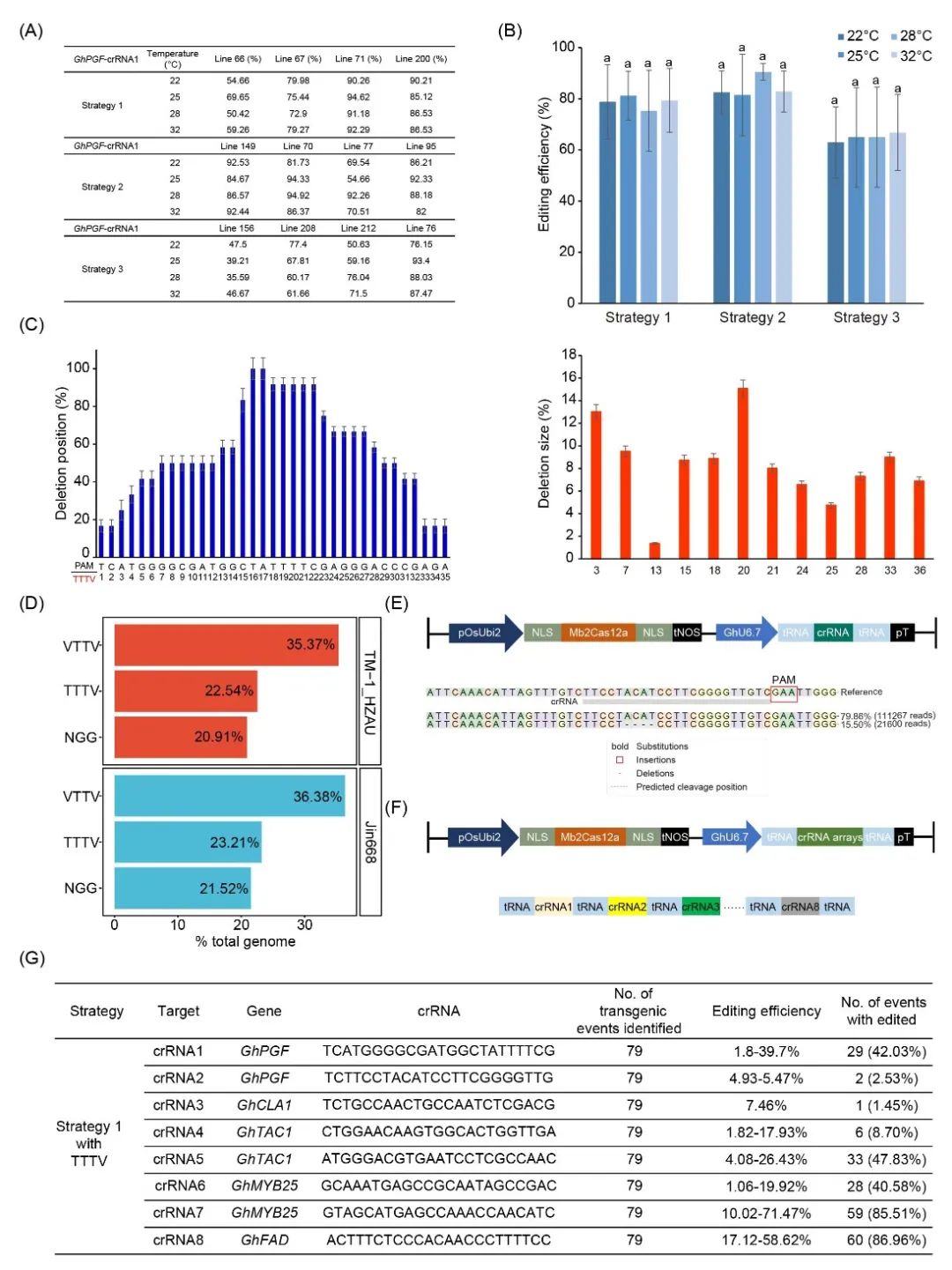
图2. 棉花中Mb2Cas12a广泛温度适应性和多基因编辑
(A)三种策略中筛选出12株编辑效率不同的T0植株进行22℃至32℃温度处理。(B)不同温度下编辑植株的统计分析。下一代测序(NGS)数据表明,不同温度处理下的编辑效率没有显著差异。Bar代表至少三个生物重复。数据以平均值±SD表示。统计差异采用t检验。(C)22℃条件下Mb2Cas12a的突变模式分析。(D)Mb2Cas12a在棉花(包括TM-1_HZAU和Jin668)基因组中可靶向PAM位点的全基因组分析。数字代表Mb2Cas12a识别的PAM序列在全基因组中的比例。(E)Mb2Cas12a在TTV PAM位点的有效编辑能力。含有TTV PAM的CRISPR/Mb2Cas12a双元载体的T-DNA区域示意图。图片表示高通量测序结果。红色框中突出显示了PAM序列。(F)多重Mb2Cas12a编辑系统示意图。表达盒包括Mb2Cas12蛋白和八个针对五个不同基因的crRNAs。关键元件包括pOsUbi2:水稻泛素
启动子;NLS:核定位信号;pGhU6.7:棉花U6.7启动子;pT:poly T序列。(G)Mb2Cas12a在八个TTTV PAM位点的编辑效率总结。
综上,这些数据表明,Mb2Cas12a是一种高效、精确的棉花基因组编辑工具,它极大地扩展了棉花基因编辑的范围,具有广泛的温度适应性。这些发现凸显了Mb2Cas12a在促进棉花遗传改良和研究方面是一种有价值的工具。
讨 论
总之,我们成功开发了棉花CRISPR/Mb2Cas12基因组编辑系统。该系统大大提高了棉花基因编辑的能力,扩大了全基因组可编辑的范围。同时,我们建立了基于CRISPR/Mb2Cas12a的高效多基因编辑系统,为创建个性化的棉花种质资源提供了宝贵的技术工具,极大地促进了该领域的基础生物学研究。这些进展为推进棉花育种和遗传研究带来了希望。
引文格式:
Hui, Fengjiao, Xu Tang, Bo Li, Muna Alariqi, Zhongping Xu, Qingying Meng, Yongxue Hu, Guanying Wang, Yong Zhang, Xianlong Zhang, and Shuangxia Jin. 2024. “Robust CRISPR/Mb2Cas12a genome editing tools in cotton plants.” iMeta 3: e209. https://doi.org/10.1002/imt2.209
作者简介

惠凤娇(第一作者)
● 华中农业大学作物遗传育种专业2021级博士生。
● 目前研究方向为棉花基因组编辑工具开发,主要聚焦于CRISPR/Cas12家族系列编辑器的研究与应用。

金双侠(通讯作者)
● 华中农业大学二级教授、国家杰出青年基金获得者、教育部青年长江学者、新疆天池英才。
● 目前主要从事棉花生物技术、基因编辑;棉花与害虫互作分子机制等研究工作。近年来开发了CRISPR/Cas9,Cas12a,Cas12b敲除系统,Cas13a/b/d/RX敲低系统,CBE/ABE碱基编辑器,dCas9-TV转录激活,dCas9介导的表观修饰工具,基因敲入等10余套基因编辑工具,在国际主流学术期刊Nature Communications、Genome Biology、Advanced Science等杂志上发表70多篇植物生物技术相关论文,其中12篇论文入选Web of Science高被引/热点论文,论文引用7000多次。目前担任Plant Biotechnology Journal杂志(影响因:13.8)执行主编、Genome Biology(影响因子12.2)编辑、Crop Journal(影响因子:6.6)副主编;2017-2021年担任棉花基础研究领域权威学术机构-国际棉花基因组协会(ICGI)共同主席。2021年获得棉花基础研究领域权威奖项-Cotton Biotechnology Award。

张献龙(通讯作者)
● 中国工程院院士,首批万人计划领军人才,首批全国创新争先奖,全国先进工作者,新中国培养的第一个棉花领域农学博士,教授,博士生导师,国家教学名师,中国作物学会棉花专业委员会会长、国际棉花基因组委员会联合主席/主席(2021-2023/2023-2025)。
● 在Nat Genet、Nat Plants等学术期刊发表研究论文300余篇。培育出华杂棉H318等25个棉花新品种,其中国审品种6个。一直是爱思唯尔“中国高被引学者":2020年以来连续入选科睿唯安“全球高被引科学家”。

张勇(通讯作者)
● 博士,西南大学生命科学学院/电子科技大学生命科学与技术学院,教授,博士生导师。
● 专注于植物基因组编辑及合成生物学相关教学、科研工作开展了系列研究工作:1)在Nature Plants、Molecular Plant、Nature Communications等期刊发表论文130余篇,Google Scholar引用10600余次,H指数40,17篇论文入选ESI高被引论文;2)入选科睿唯安2022、2023年植物与动物科学“全球高被引科学家”,评为四川省学术和技术带头人、四川省有突出贡献专家;3)当选中国遗传学会理事、中国生物工程学会理事、中国遗传学会基因组编辑分会第一、二届委员及多个省部级重点实验室学术委员会委员。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、Scopus等数据库收录。
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science







































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








