点击蓝字 关注我们
基于深度学习的CRISPR/Cas12a检测体系crRNA智能设计

iMeta主页:http://www.imeta.science
方法论文
● 期刊:iMeta [IF 23.7]
● 原文链接DOI: https://doi.org/10.1002/imt2.214
● 2024年6月15日,中国农业科学院深圳农业基因组研究所王鑫杰、军事科学院军事医学研究院生物信息中心王升启团队在iMeta在线联合发表了题为 “Deep learning enhancing guide RNA design for CRISPR/Cas12a-based diagnostics” 的方法文章。
● 本研究提出了一种用于Cas12a介导分子诊断的深度学习增强型crRNA设计系统,称为EasyDesign。这为基于Cas12a的检测中的crRNA设计提供了一种快速可靠的解决方案,可作为临床诊断和研究应用的宝贵工具。
● 第一作者:黄柏成、郭玲
● 通讯作者:王鑫杰(wangxinjie@caas.cn)、王升启(sqwang@bmi.ac.cn)
● 合作作者:尹航、吴越、曾梓菡、徐素杰、娄域峰、艾志敏、张卫强、阚兴池、余乾、杜世民、李超、吴丽娜、黄行许
● 主要单位:之江实验室、浙江大学医学院附属第一医院、剑桥大学、南京师范大学、军事科学院军事医学研究院生物信息中心、中国农业科学院深圳农业基因组研究所
亮 点
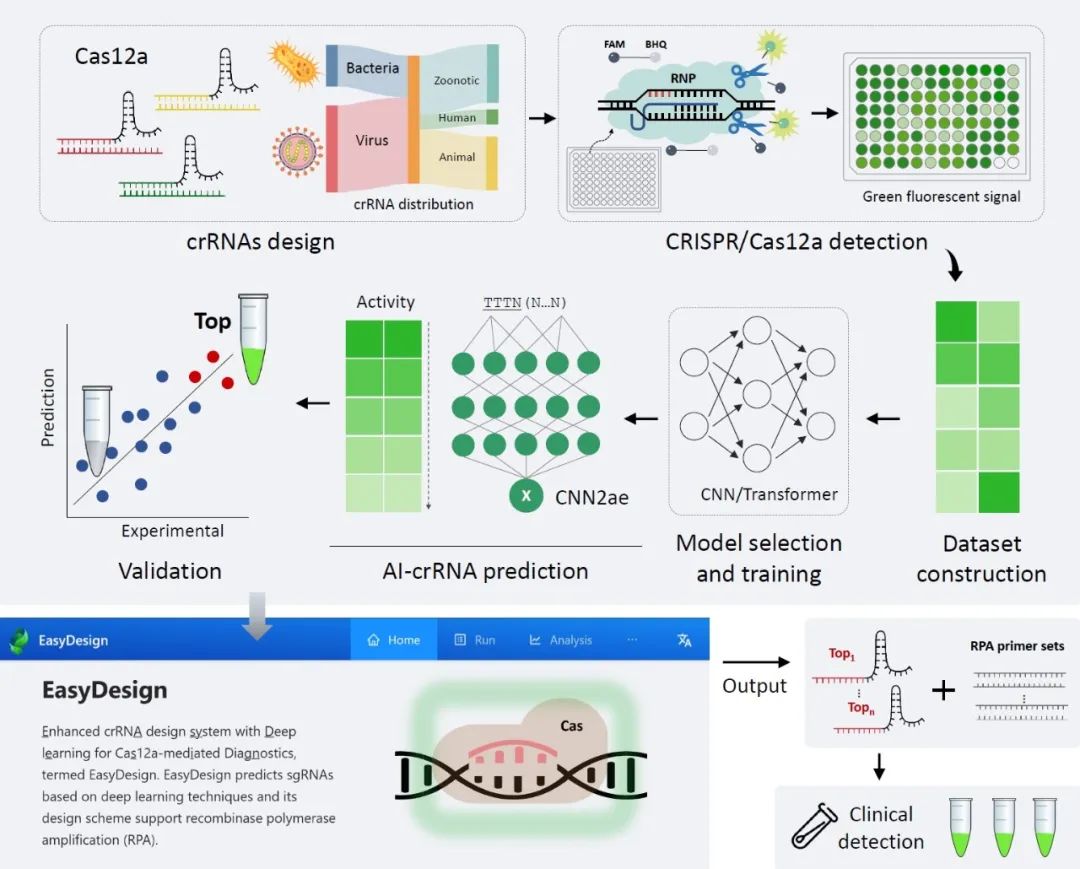
● 先进的 crRNA 设计系统:开发了EasyDesign,利用经过超过11,000个诊断目标对训练的卷积神经网络(CNN),能够为基于Cas12a的分子诊断创建高灵敏度的crRNA;
● 经过验证的预测能力:EasyDesign表现出对crRNA介导的Cas12a检测的卓越预测性能,并通过其在临床环境中针对多种病毒检测设计中的成功应用得到验证;
● 用户友好的Web平台:具有直观的Web平台,简化了基于CRISPR/Cas12a诊断工具的创建,从而提高了研究人员和临床医生的可访问性和可用性。
摘 要
快速、准确的诊断有助于改善患者治疗结果和对抗传染病。基于CRISPR/Cas12a的检测系统已成为一种有前景的现场核酸检测解决方案。但基于Cas12a检测的有效CRISPR RNA(crRNA)设计仍然具有挑战性且耗时。在此,本研究提出了一种用于Cas12a介导分子诊断的深度学习增强型crRNA设计系统,称为EasyDesign。该系统采用优化的卷积神经网络(CNN)预测模型,在包含11,496个经实验验证的涵盖了广泛的流行病原体的Cas12a检测数据集上进行训练,并进一步评估了训练数据中未包含的四种病原体(猴痘病毒、肠道病毒71、柯萨奇病毒A16和单核细胞增生李斯特菌)的crRNA设计模型性能。结果表明,EasyDesign较传统实验筛选表现更优。此外,还开发了一个交互式网络平台(https://crispr.zhejianglab.com/),它将EasyDesign与重组酶聚合酶扩增(RPA)引物设计相结合,提高了用户的可访问性。基于该平台,成功地为六种人乳头瘤病毒(HPV)亚型设计了最佳的Cas12a crRNA,针对每种HPV亚型所预测的活性排名前五名的crRNA在CRISPR检测中均具有强烈的荧光检测信号,表明该平台可以有效促进临床样本检测。总之,EasyDesign为基于Cas12a的检测中的crRNA设计提供了一种快速可靠的解决方案,可作为临床诊断和研究应用的宝贵工具。
视频解读
Bilibili:https://www.bilibili.com/video/BV1FT421Y72X/
Youtube:https://youtu.be/pnKkjtNOn1Y
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
快速准确的传染病诊断检测对于保障公众健康和及时为患者提供护理起着至关重要的作用。近期爆发的新冠肺炎和猴痘等疫情凸显了对有效诊断工具的迫切需求。CRISPR相关蛋白(Cas)中的Cas12或Cas13酶可识别特定核酸序列,随后可激活下游的非特异性旁切活性。基于CRISPR/Cas的检测具有快速、特异性、敏感性和易用性等优点,已成为传染病即时检测(POCT)的一种有前景的方法。
有效的crRNA的设计对于基于CRISPR/Cas的诊断系统至关重要。但目前的设计需要确定原型间隔区相邻基序(PAM)序列,并进行特异性和敏感性测试,耗时长、成本高,且容易受到个人经验的影响,因此迫切需要一种快速有效的方法来设计具有高特异性和敏感性的crRNA。这计算方法的发展有望简化流程,节省时间和资源,同时提高准确性。
深度学习已成为基于CRISPR的基因组编辑系统设计crRNA的强大工具。在经过实验验证的crRNA大型数据集上训练后,CNN和RNN在预测crRNA活性和特异性方面表现出色。此外,最近的研究表明,深度神经网络在设计Cas13诊断系统和Cas12编辑系统方面优于其他模型。值得注意的是,深度学习方法已成功预测了向导RNA的活动,例如 AsCpf1和CPf1。然而,仍然缺乏经过验证的用于Cas12a诊断的模型。此外,将深度学习模型转化为实际诊断实践仍具挑战,难以促进其在临床和研究应用中的广泛应用,因为所有现有模型都是基于合成文库开发的,不能反映真实世界病原体的基因组。
本研究开发的EasyDesign是基于CNN的Cas12a crRNA特异性设计系统,该模型在源自涵盖常见细菌和病毒病原体的高质量Cas12a检测序列的数据集上进行训练。EasyDesign预测生成的crRNA在检测MPXV、EV71、CV-A16、L. monocytogenes和HPV方面表现出色。此外,EasyDesign还提供了一个用户友好的Web服务器(https://crispr.zhejianlab.com/),其整合了重组酶聚合酶扩增(RPA)引物和crRNA设计。
结 果
建模的数据特征和模型评估方法
本研究包括数据采集、模型训练和验证阶段(图1A)。
1)为了开发深度学习模型的训练数据集,本研究准备了高质量的Cas12a crRNA序列以及相应的荧光检测体系。设计针对多种致病核酸序列的crRNA,涵盖了世界卫生组织(WHO)和世界动物卫生组织(WOAH)列出的主要病原体,并特别考虑到了动物源病原体对公共卫生造成的威胁。然后,应用之前建立的荧光CRISPR检测方法,使用酶标仪进行荧光检测,所得荧光读数与DNA和crRNA序列一起构成训练数据集。
2)训练和评估深度学习模型,包括CNN、Transformer及其增强版本。挑选出在预测crRNA活性方面表现出最高斯皮尔曼相关系数(ρ)的模型,以便随后在新的核酸数据上进行模型验证。模型预测与真实实验结果之间的一致性证明了其可靠性。
用于深度学习模型训练的Cas12a检测数据集
针对34种天然细菌和病毒病原体(表S1)设计了1533个crRNA,涵盖了WHO和WOAH确定的常见病原体。在原始病原体DNA模板中引入了随机突变代表固有的进化突变,产生了198个DNA模板(表S2)。随后,采用了之前建立的Cas12a荧光检测方法,在两小时的实验中每间隔两分钟收集荧光读数,以评估crRNA文库的反应性(图1B)。选取30分钟时获得的11496个荧光读数作为训练数据集(病毒来源7783,细菌来源3713),其中来自人畜共患病原体、人类病原体和动物病原体的数据数量分别为5275、1380和4841(图1C)。为了确保生成的引导与目标对的质量,进一步分析了这些目标对中的错配,并识别出3063对无错配、7939对单碱基错配和494对双碱基错配数据,所有数据的平均错配率为0.766,表明野生型和突变体对的合理组成(图1D),并观察到碱基类型呈均匀分布(图1E-H)。在所有包含错配的引导目标对中,碱基突变类型没有显着差异(图1I-M)。对于所有crRNA,PAM下游21个位置的碱基类型分布保持相对一致(图1N和S1)。
在数据集中,高错配会导致反应活动降低(图S2)。从机制上讲,crRNA 5'端Poly(T)的存在似乎与反应性的降低有关。正如预期的那样,当PAM(TTTN)中的“N”为“T”时,活性大幅下降,证明与TA、TC和TG相比,crRNA具有TT基序时活性较低(图S3A)。此外,无错配比有错配的数据具有更高的活性(图S3B)。除了特定位置碱基删除的显着影响外,PAM(TTTN)中“N”的突变对活性无显着影响(图S3C)。这些结果进一步支持训练数据的通用性。经过这些质量检查,引导目标对和相应的荧光数据构成了用于训练深度学习模型的数据集,包括训练数据(表S3)、增强数据(表S4)和测试数据(表S5)。

图1. 用于深度学习模型训练的基于Cas12a的crRNA数据集的准备和评估
(A)开发基于深度学习的CRISPR RNA (crRNA)设计模型的过程,包括高通量数据收集、机器学习和验证;(B)使用基于CRISPR荧光的测定进行数据采集的过程;(C)数据集中不匹配数据的分布,包括0、1和2个不匹配碱基的情况;(D)训练数据集中病原体分布的特征,包括病毒和细菌;(E-H)PAM及其相邻延伸位置的“TTTN1N2”区域内“N1N2”的碱基类型的分布;(I-M)数据突变类型,包括AN、TN、CN、GN和缺失-N;(N)crRNA数据集中不同位置的GC含量分布。
开发高效crRNA设计的深度学习模型
训练深度学习模型包括数据处理、模型选择、训练和验证四个步骤(图2A)。首先,使用One-hot(2D)编码作为基础编码。然后,测试了CNN衍生的CNND和Transformer衍生的TransformerD模型,并进一步训练了适应Cas12a诊断场景两个模型,即CNN12a和Transformer12a(图S4)。最后,使用最优模型预测crRNA的活性。
在crRNA候选设计过程中,对PAM靶序列(TTTN)进行了迭代搜索,提取PAM位点下游的21个核苷酸序列。为了考虑周围序列的潜在影响,在靶标的5'和3'末端将crRNA额外延伸了10个核苷酸,从而形成了45个核苷酸的靶序列(图2B)。11496个荧光读数的数据集分为10634个数据对的训练集和862个数据对的测试集。使用测试集评估四个经过训练的模型。CNN12a和Transformer12a模型针对Cas12a特征进行了调整,具有更高的Spearman相关系数(ρ值分别为0.656和0.541),其性能优于CNND和TransformerD模型(ρ值分别为0.620和0.532)(图2C)。
在训练过程中,通过数据增强进一步提高模型性能。具体来说,使用并标准化了同一测定中的20分钟和30分钟读数来创建一个增强数据集,其中包含31993个引导目标对。然后使用该增强数据集训练CNN12a和Transformer12a模型,分别生成CNN12ae和Transformer12ae模型。使用测试集进行评估后,发现CNN12ae性能优于CNN12a模型(ρ = 0.673),并且也优于Transformer12ae(ρ = 0.467)(图 2C)。值得注意的是,CNN12ae在一个更大的测试集上提升了性能(ρ = 0.812),包括原始测试数据和另外30%的高度不匹配的数据对。
与其他模型相比,CNN12ae具有不同活动水平的不同区域(图2D和S5),在低值区域表现出了卓越的性能(图2E和S5)。此外,CNN12ae模型的分类性能很强,其AUC值为0.8247,p < 0.0001(图2F)。研究发现CNN12ae的预测性能对于病毒(0.66)和细菌(0.69)大致一致(图S6),支持该模型的泛化特性。此外,CNN12ae使用5倍交叉验证方法对33351个数据点的数据集进行评估,确保训练集和测试集之间目标RNA序列的严格分离,以防止数据泄漏。每个包含26952个训练数据和6399个测试数据,共含450个epoch的训练。在数据中,ρ值分别为0.8038、0.8267、0.7962、0.7939和0.8481,Pearson相关系数分别为0.7103、0.7399、0.7202、0.6649和0.7878,表明该模型具有强大的通用性,在各自领域具有准确预测的潜力(表S6)。因此,本研究选择CNN12ae模型进行进一步的Cas12a介导的crRNA设计。

图2. 开发和评估适用于Cas12a诊断设计的深度学习模型
(A)概述了选择和训练深度学习模型的过程的四步流程图;(B)使用Spearman相关系数和Pearson相关系数的模型的性能比较;(C)CNN12A模型对所有枚举的crRNA-靶标DNA序列对使用One-hot编码,并使用“TTTN”PAM;(D)CNN12ae的真实值(x轴)和预测值(y轴)的核密度估计;(E)预测值四分位数的真实活性密度图,x轴是标准化的真实活性,y轴是预测的四分位距,它表明当CNN12a预测值低于第二个四分位数时,真实活性的分布更集中;(F)CNN12ae的保留测试集的接收者操作特性(ROC)曲线,区分非活性或活性(真实活性 = 3)的对,AUC(曲线下面积)为0.8247(p < 0.0001)。
EasyDesign助力精准病原检测
本研究通过crRNA活性实验来验证所开发模型的适用性。EasyDesign预测了来自4个致病性病原核酸序列共194个crRNA的活性排名(图3A和表S7),(表S8),包括MPXV、EV71、CV-A16和L. monocytogenes。在crRNA候选集中,深度学习模型预测的crRNA与实验验证的真实crRNA之间存在一致性,对于实验验证的活性前10名crRNA,每种病原体预测的crRNA分别有7、4、4 和3个相符;而对于活性前20名crRNA,每种病原体分别有16、15、13和11个相符(图3B),表明该模型可以从预测候选物中有效地识别出用于实验测定的高反应性crRNA,从而显著降低筛选强度。此外,实验结果表明,深度学习模型生成的预测crRNA表现出与已报道qPCR位点附近的crRNA具有相同或更高的反应性,特别是在109拷贝/反应的DNA浓度下(图3C-F和S7)。值得注意的是,当使用较低的DNA模板浓度(108拷贝/反应)时,crRNA活性排名的变化总体上与预期一致。这些发现表明EasyDesign可以预测具有高活性的crRNA,可促进有效crRNA的筛选。

图3. 验证EasyDesign对四种病原体的性能
(A)病原体模板的比较筛选测试对EasyDesign进行实验验证的流程图;(B)实验检测到的顶级crRNA数量与EasyDesign预测的顶级 crRNA 数量之间的比较;(C-F)实验CRISPR荧光与EasyDesign预测中crRNA活性的比较分析,包括猴痘病毒(MPXV)、肠道病毒71 (EV71)、柯萨奇病毒A16 (CV-A16)和单核细胞增生李斯特菌 (L. monocytogenes)。
用于端到端Cas12a诊断设计的用户友好型Web工具
为提高开发基于Cas12a的诊断方法的效率,本研究创建了EasyDesign在线平台:https://crispr.zhejianlab.com/。该平台提供了全面的基于Cas12a的诊断设计方案,集成了RPA引物设计,促进了RPA-CRISPR联合检测的设计(图4A)。Web平台用户界面友好,可指导用户完成工作流程的每个步骤,包括序列输入、参数选择和crRNA设计输出。EasyDesign提供了带注释的设计可视化,阐明了crRNA和引物与目标序列的相互作用。
EasyDesign促进HPV临床样本诊断
为了评估EasyDesign的有效性,本研究使用其设计了针对六种HPV亚型(HPV6、HPV11、HPV16、HPV18、HPV31和HPV33)临床样本的RPA-CRISPR检测方法。合成了六种HPV亚型最佳的crRNA和推荐的RPA引物(表S9)(图4B)。使用在线服务器提供的候选crRNA和RPA引物的组合,在检测合成的HPV DNA模板时可观察到明显的荧光检测信号(图4C-H)。当检测不同亚型临床样本时,观察到所有五种候选crRNA都有明显荧光信号(图4I-N和S8),表明EasyDesign的有效性。

图4. 通过EasyDesign网络服务器进行HPV临床样本检测设计
(A)基于Web的EasyDesign平台,包括基于Cas12a的检测序列输入、crRNA和扩增引物设计;(B)基于Web的设计界面的展示,包括序列上传、参数选择以及候选crRNA和扩增引物对的生成;(C-H)使用合成DNA模板的代表六种人乳头瘤病毒(HPV)亚型(HPV6、HPV11、HPV16、HPV18、HPV31和HPV33)的荧光检测动力学曲线。利用基于Web的EasyDesign工具生成最佳扩增引物和crRNA。为每个模板生成了五个最佳crRNA及其相应的引物对。荧光图显示了30分钟孵育期后获得的结果。(I-N)获得了代表六种HPV亚型的临床样本在30分钟孵育期后的荧光检测结果。阳性临床样本鉴定如下:#S1-#S3为HPV6,#S4-#S6为HPV11,#S7-#S10为HPV16,#S11-#S12为HPV18,#S13-#S14为HPV31,#S15为HPV33。NC,阴性对照。
方 法
DNA模板和crRNA的设计
针对已报道的不同病原体核酸检测靶标序列(表S1),全局设计crRNA,以最大限度地生成实验室检测数据。生成crRNA集后,对DNA模板进行区分,为每个带有突变、插入和缺失的原始DNA模板生成9个模板集。所有crRNA均根据LbCas12a crRNA的原理设计并合成。
制备Cas12a检测数据
为了进行活性定量,基于Cas12a的反应混合物包含200 ng纯化的LbCas12a、25 pM ssDNA FQ探针、1 µM crRNA和2µL反应缓冲液中的样品。反应体积20 μL,反应温度37℃。使用Varioskan® LUX酶标仪(Thermo Scientific,美国)收集CRISPR反应的荧光信号。样品中包含阴性和阳性对照,使用这些对照对荧光值进行标准化。酶标仪配置为在实验期间每2分钟记录一次荧光值。为了满足快速检测的要求,选择第30分钟的荧光值作为活性评估的指标。
预测检测活性:数据处理
实验中,每个96 孔板中设置两个反应孔(无DNA模板)作为阴性对照,并设置两个具有高荧光活性的重复测定孔作为阳性对照。为了消除背景噪音的影响,从板读数器读数中减去同一板中阴性对照的平均值,然后除以阳性对照读数的平均值。此标准化过程应用于本研究的模型训练数据。
数据增强
为了最大限度地减少实验检测中的荧光值波动并标准化数据,本研究采用了时间折叠技术,将标签值不确定性引入深度学习模型。对于每个目标DNA和crRNA对,使用两个标记值:1)30分钟记录的荧光值,2)基于20分钟记录数据的30分钟标准化荧光值。假设将t2 min时的读数归一化至t1 min,则归一化方法为:g(t1,t2) = [[f(t1+2)-f(t1-2)]^[(t1-t2)/4]]*f(t2)。这里,f(x)是x分钟时的荧光读数,g(t1, t2)是以t1时间为标准的t2时刻的荧光读数值。
One-hot(2D)编码
碱基编码的One-hot(2D)编码中,在插入或删除的情况下,目标DNA或crRNA序列中使用“-”占位符,从而产生五种可能的情况:“A”、“C”、“T”、“G”和“-”。目标DNA和crRNA的对应位置一起形成一个10维One-hot向量。因此,输入维数为(45, 10),由目标和引导序列的连接One-hot编码组成。CNN模型使用One-hot向量的输出,而在Transformer模型中,向量随后通过1D卷积(Conv)层进行处理,然后是经典的Transformer编码器和解码器层。
模型选择
关于引导RNA性能预测,已有研究表明CNN优于LR、GBT、RF、SVM、LSTM和传统MLP。因此,本研究主要比较了CNN模型与Transformer模型在解决crRNA计算问题方面的性能。
CNN模型:CNND、CNN12a和CNN12ae
CNND是源自Adapt框架的卷积神经网络模型。它已在Cas12a数据集上进行了训练,该数据集是代表各种模式的多样化样本集合。网络架构由一个卷积块、一个扩展层(Flatten)和两个密集层(Dense)组成。卷积块由一个卷积层、一个池化层、一个批量归一化层(BN层)和一个局部连接层组成。回归模型中的卷积层宽度为{1, 2},具有25个卷积核,池化层宽度为2,并使用平均池化。全连接层维度为53,局部连接层宽度为{1, 2},维度为3。使用的激活函数为ReLU,dropout概率设置为0.30,批量大小为229,未应用批量归一化。此外,不执行回归参数修剪。
CNN12a是CNND的扩展,包含两个关键修改。首先,编码机制经过调整,可以更好地捕捉Cas12a诊断场景的基本要求。其次,对用于计算预测值与实验室结果之间差异的损失函数进行了修订。这种更新后的损失函数对数据中的差异表现出更高的敏感性,在训练期间为模型提供更好的指导,从而提高诊断功效的预测。CNN12a在与CNND相同的数据集上进行训练,以确保模型之间的公平比较。
CNN12ae是CNN12a的增强版本,专注于数据增强以解决数据不平衡问题。使用数据增强技术,用部分匹配的样本增强数据集。通过在现有数据中随机引入>4个不匹配,合成引入标记为“非活性”的高度不匹配的crRNA/目标对。此增强过程还包括在20分钟时间点进行数据增强。这可获得了更平衡的数据集,随后用于训练CNN12ae,即CNN12a的改进迭代。
Transformer 模型:TransformerD、Transformer12a和Transformer12ae
TransformerD最初是基于之前的研究构建的,该研究已成功利用transformer架构预测Cas13a靶标。然后,使用数据集对TransformerD进行微调,并引入必要的修改来创建Transformer12a。这些修改包括调整编码方法以解决不匹配的样本。此外还探索了在编码层之前合并卷积层,以从输入序列中提取复杂的模式和特征。更进一步,Transformer12a经过训练以利用增强数据集,从而产生了Transformer12ae。
所开发模型的实验验证
选择四种病原体的检测用DNA序列作为输入,对所开发的模型进行实验验证,包括MPXV、EV71、CV-A16和L. monocytogenes。crRNA(表S4)展示了四种病原体的目标DNA序列,然后合成用于CRISPR检测,并将荧光结果与模型预测的crRNA活性进行比较,并用四种病原体的qPCR检测位点注释了结果,以比较crRNA活性。
RPA反应
在反应管中添加20 μL裂解试剂、3 μL正向引物(10 μM)、3 μL反向引物(10 μM)、5 μL模板DNA(根据应用而变化)和17 μL ddH2O,总计体积48 μL,充分摇匀,立即离心。对于每个样品,向管中添加2 μL活化剂(醋酸镁),小心盖上盖子,摇匀,并立即离心。将反应管放入恒温器中,39℃孵育25分钟。获得的RPA产物用于下游应用。
临床样本的CRISPR检测
本研究中使用的阳性HPV样本(n = 15)在临床实验室已通过qPCR方法得到了确认。宫颈细胞沉淀离心后用裂解液重悬,进行核酸制备,95℃加热10分钟释放HPV DNA,-20℃保存备用。对于CRISPR反应,混合以下成分制备了20 µL CRISPR反应体系:8.8 μL ddH2O,2 μL反应缓冲液,0.2 μL RNAase抑制剂,1.0 μL ssDNA FQ reporter,2 μL亚型特异性crRNA,1.0 μL LbCpf1蛋白。然后,将5 μL DNA模板加入CRISPR反应中。将96孔板装入Varioskan™ LUX酶标仪,在37℃下每隔2分钟采集一次荧光值,持续2小时。同时,30分钟后拍摄管内反应的荧光信号。
讨 论
引物和crRNA的繁琐设计限制了CRISPR/Cas12a在检测领域更广泛的应用。设计过程要求研究人员确保他们选择的靶标在所选引物和crRNA靶标序列中都具有序列保守性。这两个问题会导致切割效率降低和检测灵敏度降低。在本研究中,开发了EasyDesign,可加速CRISPR/Cas12a检测靶标的设计。
面向精度的深度学习模型需要建立在严格准确的检测数据之上。为了应对这一不断变化的挑战,本研究首先精心设计了35种病原体的DNA模板,实现了crRNA的全局设计。数据包括多种病毒和细菌,包括人畜共患病原体、人类目标病毒和动物病毒,覆盖范围广,增加了数据的普遍性,可代表不同生物物种中核酸分布特征的真实情况。使用之前建立的CRISPR荧光测定法和批量检测来实现标准化,以寻求开发准确的深度学习模型。
在应对新出现的疫情时,快速准确的核酸检测方案设计和易于使用的检测方法对于防止病毒传播和提供有效的医疗至关重要,特别是在疾病流行或已经发生疫情的地区。研究中,观察到预测crRNA活性排名与病毒的crRNA活性排名之间存在很强的相关性,表明深度学习模型在crRNA设计中具有强实用性。此外,本研究表明EasyDesign(https://crispr.zhejianlab.com/)足以支持建立不同HPV亚型的常规临床快速检测系统。
然而,目前的模型训练数据基于LbCas12a蛋白,它识别PAM序列“TTTN”。这意味着LbCas12a变体以及Cas12蛋白家族其他成员的预测性能需要进一步研究和完善。未来的研究将旨在扩展这些发现,增强此检测方法更广泛的适用性。
代码和数据可用性
研究所涉及的程序代码和实验数据可通过一下方式得到:EasyDesign可在https://github.com/scRNA-Compt/EasyDesign获取;安装和复制模型的脚本可在https://github.com/scRNA-Compt/EasyDesign/blob/main/README.md获取;Cas12a设计的在线网站:https://crispr.zhejianlab.com/。
引文格式:
Baicheng Huang, Ling Guo, Hang Yin, Yue Wu, Zihan Zeng, Sujie Xu, Yufeng Lou, Zhimin Ai, Weiqiang Zhang, Xingchi Kan, Qian Yu, Shimin Du, Chao Li, Lina Wu, Xingxu Huang, Shengqi Wang, Xinjie Wang. 2024. Deep learning enhancing guide RNA design for CRISPR/Cas12a-based diagnostics. iMeta 3: e214. https://doi.org/10.1002/imt2.214
作者简介

黄柏成(第一作者)
● 西北农林科技大学博士,之江实验室高级研究专员。
● 主要从事结合AI智能计算的生物诊断与合成生物学研究。

郭玲(第一作者)
● 清华大学计算机系硕士,之江实验室工程专家。
● 主要方向为生物多组学AI计算。

王鑫杰(通讯作者)
● 中国农业科学院深圳农业基因组研究所研究员,博士生导师。
● 长期致力于新型生物传感器的开发及其机制研究,在Nature Communications、Science Bulletin、Advanced Science等期刊发表学术论文40余篇,他引1000余次,获授权国家发明专利10余项,主持及参与5项国家自然科学基金及省部级课题,荣获第七届中国科协优秀科技论文、《Science Bulletin》年度最佳论文等奖项。

王升启(通讯作者)
● 军事科学院军事医学研究院生物信息中心研究员。
● 主要从事分子诊断与治疗技术创新和产品研发。以第一完成人获国家技术发明二等奖1项,国家科学技术进步二等奖1项,军队和省部级一等奖4项。以第一或通讯作者在Nat Microbiol、Nat Mach Intell、Nat Commun、Gut、Nucleic Acids Res、Mol Cell等国际期刊发表SCI论文200余篇,以第一发明人获发明专利36项;获我国首个反义核酸1类新药临床研究批件,并已进入II期临床;负责研发的分子检测产品获国家和军队注册证60余个。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(2023 IF 23.7)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、PubMed、Scopus等数据库收录。
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science







































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








