点击蓝字 关注我们
破译瘤胃微生物的功能群及其在塑造宿主表型中的潜在因果关系

研究论文
● 期刊: iMeta (IF 23.7)
● 原文链接DOI: https://doi.org/10.1002/imt2.225
●2024年7月15日,浙江大学动物科学学院刘建新、孙会增和湘湖实验室薛茗元等在iMeta在线发表了题为“Deciphering functional groups of rumen microbiome and their underlying potentially causal relationships in shaping host traits”的文章。
● 本研究对于高饲料效率奶牛瘤胃中营养利用功能群的探索、以及关键微生物功能蛋白及其潜在因果关系的识别,有助于帮助瘤胃微生物组研究从相关性研究迈向因果关系探究,以实现对瘤胃微生物的精确调控从而提升动物的生产性能。
● 第一作者:薛茗元
● 通讯作者:刘建新(liujx@zju.edu.cn)、孙会增(huizeng@zju.edu.cn)
● 合作作者:谢云怡、臧新威、钟一帆、马晓娇
● 主要单位:浙江大学动物科学学院、湘湖实验室(农业浙江省实验室)、浙江大学动物分子营养学教育部重点实验室
亮 点
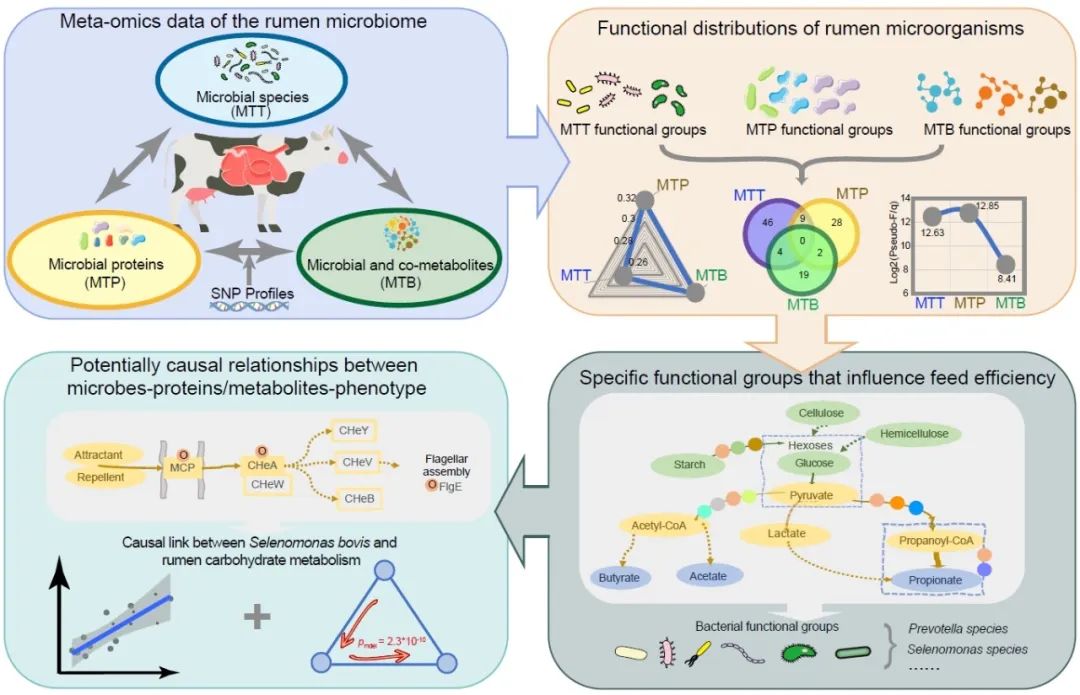
● 结合瘤胃多种宏组学数据,解析了影响饲料效率的瘤胃微生物的活性功能特征分布;
● 通过整合宿主基因组数据并将其作为中介,确立了微生物、蛋白质/代谢物和表型之间的潜在因果关系;
● 发现了牛月形单胞菌Selenomonas bovis与瘤胃碳水化合物代谢之间的因果联系,揭示了其在提高奶牛饲料利用效率方面的作用。
摘 要
瘤胃是反刍动物的关键消化器官,依赖微生物进行饲料消化,显著影响营养供应和宿主性状,尽管对瘤胃微生物进行了广泛研究,我们对微生物组在表型上的功能角色和因果效应的理解仍然有限。在本研究中,我们聚焦于瘤胃宏蛋白质组,结合宏转录组和代谢组数据,精确识别了影响饲料效率的瘤胃微生物的活性功能分布及特定功能群。通过整合宿主基因组数据,我们揭示了微生物-蛋白质/代谢物-表型之间的潜在因果关系,并破解了瘤胃微生物功能群对宿主饲料效率影响的作用模式和机制。我们发现牛月形单胞菌与瘤胃碳水化合物代谢之间存在潜在的因果关系,可能通过细菌趋化作用和两组分调控系统介导,从而影响奶牛的饲料利用效率。本研究对于高饲料效率奶牛瘤胃中营养利用功能群的探索、以及关键微生物功能蛋白及其潜在因果关系的识别,有助于帮助瘤胃微生物组研究从相关性研究迈向因果关系探究,以实现对瘤胃微生物的精确调控从而提升动物的生产性能。
视频解读
Bilibili:https://www.bilibili.com/video/BV1oi421h7Zo/
Youtube:https://youtu.be/jGvxrqE9cHU
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
瘤胃微生物在帮助反刍家畜将饲草转化为高质量的蛋白质产品的过程中起着关键作用,同时伴随大量甲烷生成,对环境产生不同程度的影响。对于奶牛来说,瘤胃微生物主要参与饲料基质的分解、利用和代谢效率,直接影响奶牛的乳前体物质和能量的生产效率,从而影响其乳品生产效率。由于瘤胃微生物复杂的分类学、广泛的功能冗余和严格的厌氧环境,目前对瘤胃微生物组的理解仍然有限。理解瘤胃中营养物质的消化和利用对于精确调控瘤胃微生物以提高奶牛饲料转化效率至关重要。
早期的瘤胃宏基因组研究主要集中于微生物的物种分类差异上,而解析瘤胃微生物在宏基因组和宏转录组水平上的功能角色一直是一项挑战,受限于潜在的DNA水平功能(包括无活性微生物)和转录水平功能(受mRNA富集和RNA不稳定性限制),从而阻碍了人们对其准确的功能描述。宏蛋白质组学的出现,帮助我们更精确地表征瘤胃微生物组的实际功能。近年来,随着对瘤胃微生物组及其与宿主相互作用研究的推动,微生物群落中高度的分类学多样性引发了关于微生物功能冗余性的讨论。功能冗余在分类学上不同的微生物之间普遍存在,它们编码类似的产能代谢功能,与每个物种占据独特代谢位的期望相矛盾。瘤胃微生物组的复杂群落结构尚未深入探索,基于功能冗余性背后的具体功能群体还有诸多空白亟待探索。
因此,本研究聚焦于瘤胃宏蛋白质组(MTP),利用宏转录组(MTT)数据作为数据库,准确表征瘤胃微生物的真实功能分布,并鉴定发挥不同角色的关键蛋白质。通过整合宏组学数据(MTT、MTP和代谢组[MTB])和宿主遗传数据,我们破解了与饲料效率密切相关的微生物功能群,并揭示了它们与表型之间的因果关系,为瘤胃微生物的功能特征化研究提供新的视角。
结 果
数据概览以及基于宏蛋白质组学的瘤胃微生物功能解析
本研究了采用来自9头高效饲料利用率和9头低效饲料利用率奶牛作为研究对象,采集瘤胃内容物样本,涵盖了MTT、MTP和MTB数据(图1)。总共鉴定出28,589个来自三种不同组学数据的特征、以及来自298头奶牛的20,571个SNP位点。MTT测序获得9,139个微生物分类群,MTB数据包含283个代谢物特征,MTP特征集包括19,167个蛋白质。宏蛋白组学分析产生了1,663,161个质谱,鉴定了85,693个肽段和19,167个蛋白质,经过严格的质量控制步骤确保结果准确性。高效率动物和低效率动物在MTT和MTP特征上有显著差异。

图1. 技术路线与特征概述
(A) 试验奶牛瘤胃转录组学(MTT)、宏基因组学(MTP)和代谢组学(MTB)的试验流程概览 (B) 高效与低效奶牛宏组学特征韦恩图。
在宏蛋白质组学水平上对瘤胃微生物组功能多样性进行分析显示,高效和低效组之间的Shannon指数没有显著差异(p > 0.05),但Chao1丰富度指数在高效组显著较高(p = 0.015)(图2A)。主成分分析(PCA)散点图显示高效和低效组之间存在分离趋势(图2B),基于Bray‒Curtis的β-多样性分析显示两组微生物群落结构存在显著差异(经过置换多元方差分析修正,置换次数 = 999,R2 = 0.32,p < 0.05)。我们在高效组中发现了247个上调和245个下调的差异表达蛋白质DEPs(adj-p < 0.05,图2C)。斯皮尔曼相关分析未显示出高效和低效动物瘤胃中差异表达基因DEGs与DEPs之间的显著相关性(p > 0.05,图2D)。

图 2. 瘤胃微生物群的主要宏蛋白质组特征
(A) 箱线图展示不同饲料效率奶牛瘤胃宏蛋白质组的α多样性和丰富度指数 (B) 主成分分析(PCA)图展示不同饲料效率奶牛瘤胃微生物宏蛋白质组的β多样性 (C) 火山图比较不同饲料效率奶牛瘤胃微生物的蛋白质表达 (D) 显著差异的转录特征与蛋白质组学特征的相关分析 (E) 显著上调的蛋白质同源物群(COGs)的功能分类。LFCR,低饲料转化率;HFCR,高饲料转化率。
瘤胃微生物组潜在功能与实际功能群的差异
通过对每个比较组内差异表达蛋白质(DEPs)进行聚类分析,根据倍数变化分成四个四分位(Q1至Q4),对每个四分位进行了GO分类、KEGG通路分析以及蛋白质域富集分析,并揭示了不同表达水平蛋白质的功能相关性。利用Q3和Q4进行基于mRNA和蛋白质的功能富集分析,这些四分位在高饲料效率个体中显著富集。通过追溯瘤胃微生物种属的分类信息,确定了与高效率饲养奶牛相关的瘤胃微生物功能群。桑基图展示了基于mRNA(图3A)和蛋白质(图3B)的潜在功能群及实际功能群的功能-微生物-分类映射。无论是基于潜在功能组还是实际功能组,桑基图的结果均提示了瘤胃微生物群落功能水平的高度冗余。
基于MTT的功能组包括来自17个家族的59种微生物物种,主要包括来自Prevotellaceae(25%)、Lachnospiraceae(18%)、Ruminococcaceae(7%)和Clostridiaceae(7%)的微生物(图3A)。Prevotella bryantii、Selenomonas bovis、Clostridium spp.和Firmicutes spp.在物种水平上具有最丰富的差异表达基因(DEG)功能。功能类别主要涵盖碳水化合物运输和代谢、转录、脂质运输和代谢以及辅酶运输和代谢。相比之下,基于MTP的功能组包括来自15个家族的39种微生物物种,主要包括来自Prevotellaceae(38%)、Selenomonadaceae(10%)、Lachnospiraceae(10%)和Succinibrionaceae(8%)的微生物(图3B)。S. bovis和P. bryantii在物种水平上是具有最丰富的差异表达蛋白质(DEP)功能的物种。
基于代谢物的功能组(MTB-based)通过代谢物起源分析和基于起源的代谢功能分析生成,MTB数据集分为四组,包括50种微生物代谢物、4种宿主特异性代谢物、114种微生物-宿主共代谢物和117种其他代谢物。经过起源基础功能分析,选择了29种功能进行下游的功能群分析,通过桑基网络可视化了细菌类群与代谢物之间的生物学关系。该结果揭示了10个KEGG通路与来自12个家族的25种微生物物种的生物学相关,其中S. ruminantium展示了比其他微生物更多的差异功能(图3C)。基于瘤胃微生物代谢物的功能群的桑基图映射结果也显示了显著的功能冗余性。

图3. 基于宏转录组、宏蛋白质组、宏代谢组的高效奶牛瘤胃微生物功能群
(A) 根据宏转录组数据分析的与高饲料效率相关的瘤胃微生物功能群分类和功能映射关系桑基图(B) 根据宏蛋白质组数据分析的分类和功能映射关系桑基图 (C) 基于代谢组学分析的分类和功能映射关系。
在此基础上进行了三个不同功能群水平的比较分析,并评估了它们在组成上的差异。我们观察到三个功能群之间的重叠很小,MTT和MTP功能组在9种物种上共享最多,包括P. bryantii、S. bovis以及来自Clostridium、Faecalibacterium和Firmicutes的三种未培养的细菌物种。为了确定MTT、MTP和MTB功能群在组成上的这些差异的显著性,我们进行了PERMANOVA分析,显示出高、低饲料效率群体之间在组成上存在显著变化。R2值(MTT为0.28,MTP为0.32,MTB为0.31)表明了功能群中由FCR因素解释的变化比例大小。我们还计算了效应大小统计量log2(F/q),用于量化FCR因素对三个功能群总变异的贡献。结果表明,饲料效率表型对MTP(12.85)的总变异影响最大,其次是MTT(12.63),而MTB的影响最小(8.41)。

图4. 基于不同组学数据的瘤胃功能群比较
(A)基于MTT、MTP和MTB的高饲料效率相关的瘤胃微生物功能群重叠的韦恩图 (B) 每个功能群的饲料效率辨别力比较,包括基于Bray‒Curtis的β多样性的PERMANOVA R2值(C)和log2(F/q)值(D)。
高饲料效率瘤胃功能群通过关键蛋白质发挥功能
我们发现基于MTP的功能性群体表现出微生物参与多种代谢途径的显著模式,特别是碳水化合物代谢、氨基酸代谢和细菌趋化性,如图5所示。为深入理解其机制,我们分析了在这些途径中涉及的关键蛋白质及其功能域。碳水化合物代谢涉及多种功能蛋白质和酶的复杂过程,包括糖酵解、戊糖磷酸途径、嘌呤代谢、三羧酸循环(TCA循环)和丙酸代谢,如图5A所示。我们鉴定了四种不同的微生物(Selenomonas sp.、Ruminobacter sp.、Succinivibrionaceae sp.和Prevotella sp.),它们可能通过α-淀粉酶以及参与淀粉和六碳糖降解的转录因子发挥功能。此外,微生物如Selenomonas sp.、Prevotella sp.和Butyrivibrio sp.通过糖酵解途径的关键酶(如磷酸甘油醛-3-磷酸脱氢酶)在将葡萄糖分解为丙酮酸过程中起到关键作用。碳水化合物代谢的后续途径则通过多种蛋白质功能群体促进,包括NapF、PorG、PorA和RecN的膜蛋白、PckA、SucD、PccA和Mdh的酶、TtdA、Sbm和MmdA的转运蛋白、以及UxaC和TktA的糖代谢蛋白及PurM和AdK的代谢调节蛋白。此外,细菌种类如S. bovis和P. bryantii(以及其他Prevotella sp.)通过其酶(如氨基酸氨基转移酶和脱羧酶)参与氨基酸代谢途径,详见图5B。

图5. 基于宏蛋白质组学的瘤胃微生物功能群中关键蛋白质在关键代谢途径中的作用图示
(A) 碳水化合物代谢通路图 (B) 氮代谢通路图。
S. bovis与功能性蛋白/代谢产物之间潜在的因果关系
值得注意的是,S. bovis在多条碳水化合物代谢途径中起到了重要作用(见图6A)。具体而言,S. bovis参与了淀粉降解(TreY)、糖酵解(GapA)、戊糖磷酸途径(SucD、RecN和Mdh)、乙酰辅酶A形成(PorG、PorA)以及丙酸形成(PccA、Sbm和MmdA)等关键过程,这些过程受其潜在的蛋白功能调控。此外,S. bovis通过关键蛋白如感知化学梯度的甲基受体趋化蛋白(MCP)、响应环境信号并将磷酸转移给响应调节蛋白的组氨酸激酶(CheA),以及鞭毛马达蛋白(FlgE)等参与了细菌趋化作用途径(图6B)。
在将S. bovis与动物高效饲料利用的MTP特征关联后,我们采用双向Mendelian随机化(MR)和中介分析来确定S. bovis是否因果性影响这些特征。在蛋白水平上,MR分析显示S. bovis丰度与MCP(r = 0.92, p < 0.05)或CheA(r = 0.72, p < 0.05)呈正相关(见图6C),结果显著(β来自逆方差加权测试,IVW,p < 0.05)。此外,中介分析显示,MCP和CheA可能介导了S. bovis对饲料利用效率参数的影响(pmdei < 0.05,见图6D)。
在代谢水平上,我们还将S. bovis与促进动物高效饲料利用的MTB特征联系起来,并进行了双向MR和中介分析以评估其因果作用。尽管未发现S. bovis与MTB特征之间的显著关联,但MR分析显示S. bovis与丙酮酸之间存在潜在正因果关系,IVW效应大小(β)= 0.15(p < 0.05,见图6C)。此外,中介分析显示,丙酮酸可能介导了S. bovis对饲料效率的影响(pmdei < 0.05,见图6D)。

图6. Selenomonas bovis参与高效饲料利用奶牛瘤胃功能中的代谢途径及其与关键功能的因果关系
(A)S. bovis参与的关键营养代谢途径中的蛋白质 (B)S. bovis参与的细菌趋化作用功能(C)通过Mendelian随机化(MR)分析评估S. bovis与功能性蛋白质/代谢物之间的因果关系,显示了MR效应大小(β)和95%置信区间(CI)(D)S. bovis通过关键功能性蛋白质/代谢物直接介导饲料效率的作用,以及来自中介分析的相应p值(pmdei)。蓝色线条表示各因素之间的相关性,显示了Spearman系数和p值;MCP代表甲基受体去化蛋白,CI代表置信区间。
总体而言,通过利用瘤胃代蛋白组、代转录组和代谢组数据,我们克服了以往研究的局限性,揭示了瘤胃微生物真实的功能分布及其对饲料效率的影响。结合宿主遗传数据,我们建立了微生物、蛋白质/代谢物和表型之间的潜在因果关系,并识别出影响宿主饲料效率的瘤胃微生物群体内的不同功能模式。值得注意的是,我们建立了S. bovis与瘤胃碳水化合物代谢之间的潜在因果关系,可能通过细菌趋化作用和双组分调节系统介导,最终影响乳牛的饲料利用效率。这些发现揭示了瘤胃微生物与宿主表型之间复杂的相互作用,为我们理解瘤胃微生物功能及其对动物生产的影响提供了重要见解。
讨 论
理解瘤胃微生物群内复杂的相互作用和关键功能群对于改善畜牧生产和减少环境影响至关重要。以往研究已确定多种关键功能群在瘤胃中发挥重要作用,包括纤维素分解细菌、甲烷生成菌、琥珀酸代谢细菌和乳酸利用细菌,它们在植物纤维分解、甲烷产生和瘤胃 pH 调节中发挥关键作用。而我们的研究侧重于识别核心功能群,并探索它们在瘤胃生态系统中的潜力和活动贡献。本研究结果显示,微生物功能群在碳水化合物代谢、氨基酸代谢和细菌趋化等多个代谢途径中发挥重要作用。关键蛋白质分析揭示了多种微生物(如Selenomonas sp.、Ruminobacter sp.、Succinivibrionaceae sp. 和 Prevotella sp.)在碳水化合物降解中的参与。特别是,S. bovis 和 P. bryantii 显著参与碳水化合物(植物纤维)和氨基酸代谢,可能原因包括它们的酶能力、遗传构成和生态位。此外,我们的研究还发现,S. bovis 通过 MCP、CheA 和 FlgE 等关键蛋白质积极参与细菌趋化作用途径。P. bryantii 在六碳糖降解、丙酮酸生成和三羧酸循环中可能发挥作用,是一种研究较多的益生菌,已在早期泌乳期奶牛中显示出对瘤胃发酵和乳品生产的有益影响。这些发现不仅突显了微生物功能群在代谢过程中的关键作用,还为细菌趋化作用途径提供了新见解。
在我们此前的报道中曾揭示了不同的相互作用机制下的瘤胃微生物群,并发现 S. bovis 是一个与琥珀酸产生菌正向相互作用的关键细菌,从而提高了饲料转化效率;结合宿主遗传分析还显示,S. bovis 是一种中度遗传的细菌,具有重要的调控潜力。结合本研究的结果,S. bovis 在改善反刍动物饲料转化效率方面的作用可以归因于几个因素:与琥珀酸产生菌的正向相互作用、遗传影响、在碳水化合物代谢中的优势、专化的生态位等因素。在我们的研究中,基于转录组学,我们发现 S. bovis 主导活跃的碳水化合物代谢功能。通过分析 S. bovis 在蛋白质水平上的功能机制和调控单元,我们发现 S. bovis 显著上调了鞭毛生成和细菌趋化作用,这与碳水化合物利用功能显著相互作用。这表明 S. bovis 通过对碳源底物的趋化和占据碳水化合物降解生态位,在高效牛瘤胃中发挥着重要作用。
我们的研究发现,在高效率奶牛的瘤胃中,S. bovis表现出显著的趋化反应能力。特别是,我们观察到其双组分信号转导系统中的甲基趋化蛋白MCP受体显著上调,伴随着鞭毛合成和趋化功能的增强。这些发现提示,通过MCP受体, S. bovis对碳源底物的趋化反应被有效调控,从而优化其在瘤胃内的营养利用能力,尤其是在碳水化合物降解过程中的生态位竞争。此外,我们的研究还揭示,高效率动物中,S. bovis的CheA和CheY也表现出显著上调,这表明其可能通过经典的趋化基因簇参与调控趋化行为。细菌的趋化能力使其能够在复杂的环境中精准定位和获取必需的营养物质,这对于提高奶牛的营养利用效率具有重要意义。为深入了解S. bovis趋化信号传导途径的详细机制,未来的研究可以采用先进的分子生物学和生物信息学技术,如微生物的纯培养方法、基因编辑技术以及分子水平的对接实验。这些方法将有助于揭示MCP受体介导的经典趋化基因簇在S. bovis中的存在及其下游信号传导路径,进一步理解细菌如何通过趋化机制在瘤胃微环境中进行生态适应和资源利用优化的策略。
结 论
在本研究中,我们报道了瘤胃中的蛋白质水平信息,并整合了多种宏组学技术,探索了RNA、蛋白质和代谢产物水平的功能群及其冗余性。此外,我们发现了S. bovis与瘤胃碳水化合物代谢之间的因果联系,可能通过细菌趋化作用和双组分调控系统介导,影响奶牛饲料利用效率。我们的研究结果暗示了对瘤胃微生物群与其功能终点及其与表型关联的微妙理解。我们对高饲料效率奶牛瘤胃中关键营养功能群的解析、以及鉴定关键微生物功能蛋白质的锚定,为瘤胃微生物研究从探索组学发现之间的相关性过渡到阐明潜在因果关系提供了前瞻性基础。未来,致力于探索因果关系的更大规模研究、分离和培养关键功能菌株、进行菌株水平的机制研究,以及利用合成生物学实施微生物群靶向修饰,将进一步推动我们对瘤胃微生物群落和功能的理解,并在此研究的洞见基础上开展创新应用。
引文格式:
Ming-Yuan Xue, Yun-Yi Xie, Xin-Wei Zang, Yi-Fan Zhong, Xiao-Jiao Ma, Hui-Zeng Sun, Jian-Xin Liu. 2024. "Deciphering functional groups of rumen microbiome and their underlying potentially causal relationships in shaping host traits." iMeta e225. https://doi.org/10.1002/imt2.225.
作者简介

薛茗元(第一作者)
● 湘湖实验室(农业浙江省实验室)研究员,国家“博新计划”获得者。
● 研究方向为动物消化道微生物资源挖掘与工程化利用。以第一作者(含共同第一作者)在Nature Microbiology、Microbiome、Applied and Environmental Microbiology等微生物学权威期刊发表系列研究成果。主持国家自然科学基金、国家重点研发计划课题任务、中国博士后科学基金等项目。相关微生物组学和生物信息学研究成果获得软件著作权3项。

刘建新(通讯作者)
● 浙江大学动物科学学院教授,浙江大学求是特聘教授,浙江大学畜牧学科负责人,国家杰出青年科学基金获得者。
● 研究方向为反刍动物营养、乳品质形成机理及其调控、饲料资源开发利用。以通讯作者在Microbiome、Bioinformatics、Journal of Dairy Science等期刊发表研究成果。承担国家973计划课题、国家杰出青年基金、国家自然科学基金、国家支撑计划课题、国际合作重点项目等30多项国家、省部级科研项目。

孙会增(通讯作者)
● 浙江大学动物科学学院研究员,国家优秀青年科学基金获得者。
● 研究方向为奶牛营养与代谢、胃肠道功能与微生物组。以通讯作者在Nature Microbiology、Microbiome、Journal of Advanced Research等高影响期刊发表研究成果。承担国家优秀青年科学基金、国家重点研发计划青年科学家项目、浙江省杰出青年科学基金项目等国家、省部级科研项目。
更多推荐
(▼ 点击跳转)
iMeta | 引用13000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百千华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表所有领域高影响力的研究、方法和综述,重点关注微生物组、生物信息、大数据和多组学等。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!发行后相继被Google Scholar、ESCI、PubMed、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.7,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,同学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,是定位IF>10的高水平综合期刊,欢迎投稿!
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science









































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








