前言
接了个项目,需要将病理细胞图片svs格式的图片显示在前端页面上。找遍网络,有不可行的,有可行但却有bug的。在此整理份完整可行的方案,供大家参考。
细胞图片长这样:
此类svs图片一般大小在几百兆到几个G之间,着实很大,浏览器无法加载。pc一般是通过桌面软件加载查看的。
解决方案
解决方案有两种常见的方案:
- openslide
此方案这里不在赘述,我测试过,加载了一个300多兆的svs图,运行时直接崩了。因为过程繁琐,太耗内存,就此作罢。感兴趣的同学可以自行实验。官网地址在此: openslide - libvips
此方案是我们这次要用到的。通过安装此库文件,把svs转换后即可显示。
开干吧!
1.安装 pyvips
pip install pyvips
注意 :如果这时候直接这样写代码
import pyvips
这样是会报错的。
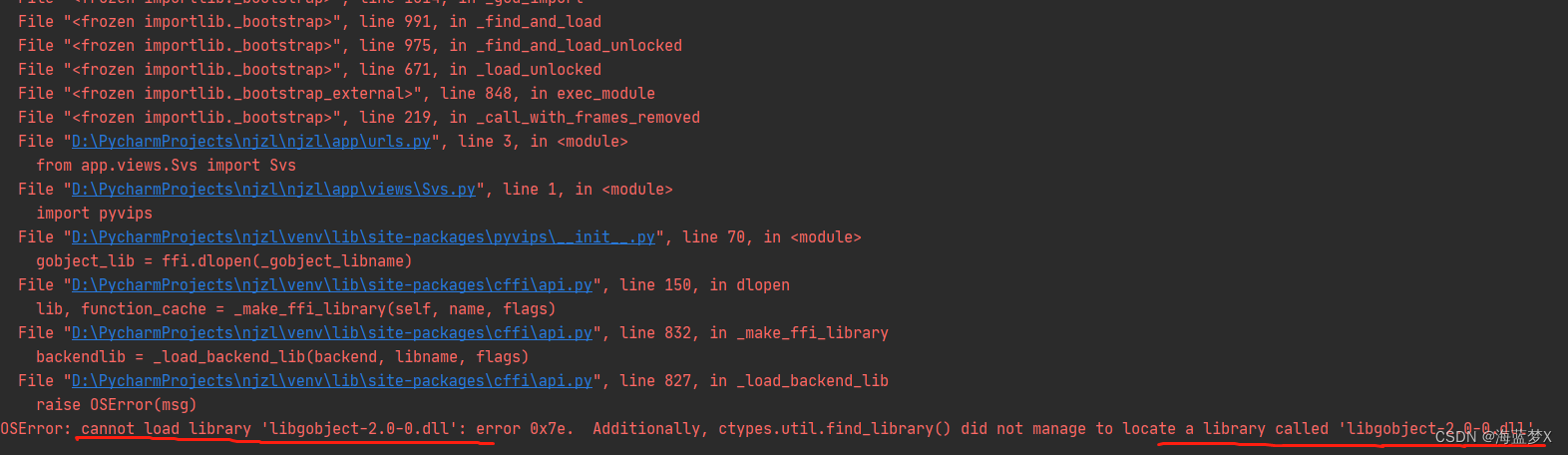
以上是windows的报错,linux也会有相似的报错。这是因为没有加载 libgobject 库文件。
2. 加载 libgobject 库文件
linux系统很简单:
sudo apt install libvips
windows系统就复杂点了
- 下载一个库文件 vips-dev
下载地址:https://github.com/libvips/build-win64-mxe/releases
我下载的是 v8.14.3版本
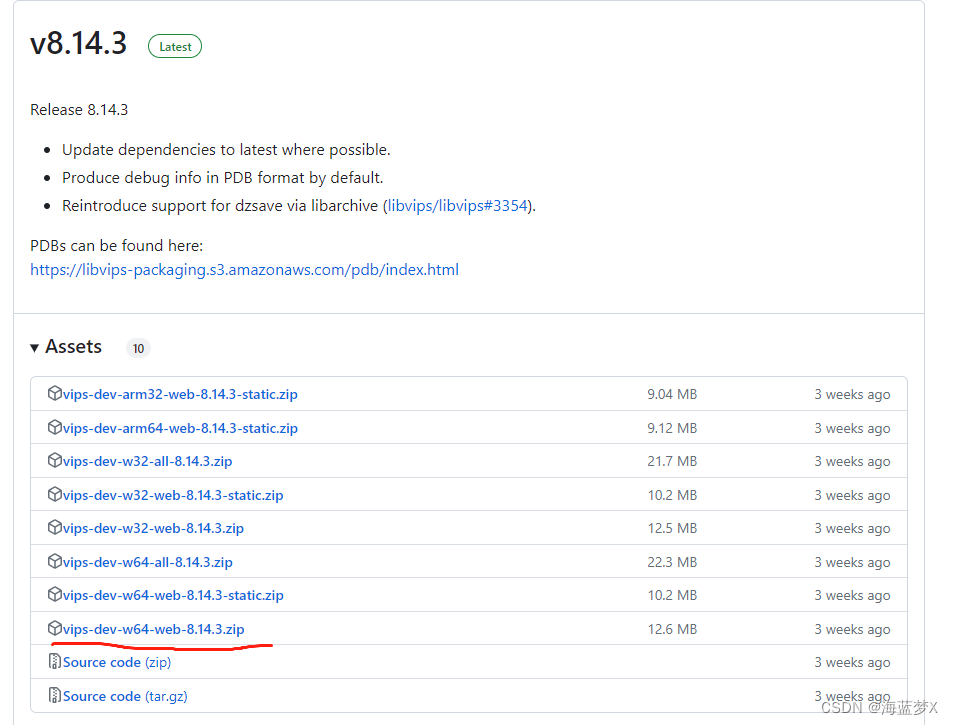
下载完后解压 - 找到你的python安装目录,或虚拟环境的目录,打开这个目录:
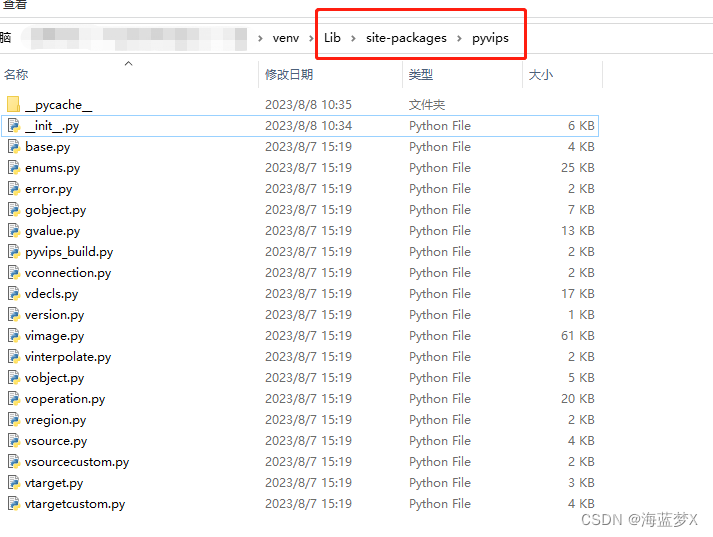
我是虚拟目录,所以在venv目录里。然后打开 _init_.py文件,在开头添加如下代码
import logging
import os
# =========在这里添加如下代码(vipshome地址是上面解压的文件所在的路径)========
vipshome = r'D:\PycharmProjects\njzl\install_package\vips-dev-8.14\bin'
os.environ['PATH'] = vipshome + ';' + os.environ['PATH']
#=======结束=======
import sys
import atexit
logger = logging.getLogger(__name__)
# user code can override this null handler
logger.addHandler(logging.NullHandler())
保存后在看下,是不不报错了
3. 开始写代码了
# 加载svs
img = pyvips.Image.new_from_file('static/files/1.svs', access='sequential')
# 生成dzi文件
img.dzsave('static/files/2')
加载svs文件,然后生成 dzi 文件。
dzi 文件是类似地图的瓦片文件,就是按层级分割成不同大小的图片,然后组合成一张大图。文件像下面这样:
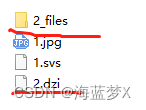
2_files 如下:
这样的文件,前端是可以加载显示的。
4.前端如何加载
前端vue 安装导入 openseadragon 库文件,然后加载远程dzi文件地址,就可以显示了。
前端代码不再这里追溯。参考文档:openseadragon





















 633
633











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








