小小实验室有钱啊,导师丢我两显卡,希望我加快计算进程。(绞尽脑汁,尝试修改alphafold官网提供的源代码,,,,未果,能力有限,无法从源头上实现数据并行【蛋白序列很多,为了加速运行,让显卡各自运行不同的模型,这可以减少整个项目的完成时间】或者是模型并行【相当于蛋白序列很长,一个显卡显存不够用,所以模型跨越在两张显卡上】)走了很多“邪门歪道”,可以实现,我不仅把双显卡分开用(即实现数据并行),还把内存条分开用了( ̄□ ̄||),真是个小机灵鬼。
1.首先先把alphafold软件文件内的几个文件修改一下
文件所在位置如下显示:
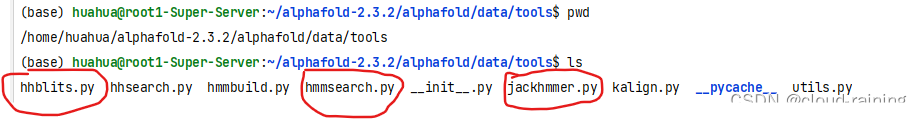
1.1使用htop查看几条内存条可用,下图显示本机有32条可用,
 1.2上一步中得到本机的内存条数后,按需求修改下列文件
1.2上一步中得到本机的内存条数后,按需求修改下列文件
vim hhblits.py #注意hhblits.py所在路径要对哈
我记得不错的话,原文件的n_cpu: int = 8,根据我的需要我把它改为16(本机内存条数为32,即显卡分开用,那么内存也分开用,达到利用最大化好了,,,)

vim hmmsearch.py
解释:同修改上一个文件的原因一样

vim jackhmmer.py

2.vim创建两个脚本,例子:al.sh和alpha.sh
代码中有些地方看不懂的话,看这篇博文的解释o(╥﹏╥)o本地版alphafold2批量处理序列-CSDN博客
vim al.sh #内容如下
#!/bin/bash
# 遍历Fasta文件目录下的所有文件
#!/bin/bash
export TF_FORCE_UNIFIED_MEMORY=1
export XLA_PYTHON_CLIENT_MEM_FRACTION=4.0
# 遍历Fasta文件目录下的所有文件
for file in /data1/huahua/proin/proin/crh1/xin-crh1-2/*; do
# 提取文件名(不包含路径)
filename=$(basename "$file")
# 运行AlphaFold预测命令
echo "Predicting $filename..."
CUDA_VISIBLE_DEVICES=1 taskset -c 0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15 python run_alphafold.py --data_dir=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes --uniref90_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/uniref90/uniref90.fasta --mgnify_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/mgnify/mgy_clusters_2022_05.fa --template_mmcif_dir=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/pdb_mmcif/mmcif_files --max_template_date=2021-11-01 --obsolete_pdbs_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/pdb_mmcif/obsolete.dat --model_preset=multimer --uniprot_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/uniprot/uniprot.fasta --pdb_seqres_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/pdb_seqres/pdb_seqres.txt --db_preset=reduced_dbs --small_bfd_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldData/small_bfd/bfd-first_non_consensus_sequences.fasta --fasta_paths="$file" --output_dir=/data1/huahua/proin/out-m/crh1/xin-crh1-2 --use_gpu_relax=true --benchmark=true --models_to_relax=best --num_multimer_predictions_per_model=1 --use_precomputed_msas=true
done
#其中CUDA_VISIBLE_DEVICES=1指定显卡1
#taskset -c 0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15 指定显存条前16个
#后面代码是alphafold2的运行常规代码。本例为预测复合体。
#注意所有文件所在路径!!!!!!vim alpha.sh #内容如下
#!/bin/bash
# 遍历Fasta文件目录下的所有文件
#!/bin/bash
export TF_FORCE_UNIFIED_MEMORY=1
export XLA_PYTHON_CLIENT_MEM_FRACTION=4.0
# 遍历Fasta文件目录下的所有文件
for file in /data1/huahua/proin/proin/crh1/xin-crh1-1/*; do
# 提取文件名(不包含路径)
filename=$(basename "$file")
# 运行AlphaFold预测命令
echo "Predicting $filename..."
CUDA_VISIBLE_DEVICES=0 taskset -c 16,17,18,19,20,21,22,23,24,25,26,27,28,29,10,31 python run_alphafold.py --data_dir=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes --uniref90_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/uniref90/uniref90.fasta --mgnify_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/mgnify/mgy_clusters_2022_05.fa --template_mmcif_dir=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/pdb_mmcif/mmcif_files --max_template_date=2021-11-01 --obsolete_pdbs_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/pdb_mmcif/obsolete.dat --model_preset=multimer --uniprot_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/uniprot/uniprot.fasta --pdb_seqres_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldDataRes/pdb_seqres/pdb_seqres.txt --db_preset=reduced_dbs --small_bfd_database_path=/data1/luping/alphaFold/alphaFold2/alphafold/scripts/AlphaFoldData/small_bfd/bfd-first_non_consensus_sequences.fasta --fasta_paths="$file" --output_dir=/data1/huahua/proin/out-m/crh1/xin-crh1-1 --use_gpu_relax=true --benchmark=true --models_to_relax=best --num_multimer_predictions_per_model=1 --use_precomputed_msas=true
done
#其中CUDA_VISIBLE_DEVICES=0指定显卡0
#taskset -c 16,17,18,19,20,21,22,23,24,25,26,27,28,29,10,31 指定显存条后16个
#后面代码是alphafold2的运行常规代码。本例为预测复合体。
#注意所有文件所在路径!!!!!!3.最后使用nohup分别运行这两个文件即可
nohup al.sh > out.txt & #注意al.sh所在路径!!! 其中&表示在后台运行
nohup alpha.sh > out1.txt &
注:运行后检查是否有报错,分别运行cat out.txt和cat out1.txt查看是否有其他报错(只要前期alphafold2安装好、数据库下载好、环境配置好、文件所在路径正确、运行nohup时是在alphafold的安装路径下、两个脚本的路径正确,那么你就成功了)。





















 427
427











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








