1、原理简介
我们常见的缺失值处理方法主要有: 删除、 用平均值或中位数替代、回归插值 、多元回归插值。而这次我们主要看一下多重插补的代码实现。多重插补主要是根据链式方程进行,大致步骤如下:1、针对存在缺失值的k个变量,每个变量指定一个插补模型。 2、分别对存在缺失值的变量,由其观测值的总体分布中抽取用于替换缺失值的插补值,但注意,这并不等于最终插补值。 3、对于存在缺失值的第一个变量——先回归:基于其他变量对第一个变量进行回归。提取回归参数:从上一步回归中提取该回归模型的估计回归系数及方差-协方差矩阵。计算分布:利用上一步获得的扰动回归系数,针对每个具有缺失数据的个体,确定第一个变量的条件性分布。数值插补:根据上一步获得的条件性分布,为每一个缺失数据选择插补值。 4、重复步骤3,插补每一个具有缺失数据的变量。 5、重复步骤3,步骤4,设定重复次数,一般推荐5-20次;.最终的插补值被作为第一个插补数据集中的插补值; 6、重复步骤3、4、5,共N次,产生N个插补数据集。
决策曲线分析:该方法中有两个重要概念,风险阈值和净收益。当超过风险阈值时,将对该个体采取干预措施。而净收益则可以表示为“净阳性人数”/研究总人数。而在决策曲线中一条线是不对任何人进行干预,称为treat_none,另一条线是对所有人进行干预,称为treat_all。对于预测模型,在某个阈值概率下,只有其NB均高于treat_all和treat_none的情况下,模型才有实际价值。如果模型NB低于treat_all和treat_none中任意一条,则该模型不太具有价值。
2、多重插补的代码实现
这里我们使用了mice包
install.packages("mice")
library(mice)
install.packages("VIM")
library(VIM)
sleep#VIM中的一个数据集
#sleep
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.00 NA NA 3.3 38.6 645.0 3 5 3
2 1.000 6.60 6.3 2.0 8.3 4.5 42.0 3 1 3
3 3.385 44.50 NA NA 12.5 14.0 60.0 1 1 1
4 0.920 5.70 NA NA 16.5 NA 25.0 5 2 3
5 2547.000 4603.00 2.1 1.8 3.9 69.0 624.0 3 5 4
6 10.550 179.50 9.1 0.7 9.8 27.0 180.0 4 4 4
7 0.023 0.30 15.8 3.9 19.7 19.0 35.0 1 1 1
8 160.000 169.00 5.2 1.0 6.2 30.4 392.0 4 5 4
9 3.300 25.60 10.9 3.6 14.5 28.0 63.0 1 2 1
10 52.160 440.00 8.3 1.4 9.7 50.0 230.0 1 1 1
11 0.425 6.40 11.0 1.5 12.5 7.0 112.0 5 4 4
12 465.000 423.00 3.2 0.7 3.9 30.0 281.0 5 5 5
13 0.550 2.40 7.6 2.7 10.3 NA NA 2 1 2
14 187.100 419.00 NA NA 3.1 40.0 365.0 5 5 5
15 0.075 1.20 6.3 2.1 8.4 3.5 42.0 1 1 1
16 3.000 25.00 8.6 0.0 8.6 50.0 28.0 2 2 2
17 0.785 3.50 6.6 4.1 10.7 6.0 42.0 2 2 2
18 0.200 5.00 9.5 1.2 10.7 10.4 120.0 2 2 2
19 1.410 17.50 4.8 1.3 6.1 34.0 NA 1 2 1
20 60.000 81.00 12.0 6.1 18.1 7.0 NA 1 1 1
21 529.000 680.00 NA 0.3 NA 28.0 400.0 5 5 5
22 27.660 115.00 3.3 0.5 3.8 20.0 148.0 5 5 5
23 0.120 1.00 11.0 3.4 14.4 3.9 16.0 3 1 2
24 207.000 406.00 NA NA 12.0 39.3 252.0 1 4 1
25 85.000 325.00 4.7 1.5 6.2 41.0 310.0 1 3 1
26 36.330 119.50 NA NA 13.0 16.2 63.0 1 1 1
27 0.101 4.00 10.4 3.4 13.8 9.0 28.0 5 1 3
28 1.040 5.50 7.4 0.8 8.2 7.6 68.0 5 3 4
29 521.000 655.00 2.1 0.8 2.9 46.0 336.0 5 5 5
30 100.000 157.00 NA NA 10.8 22.4 100.0 1 1 1
31 35.000 56.00 NA NA NA 16.3 33.0 3 5 4
32 0.005 0.14 7.7 1.4 9.1 2.6 21.5 5 2 4
33 0.010 0.25 17.9 2.0 19.9 24.0 50.0 1 1 1
34 62.000 1320.00 6.1 1.9 8.0 100.0 267.0 1 1 1
35 0.122 3.00 8.2 2.4 10.6 NA 30.0 2 1 1
36 1.350 8.10 8.4 2.8 11.2 NA 45.0 3 1 3
37 0.023 0.40 11.9 1.3 13.2 3.2 19.0 4 1 3
38 0.048 0.33 10.8 2.0 12.8 2.0 30.0 4 1 3
39 1.700 6.30 13.8 5.6 19.4 5.0 12.0 2 1 1
40 3.500 10.80 14.3 3.1 17.4 6.5 120.0 2 1 1
41 250.000 490.00 NA 1.0 NA 23.6 440.0 5 5 5
42 0.480 15.50 15.2 1.8 17.0 12.0 140.0 2 2 2
43 10.000 115.00 10.0 0.9 10.9 20.2 170.0 4 4 4
44 1.620 11.40 11.9 1.8 13.7 13.0 17.0 2 1 2
45 192.000 180.00 6.5 1.9 8.4 27.0 115.0 4 4 4
46 2.500 12.10 7.5 0.9 8.4 18.0 31.0 5 5 5
47 4.288 39.20 NA NA 12.5 13.7 63.0 2 2 2
48 0.280 1.90 10.6 2.6 13.2 4.7 21.0 3 1 3
49 4.235 50.40 7.4 2.4 9.8 9.8 52.0 1 1 1
50 6.800 179.00 8.4 1.2 9.6 29.0 164.0 2 3 2
51 0.750 12.30 5.7 0.9 6.6 7.0 225.0 2 2 2
52 3.600 21.00 4.9 0.5 5.4 6.0 225.0 3 2 3
53 14.830 98.20 NA NA 2.6 17.0 150.0 5 5 5
54 55.500 175.00 3.2 0.6 3.8 20.0 151.0 5 5 5
55 1.400 12.50 NA NA 11.0 12.7 90.0 2 2 2
56 0.060 1.00 8.1 2.2 10.3 3.5 NA 3 1 2
57 0.900 2.60 11.0 2.3 13.3 4.5 60.0 2 1 2
58 2.000 12.30 4.9 0.5 5.4 7.5 200.0 3 1 3
59 0.104 2.50 13.2 2.6 15.8 2.3 46.0 3 2 2
60 4.190 58.00 9.7 0.6 10.3 24.0 210.0 4 3 4
61 3.500 3.90 12.8 6.6 19.4 3.0 14.0 2 1 1
62 4.050 17.00 NA NA NA 13.0 38.0 3 1 1
sleep[!complete.cases(sleep),]#单独查看含有缺失值数据
#含缺失值数据
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 3
3 3.385 44.5 NA NA 12.5 14.0 60 1 1 1
4 0.920 5.7 NA NA 16.5 NA 25 5 2 3
13 0.550 2.4 7.6 2.7 10.3 NA NA 2 1 2
14 187.100 419.0 NA NA 3.1 40.0 365 5 5 5
19 1.410 17.5 4.8 1.3 6.1 34.0 NA 1 2 1
20 60.000 81.0 12.0 6.1 18.1 7.0 NA 1 1 1
21 529.000 680.0 NA 0.3 NA 28.0 400 5 5 5
24 207.000 406.0 NA NA 12.0 39.3 252 1 4 1
26 36.330 119.5 NA NA 13.0 16.2 63 1 1 1
30 100.000 157.0 NA NA 10.8 22.4 100 1 1 1
31 35.000 56.0 NA NA NA 16.3 33 3 5 4
35 0.122 3.0 8.2 2.4 10.6 NA 30 2 1 1
36 1.350 8.1 8.4 2.8 11.2 NA 45 3 1 3
41 250.000 490.0 NA 1.0 NA 23.6 440 5 5 5
47 4.288 39.2 NA NA 12.5 13.7 63 2 2 2
53 14.830 98.2 NA NA 2.6 17.0 150 5 5 5
55 1.400 12.5 NA NA 11.0 12.7 90 2 2 2
56 0.060 1.0 8.1 2.2 10.3 3.5 NA 3 1 2
62 4.050 17.0 NA NA NA 13.0 38 3 1 1
md.pattern(sleep)
aggr(sleep,prop=FALSE,numbers=TRUE)#可视化缺失值总体情况
缺失值情况可视化如下,当然这只是在数据量大时帮助我们判断整体情况的工具,最主要的是拿到我们插补的数据集。
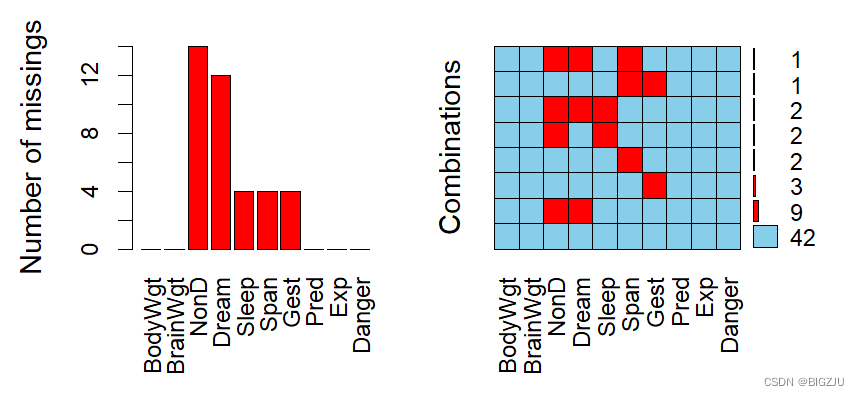
接下来我们使用mice()进行插补,并查看插补数据集,可见确实数据已经补上。
imp<-mice(sleep,seed=5)#进行数据集插补,一般默认插补次数为5
#mice(sleep, meth = c("sample", "pmm", "logreg", "norm")) 可以给每一列设置不同的插补方法
complete(imp)#查看具体的插补情况
#第一个插补数据集
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.00 3.3 0.5 3.3 38.6 645.0 3 5 3
2 1.000 6.60 6.3 2.0 8.3 4.5 42.0 3 1 3
3 3.385 44.50 10.8 1.9 12.5 14.0 60.0 1 1 1
4 0.920 5.70 15.8 0.5 16.5 4.7 25.0 5 2 3
5 2547.000 4603.00 2.1 1.8 3.9 69.0 624.0 3 5 4
6 10.550 179.50 9.1 0.7 9.8 27.0 180.0 4 4 4
7 0.023 0.30 15.8 3.9 19.7 19.0 35.0 1 1 1
8 160.000 169.00 5.2 1.0 6.2 30.4 392.0 4 5 4
9 3.300 25.60 10.9 3.6 14.5 28.0 63.0 1 2 1
10 52.160 440.00 8.3 1.4 9.7 50.0 230.0 1 1 1
11 0.425 6.40 11.0 1.5 12.5 7.0 112.0 5 4 4
12 465.000 423.00 3.2 0.7 3.9 30.0 281.0 5 5 5
13 0.550 2.40 7.6 2.7 10.3 2.3 42.0 2 1 2
14 187.100 419.00 3.2 0.5 3.1 40.0 365.0 5 5 5
15 0.075 1.20 6.3 2.1 8.4 3.5 42.0 1 1 1
16 3.000 25.00 8.6 0.0 8.6 50.0 28.0 2 2 2
17 0.785 3.50 6.6 4.1 10.7 6.0 42.0 2 2 2
18 0.200 5.00 9.5 1.2 10.7 10.4 120.0 2 2 2
19 1.410 17.50 4.8 1.3 6.1 34.0 164.0 1 2 1
20 60.000 81.00 12.0 6.1 18.1 7.0 120.0 1 1 1
21 529.000 680.00 4.7 0.3 3.9 28.0 400.0 5 5 5
22 27.660 115.00 3.3 0.5 3.8 20.0 148.0 5 5 5
23 0.120 1.00 11.0 3.4 14.4 3.9 16.0 3 1 2
24 207.000 406.00 8.3 3.9 12.0 39.3 252.0 1 4 1
25 85.000 325.00 4.7 1.5 6.2 41.0 310.0 1 3 1
26 36.330 119.50 10.8 2.4 13.0 16.2 63.0 1 1 1
27 0.101 4.00 10.4 3.4 13.8 9.0 28.0 5 1 3
28 1.040 5.50 7.4 0.8 8.2 7.6 68.0 5 3 4
29 521.000 655.00 2.1 0.8 2.9 46.0 336.0 5 5 5
30 100.000 157.00 8.4 2.3 10.8 22.4 100.0 1 1 1
31 35.000 56.00 10.6 0.5 11.0 16.3 33.0 3 5 4
32 0.005 0.14 7.7 1.4 9.1 2.6 21.5 5 2 4
33 0.010 0.25 17.9 2.0 19.9 24.0 50.0 1 1 1
34 62.000 1320.00 6.1 1.9 8.0 100.0 267.0 1 1 1
35 0.122 3.00 8.2 2.4 10.6 4.5 30.0 2 1 1
36 1.350 8.10 8.4 2.8 11.2 3.5 45.0 3 1 3
37 0.023 0.40 11.9 1.3 13.2 3.2 19.0 4 1 3
38 0.048 0.33 10.8 2.0 12.8 2.0 30.0 4 1 3
39 1.700 6.30 13.8 5.6 19.4 5.0 12.0 2 1 1
40 3.500 10.80 14.3 3.1 17.4 6.5 120.0 2 1 1
41 250.000 490.00 4.9 1.0 6.1 23.6 440.0 5 5 5
42 0.480 15.50 15.2 1.8 17.0 12.0 140.0 2 2 2
43 10.000 115.00 10.0 0.9 10.9 20.2 170.0 4 4 4
44 1.620 11.40 11.9 1.8 13.7 13.0 17.0 2 1 2
45 192.000 180.00 6.5 1.9 8.4 27.0 115.0 4 4 4
46 2.500 12.10 7.5 0.9 8.4 18.0 31.0 5 5 5
47 4.288 39.20 8.6 4.1 12.5 13.7 63.0 2 2 2
48 0.280 1.90 10.6 2.6 13.2 4.7 21.0 3 1 3
49 4.235 50.40 7.4 2.4 9.8 9.8 52.0 1 1 1
50 6.800 179.00 8.4 1.2 9.6 29.0 164.0 2 3 2
51 0.750 12.30 5.7 0.9 6.6 7.0 225.0 2 2 2
52 3.600 21.00 4.9 0.5 5.4 6.0 225.0 3 2 3
53 14.830 98.20 2.1 0.6 2.6 17.0 150.0 5 5 5
54 55.500 175.00 3.2 0.6 3.8 20.0 151.0 5 5 5
55 1.400 12.50 7.4 4.1 11.0 12.7 90.0 2 2 2
56 0.060 1.00 8.1 2.2 10.3 3.5 30.0 3 1 2
57 0.900 2.60 11.0 2.3 13.3 4.5 60.0 2 1 2
58 2.000 12.30 4.9 0.5 5.4 7.5 200.0 3 1 3
59 0.104 2.50 13.2 2.6 15.8 2.3 46.0 3 2 2
60 4.190 58.00 9.7 0.6 10.3 24.0 210.0 4 3 4
61 3.500 3.90 12.8 6.6 19.4 3.0 14.0 2 1 1
62 4.050 17.00 2.1 0.9 2.9 13.0 38.0 3 1 1接下来我们就可以利用插补数据集进行数据分析。具体的分析方法可根据自己的目的使用。最后使用pool()将多个插补数据集的结果合并。
fit<-with(imp,lm(Dream~Span+Gest))#具体的数据分析
pooled<-pool(fit)#合并最终结果
summary(pooled)
#合并结果
term estimate std.error statistic df p.value
1 (Intercept) 2.588645530 0.255292581 10.1399168 51.80427 6.701900e-14
2 Span -0.004811583 0.012047819 -0.3993738 53.08899 6.912200e-01
3 Gest -0.004009873 0.001528571 -2.6232814 48.41812 1.161242e-02
3、决策曲线分析的代码实现
这里我们主要使用rmda包中的decision_curve ()。
install.packages("rmda")
library(rmda)
data("dcaData", package="rmda")#dcaData为包里的一个癌症数据集
head(dcaData)
#数据集中包含年龄、性别、吸烟、Marker1、Marker2等变量以及结局事件Cancer
Age Female Smokes Marker1 Marker2 Cancer
<int> <dbl> <lgl> <dbl> <dbl> <int>
1 33 1 FALSE 0.245 1.02 0
2 29 1 FALSE 0.943 -0.256 0
3 28 1 FALSE 0.774 0.332 0
4 27 0 FALSE 0.406 -0.00569 0
5 23 1 FALSE 0.508 0.208 0
6 35 1 FALSE 0.186 1.41 0
7 34 1 FALSE 0.621 0.615 0
8 29 1 FALSE 0.402 1.16 0
9 35 1 FALSE 0.390 1.38 0
10 27 1 FALSE 0.151 2.44 0接下来我们进行决策曲线的分析及图形绘制。
model <- decision_curve(Cancer~Age + Female + Smokes,
family=binomial(link=logit), #logistic回归模型进行拟合
data = dcaData,
thresholds = seq(0, 0.4, by = .005), #设置横坐标阈概率的范围
bootstraps = 10)
plot_decision_curve(model,
curve.names = "logistic model",
confidence.intervals=F,
legend.position = "topright",
standardize = FALSE,
cost.benefit.axis =FALSE) #不显示cost.benefit轴拟合结果如下,

我们也可以同时对多条决策曲线进行拟合。这里我们加入别的变量,拟合出另外两个模型。
model1 <- decision_curve(Cancer~Age + Female + Smokes+Marker1,
family=binomial(link=logit),
data = dcaData,
thresholds = seq(0, 0.4, by = .005),
bootstraps = 10)
model2 <- decision_curve(Cancer~Age + Female + Smokes+Marker1+Marker2,
family=binomial(link=logit),
data = dcaData,
thresholds = seq(0, 0.4, by = .005),
bootstraps = 10)
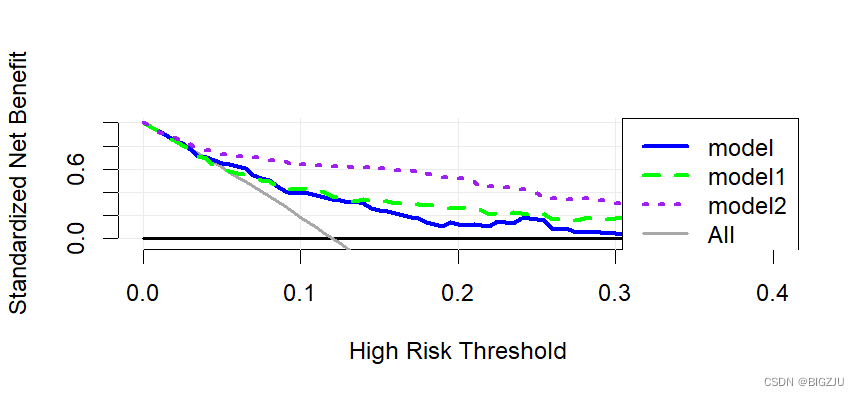
plot_decision_curve(list(model,model1,model2),
curve.names = c("model", "model1","model2"),
col = c("blue", "green","purple"),#设置线的颜色
lty = c(1,2,3), # 设置线形
lwd = c(3,3,3, 2, 2), #设置线宽,
confidence.intervals = F,
legend.position = "topright",
cost.benefit.axis =FALSE)结果如下,这样我们就可以清晰地对比不同模型的临床决策曲线,判断哪个模型更胜一筹了。
本期分享就到这里,我们下期见。





















 7667
7667











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








