WSL2-Ubuntu22.04 LTS安装conda,cuda,cudnn从0实现到验证安装成功,步骤详细
1.前言
1.之前跑深度学习都是在windows下,但是现在从github上下载的代码都是.sh文件,需要linux环境。加之我自己本身只有一台windows主机,所以需要在windows下安装wsl2(网上都说这个才可以支持显卡调用),所以就放弃了安装虚拟机等方法,在wsl2上安装了Ubuntu22.04版本,下面的步骤就是基于上述情况进行展开。
2.后续的目标是通过本地windows的PyChram专业版通过Remote Development远程开发连接到我自己的wsl2-Ubtuntu中,最终实现在windows下对linux代码的模型训练和验证。
2.首先检验自己的目前开发环境
在Ubuntu中进入自己的目录yjj中,运行下述代码,可以发现当前Ubuntu中没有任何的cuda工具包。
nvcc -V

3.下载并配置anaconda
3.1 下载anaconda
anaconda官网下载地址
可以发现Linux下有三个可以选择的安装包。

首先输入命令,查看自己符合的安装包。
uname -m
我的电脑是x86_64,所以下载第一个就可以。

接下来将下载好的安装包,我是放在windows下的E:\application\Anaconda3-2024.10-1-Linux-x86_64.sh中,此时需要在Ubuntu中进入mnt目录,因为Ubuntu不能识别windows路径,wsl2会将Windows的磁盘挂载在mnt路径下,如下图所示,进入mnt目录可以看到c盘和e盘,就是对应windows下的c盘和e盘。

3.2 安装anaconda
之后通过运行下面的代码,安装anaconda。一路点击空格+yes。跳转下方界面,会提示安装路径默认在/home/yjj/anaconda3下,选择默认路径就好。
bash e/application/Anaconda3-2024.10-1-Linux-x86_64.sh

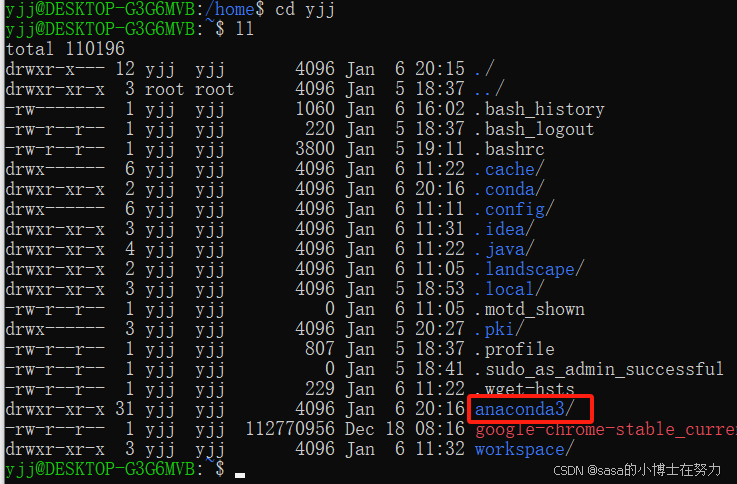
然后进入yjj目录,发现anaconda3已经安装到所在目录中。
3.3配置anaconda环境变量
接下来需要配置环境变量,首先输入
nano ~/.bashrc
进入环境变量文件,在最后一行导入anaconda的环境变量,如下图所示,之后ctrl+X,点击y,然后回车退出文件。(养成好习惯,最后输入source ~./bashrc更新配置环境文件)
export PATH="/home/yjj/anaconda3/bin:$PATH"

最后验证conda --version,显示版本,至此anaconda3在Ubuntu上安装完毕。

4.下载并配置cuda
4.1 下载并安装cuda
首先确定好自己的第一个项目所需要的虚拟环境的cuda版本,以我为例,因为我需要实现segment anything,github官方给出的建议是The code requires python>=3.8, as well as pytorch>=1.7 and torchvision>=0.8. Please follow the instructions here to install both PyTorch and TorchVision dependencies. Installing both PyTorch and TorchVision with CUDA support is strongly recommended.
根据pytorch官网,找到符合满足条件的cuda版本,我这里下载cuda12.2,下图所示cuda官网。在Ubuntun中依此运行下面七行代码。

运行完毕之后,打开Ubuntu的usr/local目录下,cuda被安装到这里

4.2 配置cuda环境变量
接下来需要配置cuda的环境变量。输入下述代码进入配置环境变量文件。
nano ~/.bashrc
根据自己下载的包,添加如下路径
export DISPLAY=172.21.64.1:0 #这个是之前配置wsl的IPV4地址,这里不需要配置
export PATH="/home/yjj/anaconda3/bin:$PATH" # 这个是上面配置anaconda的,这里也不需要配置
export PATH=/usr/local/cuda/bin:$PATH
export LD_LIBRARY_PATH=/usr/local/cuda/lib64:$LD_LIBRARY_PATH
export PATH=/usr/local/cuda-12.2/bin${PATH:+:${PATH}}
export LD_LIBRARY_PATH=/usr/local/cuda-12.2/lib64${LD_LIBRARY_PATH:+:${LD_LIBRARY_PATH}}
export CUDA_HOME=/usr/local/cuda-12.2

最后ctrl+x,y,回车退出,然后运行source ~/.bashrc更新配置文件,使改动生效。
最终输入nvcc -V验证cuda安装是否成功,如下图所示安装成功

5.下载并配置cudnn
5.1 下载cudnn
进入cudnn官网,选择适合上面安装cuda的cudnn版本https://developer.nvidia.com/rdp/cudnn-archive

之后把下载包复制到Ubuntu下的yjj目录下

5.2 解压并安装cudnn
依此执行下述命令,解压,复制cudnn中的bin等目录到cuda中(类似windows中把cudnn中的bin,lib,include三个目录中的文件复制到cuda对应目录中)。
tar -xvf cudnn-linux-x86_64-8.9.4.25_cuda12-archive.tar.xz
sudo cp cudnn-*-archive/include/cudnn*.h /usr/local/cuda/include
sudo cp -P cudnn-*-archive/lib/libcudnn* /usr/local/cuda/lib64
sudo chmod a+r /usr/local/cuda/include/cudnn*.h /usr/local/cuda/lib64/libcudnn*
最后输入下述命令验证是否安装成功,显示输出8,9,4代表安装的cudnn版本为8.9.4和上述下载的版本一致,至此cudnn的下载和安装结束。(提示 cannot use constexpr here since this is a C-only file 的问题是由于 constexpr 是 C++ 中的关键字,而这个文件是用 C 语言编写的(通常 C 文件以 .c 为后缀)。因此,constexpr 不能直接用于 C 语言文件中。
这并不表示 CUDNN 没有安装好,而是编译代码时出现了语言特性不兼容的问题。)
cat /usr/local/cuda/include/cudnn_version.h | grep CUDNN_MAJOR -A 2
























 282
282

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








