1 fastqc的常用参数
-o 设置输出目录 -t 线程,同时处理几个样本
2 脚本运行的三种方式
2.1 直接运行——霸占控制台
fastqc -t 6 -o ./ SRR*.fastq.gz
2.2 脚本后台运行:nohup &——适用于比较简单的命令
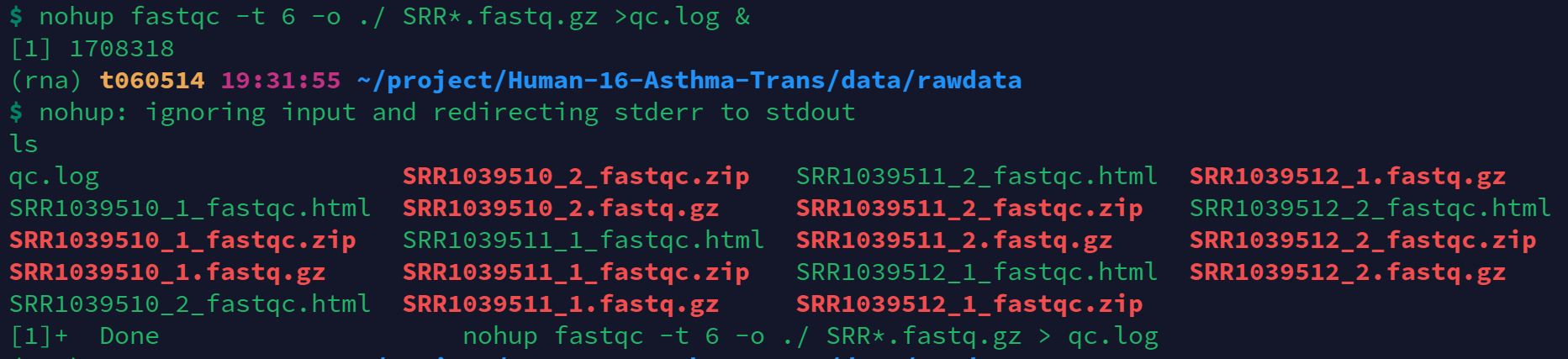 no hup:no hang up不挂起,退出终端不会影响程序的运行 &:后台运行 大于号:重定向,运行的过程写入日志文件中
no hup:no hang up不挂起,退出终端不会影响程序的运行 &:后台运行 大于号:重定向,运行的过程写入日志文件中
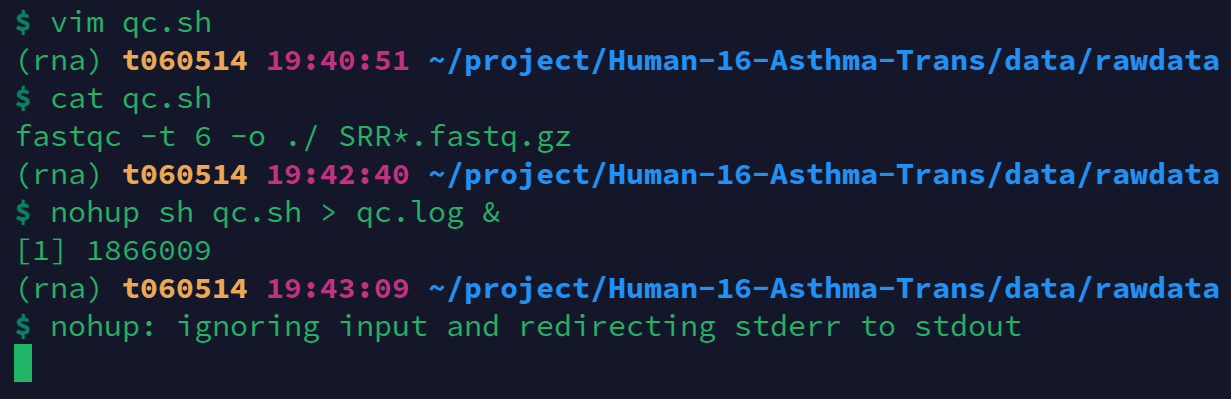
2.3 命令写入sh脚本,使用nohup+&运行脚本 ——适用于比较长和复杂的命令
vim三种模式命令的复习: vim进入命令行模式(默认) i进入编辑模式 esc返回命令行模式 :进入末行模式 wq保存并退出
3 fastqc运行结果
file:///C:/Users/Helen/AppData/Local/Temp/tmp-18968-M26D2xsk3OHC/SRR1039510_1_fastqc.html
3.1 Basic Statistics
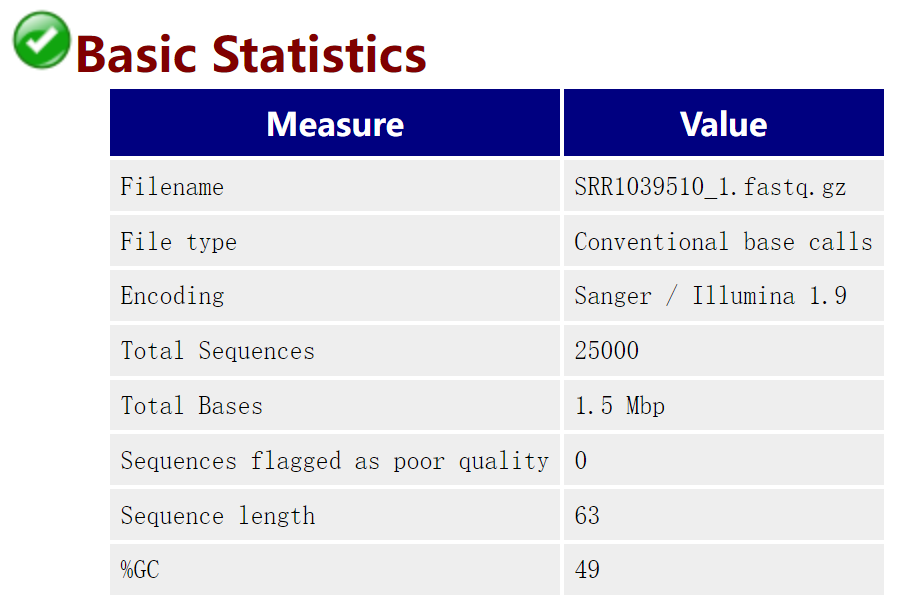 Total Sequences:测序数据的总reads数
Total Sequences:测序数据的总reads数
数据量统计方式
生物学中单位 以碱基(对)数或者测序read数统计测序数据量的大小
 计算机中物理存储单位
计算机中物理存储单位
3.2 Per base sequence quality每个碱基的序列质量值
横坐标:碱基的位置信息 纵坐标:每个位置上每个碱基对应的Q值——画成箱式图
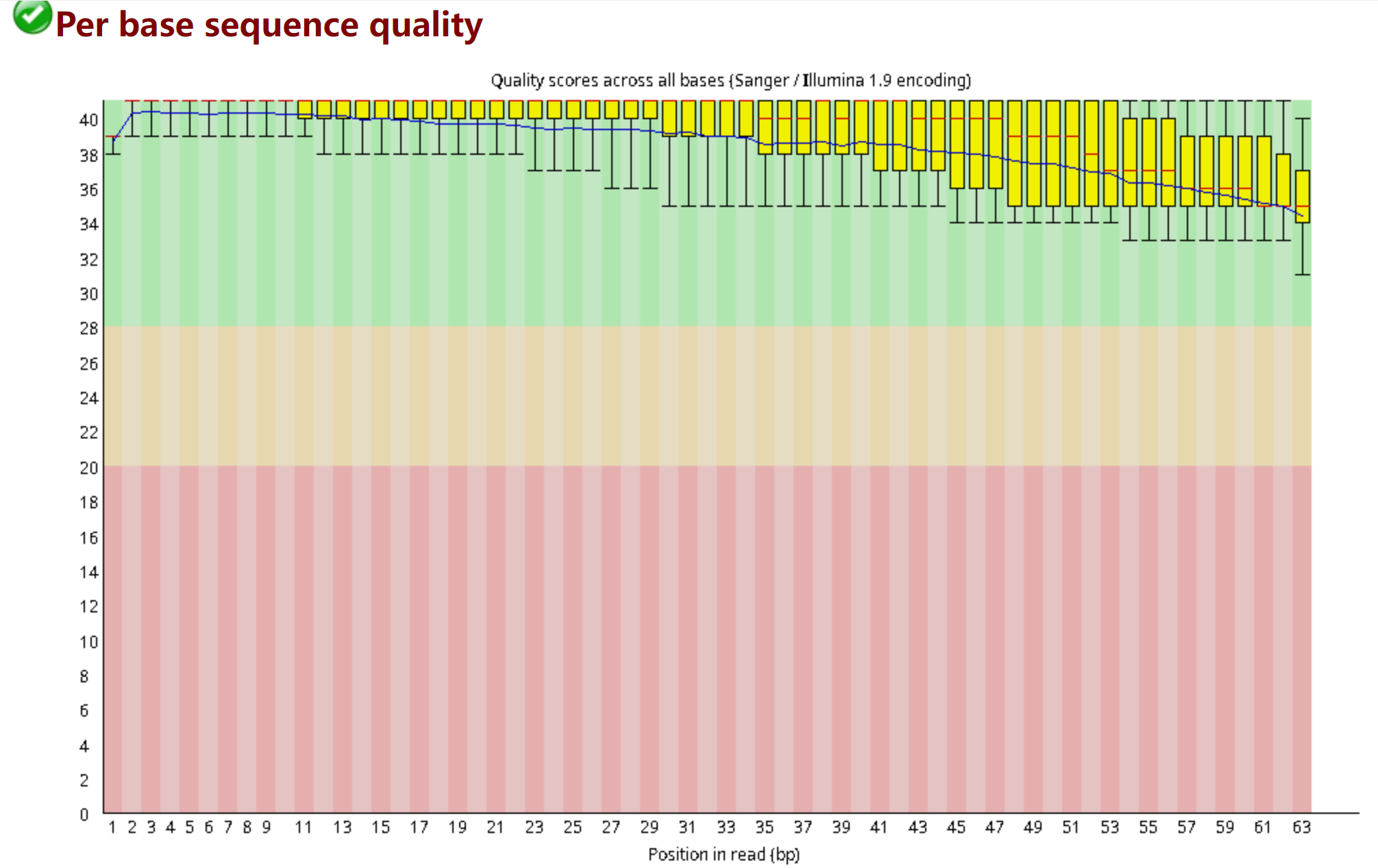 每个位置上碱基的Q值都落在绿色区域(Q30以上)
每个位置上碱基的Q值都落在绿色区域(Q30以上)
3.3 Per Tile Sequence Quality
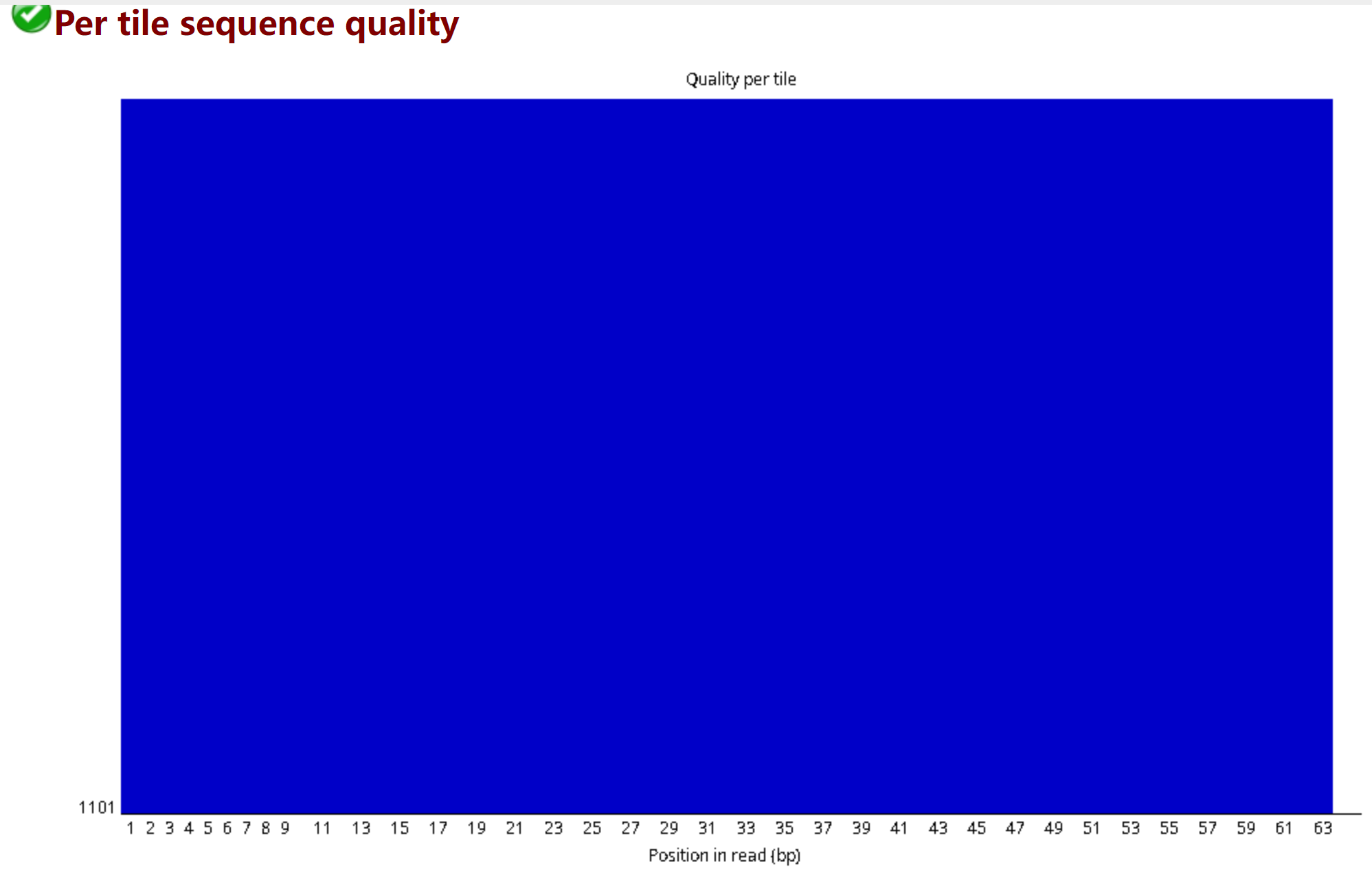 好的结果为全蓝色 测试数据只用了25000条
好的结果为全蓝色 测试数据只用了25000条
3.4 Per Sequence Quality Scores每条序列的碱基质量值
横坐标:平均质量值 纵坐标:每个质量值对应的reads数 真实数据拖尾分布 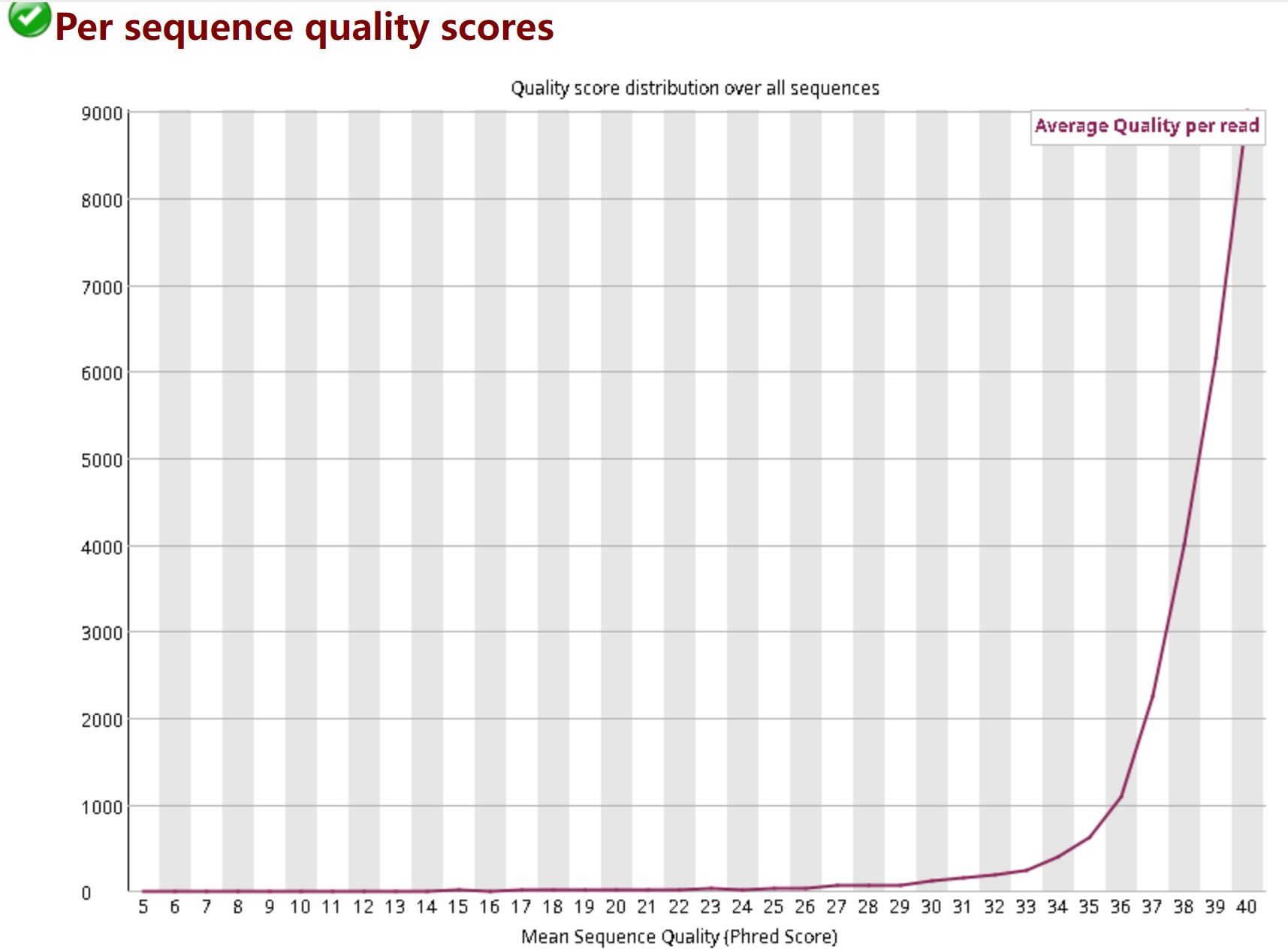
3.5 Per Base Sequence Content——每个碱基位置上ATGC含量分布图
理论上,G和C、A和T的含量每个测序循环上应分别相等,且整个测序过程稳定不变,呈水平线 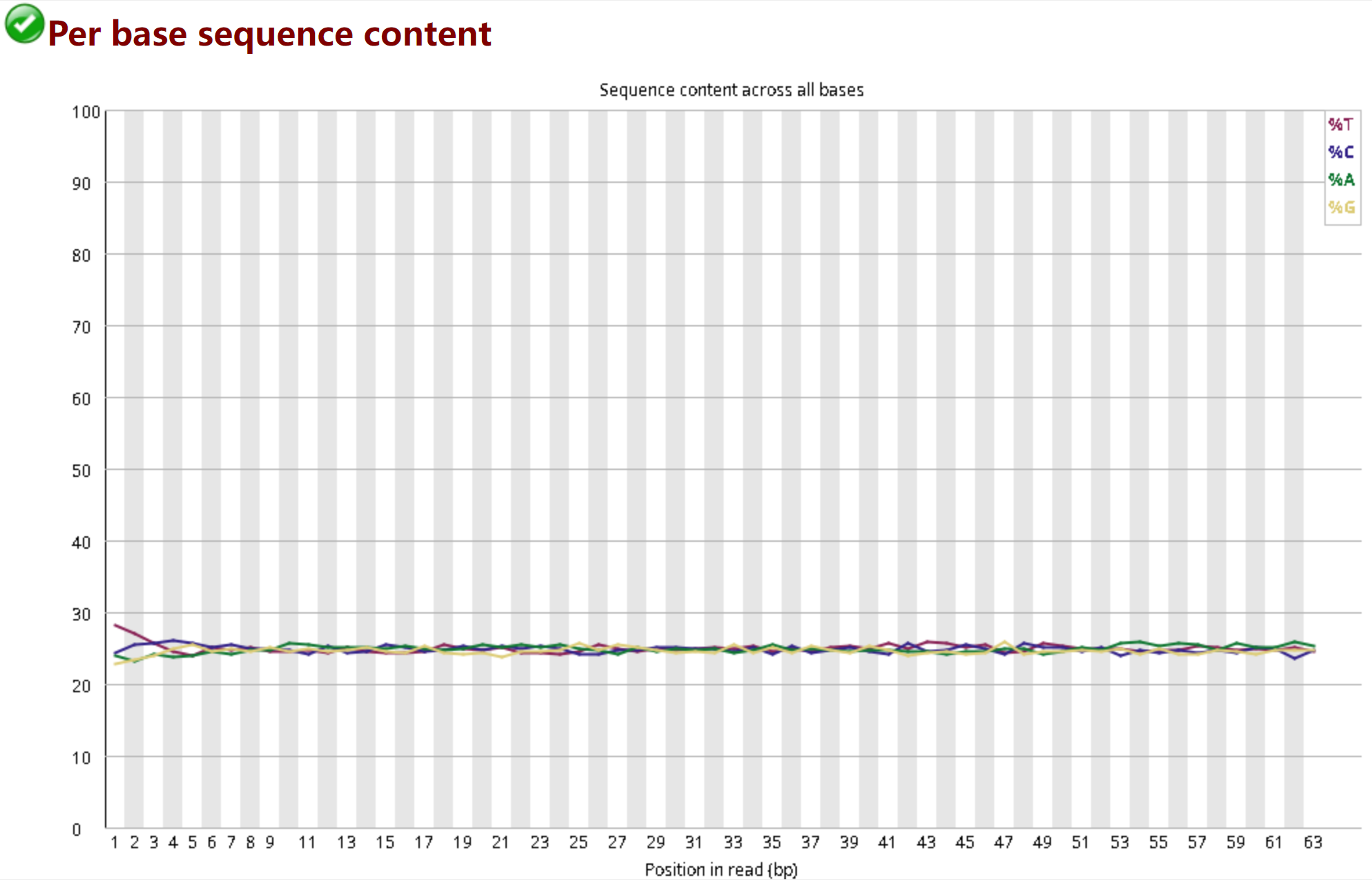
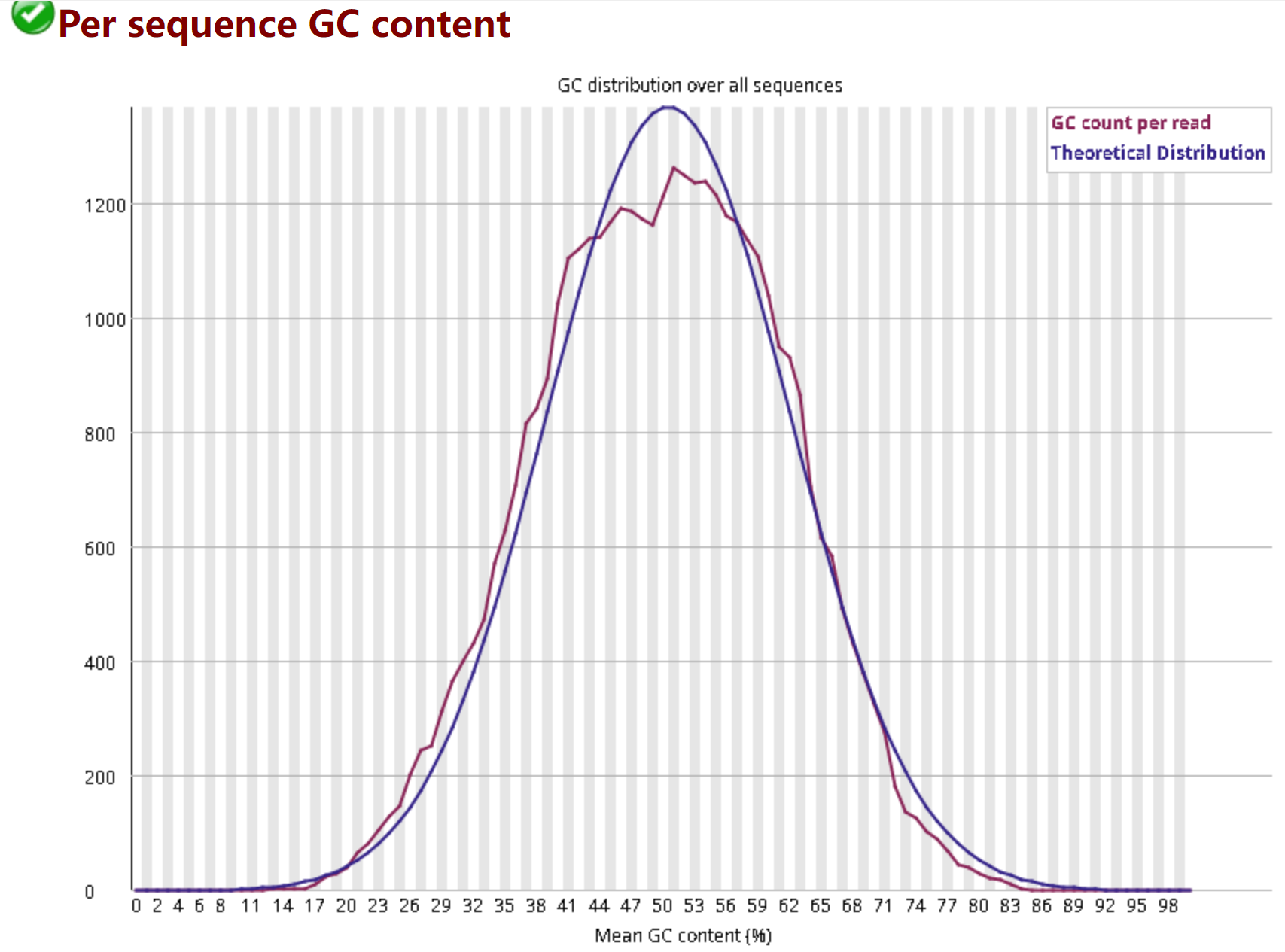
3.6 Per sequence GC content——GC含量分布图
横轴为平均GC含量 纵轴为每个GC含量对应的序列数量 蓝线为理论分布 红线为测量值,二者越接近越好
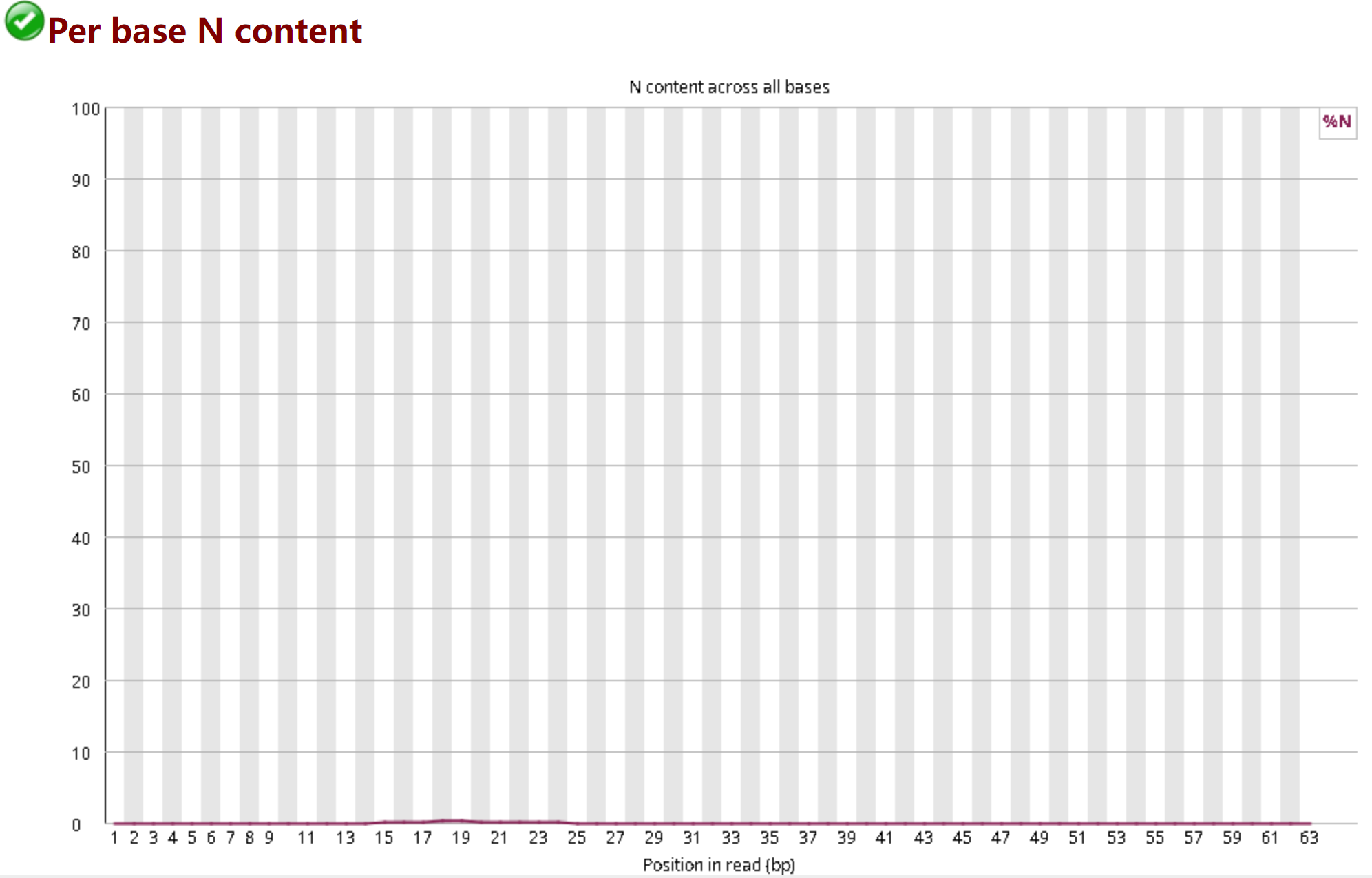
3.7 Per base N content——N含量分布图
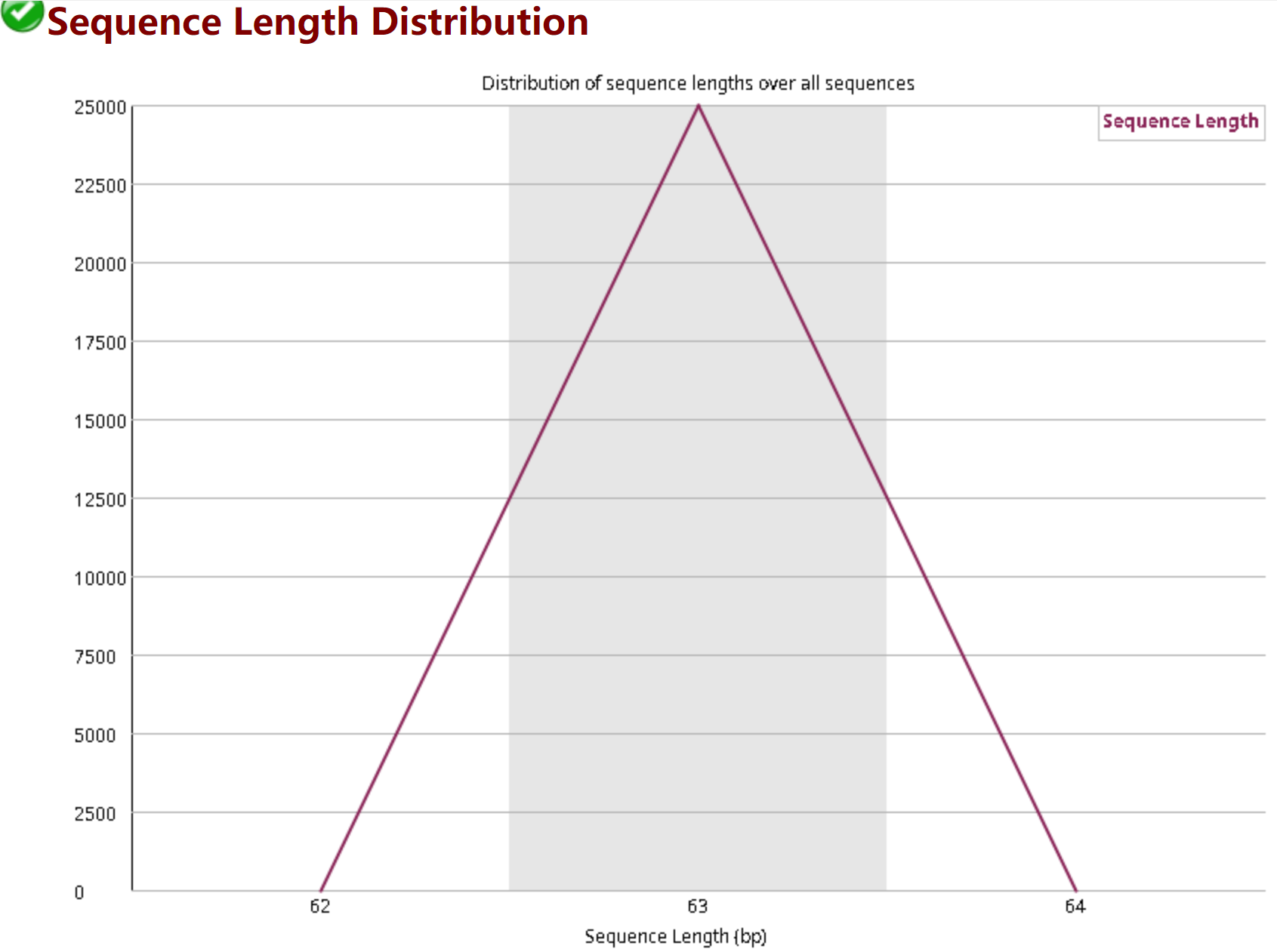
3.8 Sequence Length Distribution——序列长度分布图
对应的长度有多少条序列
3.9 Sequence Duplication Levels——序列重复性分布
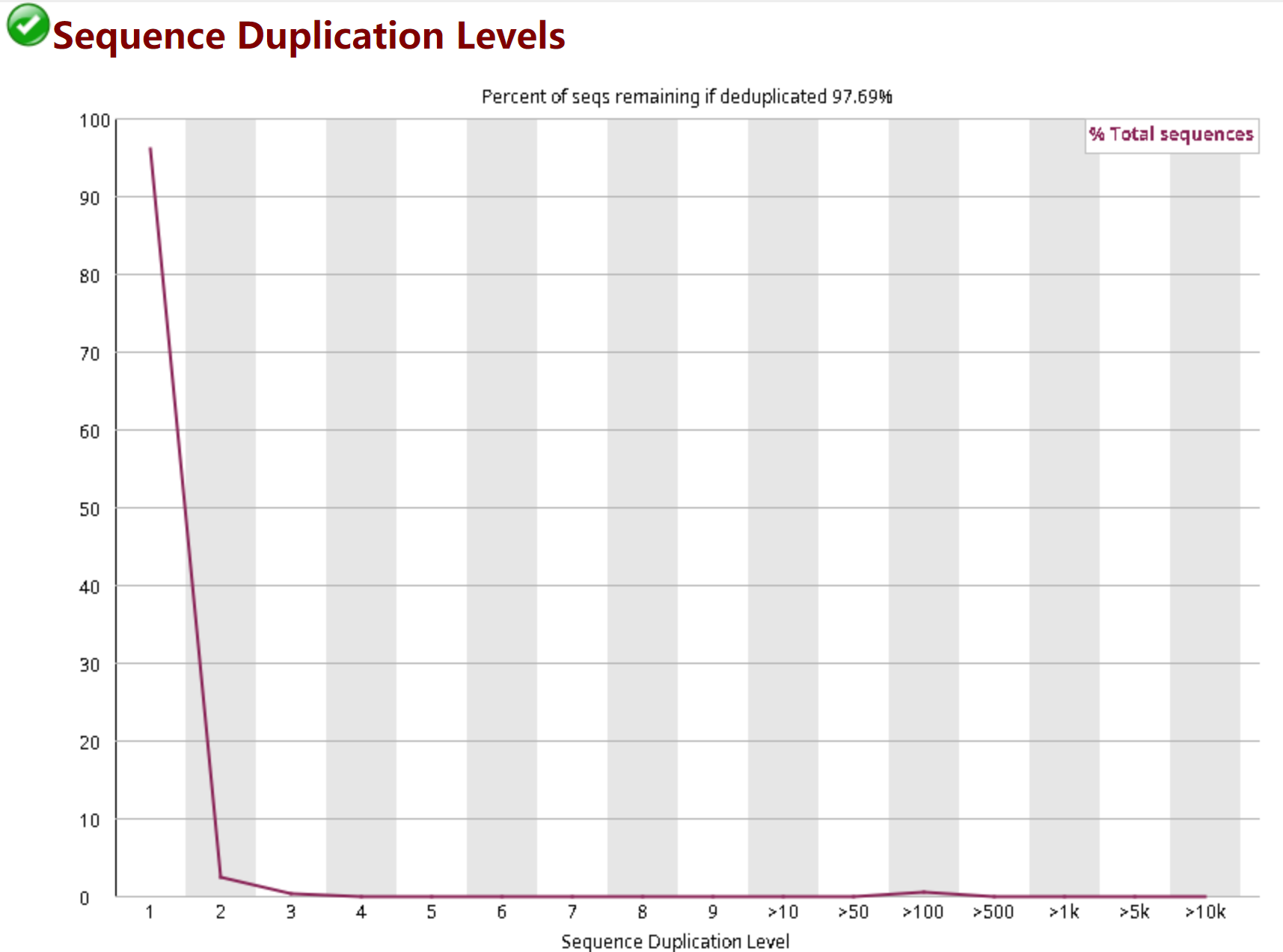 大多数序列只出现了一次
大多数序列只出现了一次
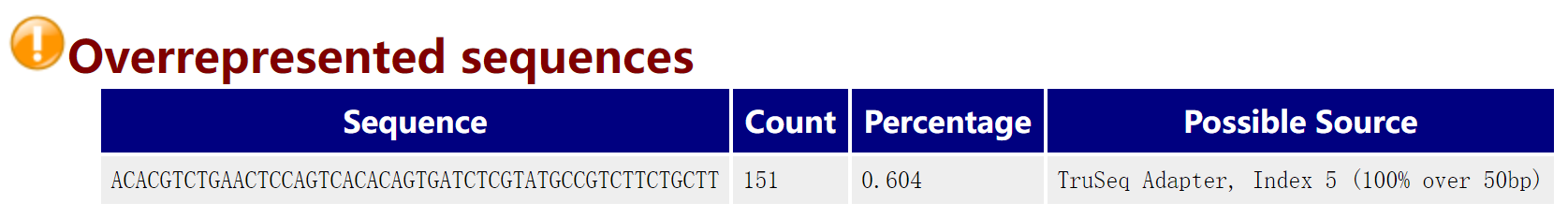
3.10 Overrepresented sequences
两种可能:基因本身有生物学意义/文库被污染
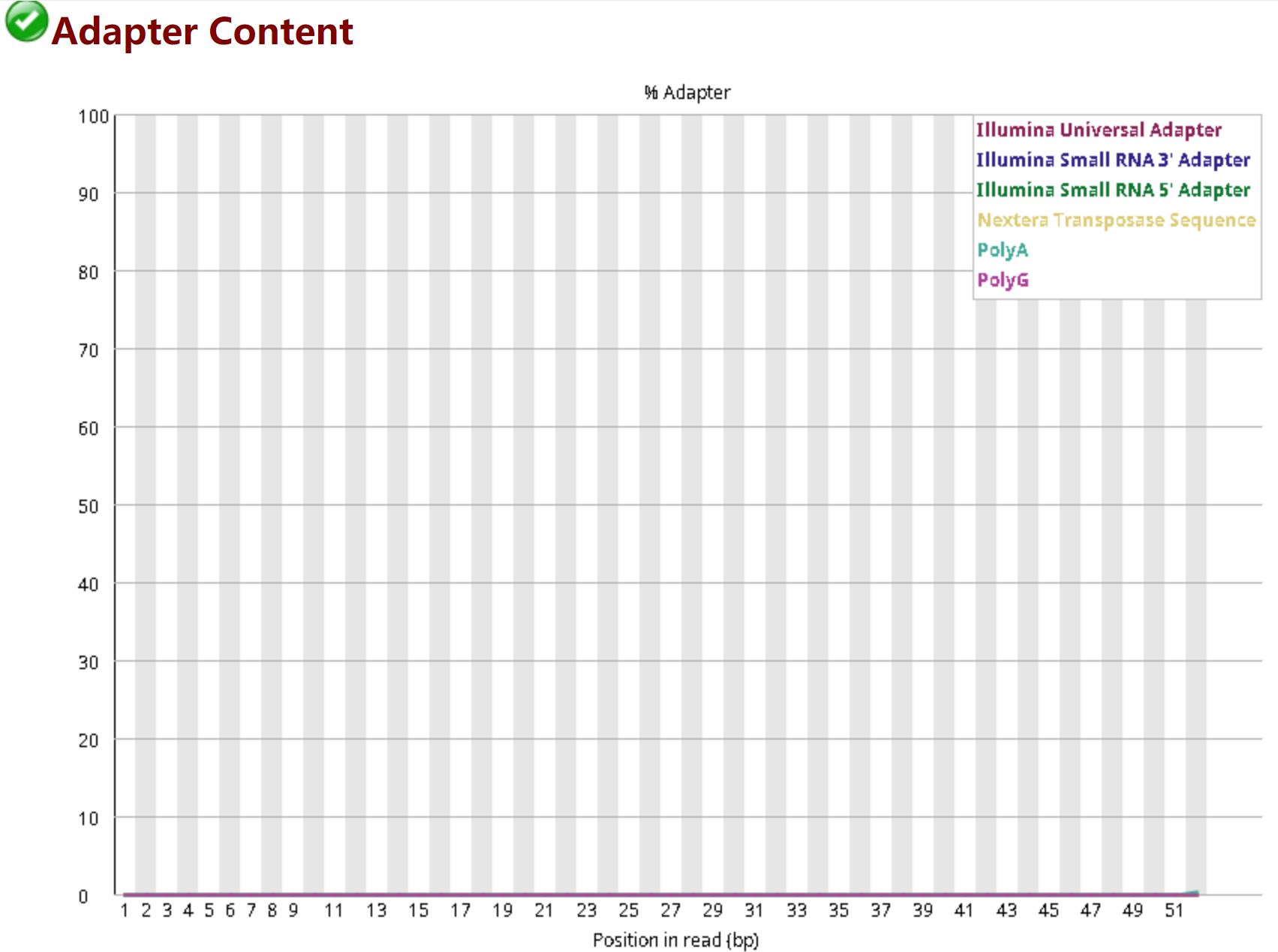
3.11 Adapter Content——接头含量
3.12 Kmer Content
Kmer:一段长度为k的DNA片段,是由测序reads剪切一部分得到的 怎么才能把所有样本的html报告都看一遍?
4 multiQC进行数据整合
在multiqc的下载过程中出现了一些问题,查了一下可能是python版本的问题,这里暂时略过这一部分……
本文由 mdnice 多平台发布





















 810
810

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








