软件简介
visNano 其主要是对下机数据的fastq文件以及经过比对后的bam文件进行可视化的summary,该软件发布在github上,具体用法可以自行研究,下面会展示绘制的可视化图效果。
安装
1. conda安装依赖
# conda 安装所需依赖
conda install numpy pandas tqdm matplotlib samtools
2. git clone获取软件包
windows 系统下可安装git软件,通过git bash下载该软件包
github地址:
https://github.com/renzilin/visNano
linux 系统可直接在指定目录下使用以下命令完成克隆
git clone https://github.com/renzilin/visNano.git
使用方法
# 查看软件帮助
python visNano.py -h
安装成功后运行以上命令会出现以下结果

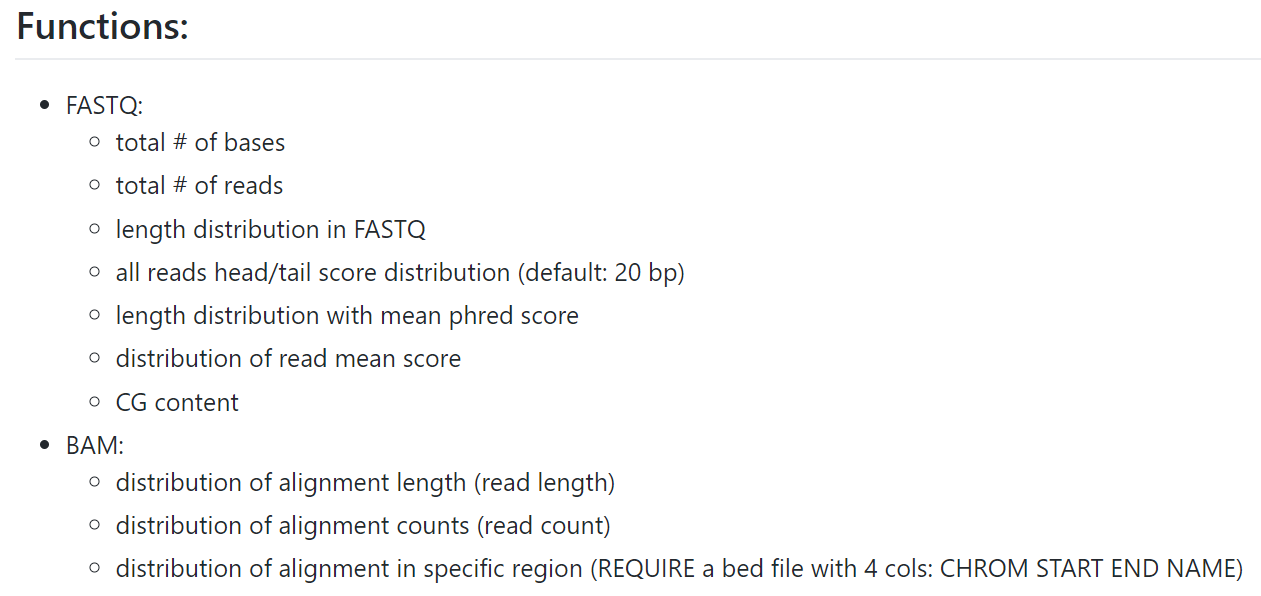
软件功能如下
使用示例
1. fq信息可视化
目前软件fq文件输入不支持.gz文件,可使用以下命令对.gz文件进行解压后进行可视化
gzip -d ./sample1.fq.gz # 解压当前目录下.gz文件
python /public/software/visNano/visNano.py fq \
--fq ./sample1.fq \ # 当前目录下fq文件
--s sample1
源代码解析如下:
 gzip
gzip
2. bam信息可视化
python /public/software/visNano/visNano.py bam \
--s sample1 \
--b ./sample1.bam \ # 当前目录下bam文件
--st /public/software/samtools
运行后软件需等待一段时间,开始运行后显示以下进度条
 运行结束后,在LOG-visnano文件下会出现一张.jpg可视化的图片。
运行结束后,在LOG-visnano文件下会出现一张.jpg可视化的图片。
其中–s输入为样本ID,–b输入为bam/sam文件路径,–st为samtools软件路径
samtools 软件路径查看可使用which:
which samtools
# 如果which samtools无法显示软件路径,可使用find命令查找
find / -name samtools
源代码解析如下:

3. bam+bed信息可视化
python /public/software/visNano/visNano.py bed \
--s sample1 \
--b ./sample1.bam \ # 当前目录下bam文件
--bd ./sample1.bed \ # 当前目录下bed文件
--st /public/software/samtools
可视化和上面用法类似,可以自行实践。
源代码解析如下:

Fastq文件信息可视化

BAM文件信息可视化
BAM+BED文件可视化




























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










