

大多数细菌和古菌都拥有多种抗病毒防御系统,具有“天然免疫系统”,可以抵御噬菌体、古菌病毒和可移动遗传元件的感染。因此,揭示原核生物的抗病毒天然免疫机制有助于理解生物抗病毒免疫的起源和进化。
在过去几年里,我们对防御系统多样性的理解大大增加,包括CRISPR-Cas系统、限制修饰系统(RM)和顿挫感染(Abi)等。2018年一篇里程碑式的论文从4.5万种细菌/古细菌基因组中发现了多种新型抗病毒免疫系统(Doron, S. et al., 2018, Science 359, eaar4120)看[1],揭开了大规模发现抗病毒系统的序幕。此后,又发现了多种抗病毒系统。研究者逐渐意识到,原核生物的免疫远比原先预计的复杂。目前已有多种软件可以预测原核生物抗病毒系统,并已经开发出能够在全基因组水平对所有已知抗病毒系统进行预测的计算工具,包括DefenseFinder、PADLOC等原核生物抗病毒系统预测软件。
凌恩生物
本次升级的个性化分析内容
依次使用上述两种方法进行
原核生物抗病毒系统预测
(1)基于DefenseFinder原核生物抗病毒系统预测
DefenseFinder软件能够对所有已知的原核抗病毒系统进行自动化注释,并利用该软件对21000多个微生物基因组完成图进行了注释,分析了抗病毒系统的分布特点。DefenseFinder是在一个名为MacSyFinder的程序基础上开发的,MacSyFinder程序专门用于大分子系统的探测。使用MacSyFinder时,需要对每类系统都单独构建一个模型。因此,为了构建DefenseFinder,作者对抗病毒机制进行了大量的文献检索,并据此为每类抗病毒系统构建模型。当前版本的DefenseFinder收录了2021年11月之前所有报道过的抗病毒系统,覆盖了60个抗病毒系统家族,一些系统可以分为不同的型和亚型,60个家族总计分为151(亚)型。评估显示,对每类系统,该工具都达到了很高的灵敏度(97.4% - 99.4%)和特异性(96.7% - 99.97%)。

图 DefenseFinder检测原理[2]
(2)基于PADLOC原核生物抗病毒系统预测
PADLOC是一种用于识别原核基因组中抗病毒防御系统的软件工具。PADLOC根据HMM和系统分类数据库筛选基因组,以基于序列同源性和遗传结构找到和注释防御系统。PADLOC允许用户分析全基因组、宏基因组重叠群、质粒、噬菌体和古菌病毒。该网络服务器包括检测到的防御系统类型增加了5倍多(自首次发布以来),并扩展了能够检测CRISPR阵列和逆转录病毒ncRNA的功能。PADLOC网络服务器还存储了一个预先计算的防御系统数据库,该数据库包含230000个RefSeq基因组。
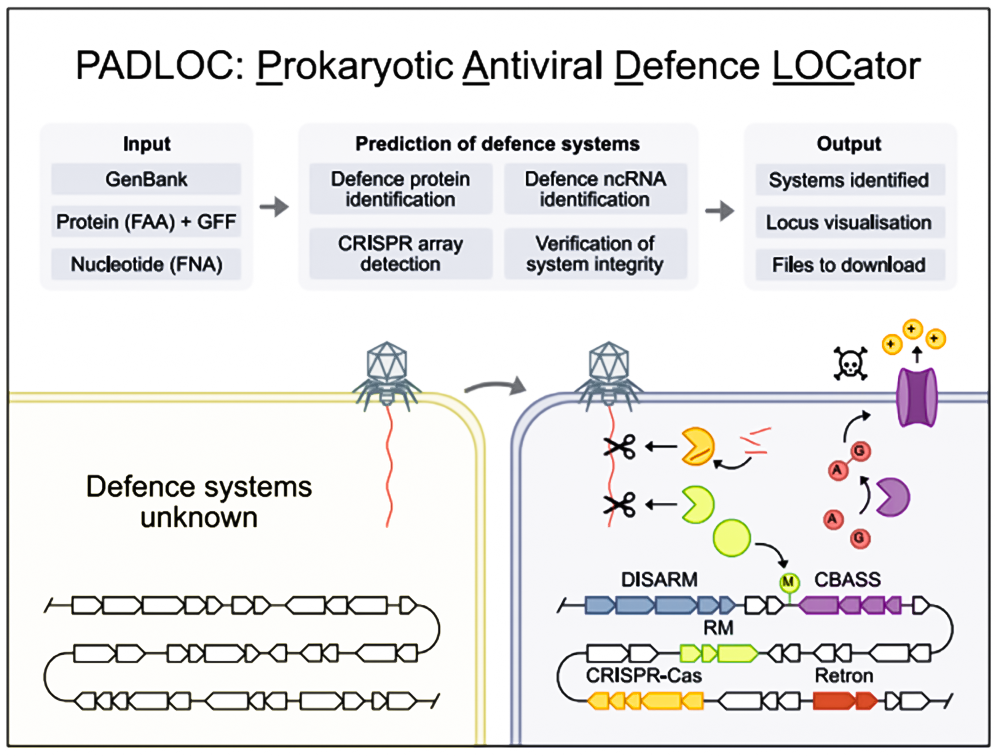
图 PADLOC工作原理[3]
研究示例

-
题目:深海冷渗沉积物中的病毒具有多种生存机制并在物种内具有基因保守性[4]
-
期刊:ISME
-
影响因子:10.302
-
DOI:10.1038/s41396-023-01491-0
深海冷泉沉积物已被发现含有新的、丰富的、多样的细菌和古菌病毒。然而,冷泉病毒的多样性和分布模式、病毒-微生物相互作用、对环境的适应机制以及病毒的遗传多样性等方面的研究仍相对较少。
为理解冷泉病毒的适应性生存机制和遗传微多样性,研究者从南海海马冷泉地区的16个沉积物岩心样品中提取了病毒基因组。研究共鉴定出338个种水平上的高质量病毒基因组,预测发现这些病毒可侵染36个细菌和古菌门类。在获得的487个原核微生物基因组中共识别到43种抗病毒系统(antiviral system)家族(图1),多样化的抗病毒防御系统揭示了冷泉微生物-病毒之间存在频繁的相互作用。
为应对宿主微生物的抗病毒系统,作者发现冷泉病毒具有多种环境适应策略,包括抗防御系统(counter-defense system,如anti-CRISPR蛋白、甲基转移酶和抗毒素蛋白)、逆转录酶(reverse transcriptase)、辅助代谢基因(auxiliary metabolic gene)和可变遗传编码现象(alternative genetic code)等(图2)。
作者进一步发现,在进化模式上,冷泉病毒种群具有极低的核苷酸多样性,绝大多数冷泉病毒基因受到强烈的纯化选择,并随冷泉发育阶段不同而变化。微生物宿主、沉积物深度和冷泉地质等多种因素,可能影响了冷泉病毒的进化模式(图3)。
总体上,这项工作首次讨论了海底生物圈中病毒的环境适应机制和进化模式,将为未来针对极端环境病毒进化和病毒-宿主系统的研究提供参考。

图1 冷泉细菌和古菌基因组中发现多样化抗病毒系统

图2 冷泉病毒适应环境的多样化策略

图3 冷泉病毒基因组范围的进化指标
参考文献
[1]Doron S, Melamed S, Ofir G, et al. Systematic discovery of antiphage defense systems in the microbial pangenome. Science. 2018 Mar 2;359(6379):eaar4120. doi: 10.1126/science.aar4120. Epub 2018 Jan 25.
[2]Tesson F, Hervé A, Mordret E, et al. Systematic and quantitative view of the antiviral arsenal of prokaryotes. Nat Commun. 2022 May 10;13(1):2561. doi: 10.1038/s41467-022-30269-9.
[3]Payne LJ, Meaden S, Mestre MR, et al. PADLOC: a web server for the identification of antiviral defence systems in microbial genomes. Nucleic Acids Res. 2022 Jul 5;50(W1):W541-W550. doi: 10.1093/nar/gkac400.
[4]Peng Y, Lu Z, Pan D, et al. Viruses in deep-sea cold seep sediments harbor diverse survival mechanisms and remain genetically conserved within species. ISME J. 2023 Oct;17(10):1774-1784. doi: 10.1038/s41396-023-01491-0. Epub 2023 Aug 12.







 研究人员在深海冷泉沉积物中发现了丰富的病毒及复杂的抗病毒防御系统。DefenseFinder和PADLOC两种软件被用来预测原核生物的抗病毒系统,揭示了冷泉病毒的适应策略,如抗CRISPR蛋白和可变遗传编码。这项研究为理解极端环境病毒进化提供了新视角。
研究人员在深海冷泉沉积物中发现了丰富的病毒及复杂的抗病毒防御系统。DefenseFinder和PADLOC两种软件被用来预测原核生物的抗病毒系统,揭示了冷泉病毒的适应策略,如抗CRISPR蛋白和可变遗传编码。这项研究为理解极端环境病毒进化提供了新视角。


















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










