抗菌肽(Antimicrobial Peptides, AMPs)是一类广泛存在于自然界生物体中的小分子多肽。作为下一代抗菌药物,AMP具有广谱抗微生物活性,被誉为“天然抗生素”,针对性强,且其耐药性演变速度很低。
AMP独特机制(如破坏微生物细胞膜)可有效对抗细菌、真菌、病毒甚至耐药菌,也可参与微生物互作。在医学中可用于抗感染治疗、抗肿瘤免疫、抗病毒制剂、组织修复等领域,在环境中可用于生物防治、工业防腐、污水处理等方面。因此,抗菌肽研究具有非常重要的意义,有望替代抗生素成为一种潜在的治疗方法。
那么如何通过宏基因组测序手段来研究自然界生物体中的抗菌肽呢?先来看看几篇经典研究~
动物肠道微生物中抗菌肽研究:
2024年发表于《Cell》的文章“Mining human microbiomes reveals an untapped source of peptide antibiotics”,从1,773个健康个体的肠道样本进行宏基因组测序,共识别出了323种抗菌肽,这些抗菌肽是由小开放阅读框(smORFs)编码的。然后合成了78种肽并进行了体外抗菌活性筛选,发现其中70.5%的肽显示出对病原体或共生菌的活性。

图 从smORFs中发现SEPs(smORF-encoded peptides)的计算实验平台示意图[1]
环境微生物中抗菌肽研究:
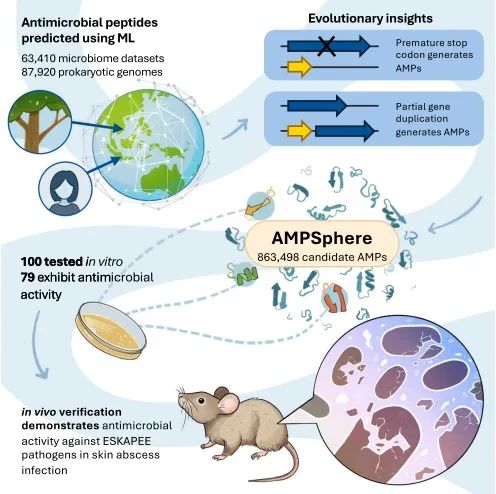
2024年发表于《Cell》的另一篇文章“Discovery of antimicrobial peptides in the global microbiome with machine learning”,收集了全球来自75个不同栖息地的63,410个样本公共宏基因组数据+87,920个高质量的微生物基因组数据,构建了一个全球微生物小开放阅读框(smORFs)目录。该文章提出了一个利用机器学习预测全球微生物组中抗菌肽(AMPs)的新方法。该方法识别了近一百万个抗菌肽序列,并验证了关注功能的抗菌肽(AMPs)的抗菌活性,为抗生素发现提供了一个开放的资源。
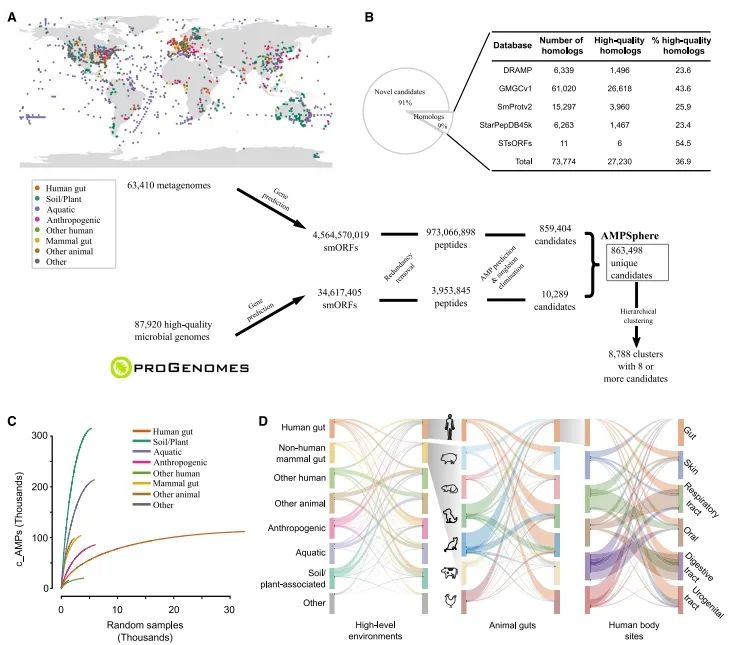
图 AMPSphere由来自数千个宏基因组和微生物基因组的非冗余cAMPs组成[2]
此外,2024年发表于《PNAS》的文章“Metagenomic study of lake microbial mats reveals protease-inhibiting antiviral peptides from a core microbiome member”,以印度奇利卡湖藻-菌共生这一复杂且相互作用丰富的生态系统为对象,以宏基因组数据重建了超过1,300个环境基因组(含2,200余个生物合成基因簇,BGCs),并鉴定出蛋白酶抑制型抗病毒肽(protease-inhibiting antiviral peptides),这些肽属于核糖体合成-翻译后修饰肽(RiPPs)的graspetide和spliceotide家族,来源于核心微生物组成员Rheinheimera属细菌。其功能聚焦于抑制病毒复制(如通过阻断病毒蛋白酶活性)。研究突出了核糖体合成-翻译后修饰肽(RiPPs)作为抗病毒药物开发未被充分挖掘的巨大资源库的价值。
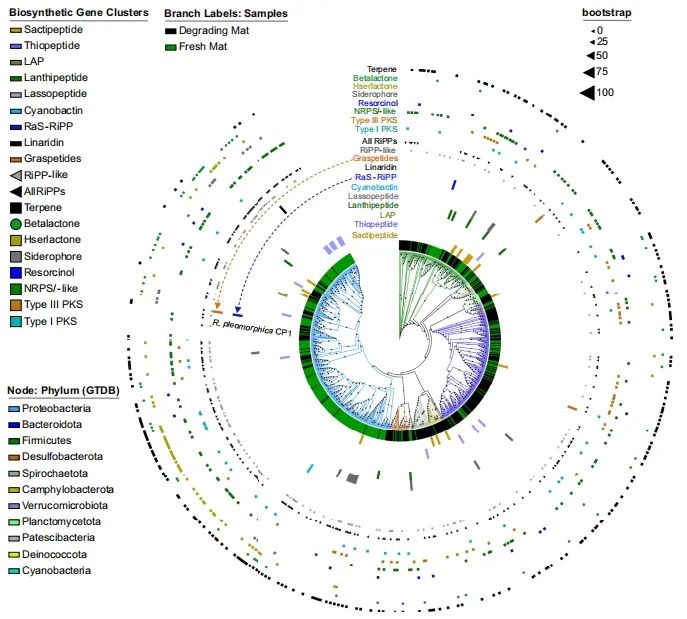
图 MAGs和检测到的biosynthetic gene clusters(BGC)类别的系统发育分布[3]
凌恩生物宏基因组抗菌肽研究策略
凌恩生物根据Cell[2]的方法,开发出了基于宏基因组的高级分析——深度挖掘不同生境中AMP种类和功能!该分析应用AmPEP、APIN、ampri和amp-scanner-v2等四种近期发表的主流方法进行AMPs的鉴定,多个软件同时分析AMP相关peptide。给到每种软件预测的AMP相关gene ID和peptide序列,并给到AMP相关肽的软件得分。
已有宏基因组数据、宏基因组binning数据,想要更特色的分析内容?发更有特色的文章?想从动物肠道或环境微生物中发现更多功能性抗菌肽?
快来联系我们吧!凌恩生物将为您提供“宏基因组抗菌肽深度挖掘”一站式分析体系,助力高效发现功能肽!
参考文献
[1]Torres MDT, et al. Mining human microbiomes reveals an untapped source of peptide antibiotics. Cell. 2024 .
[2]Santos-Júnior CD, et al. Discovery of antimicrobial peptides in the global microbiome with machine learning. Cell. 2024.
[3]Padhi C, et al. Metagenomic study of lake microbial mats reveals protease-inhibiting antiviral peptides from a core microbiome member. Proc Natl Acad Sci U S A. 2024.
原文链接:
https://doi.org/10.1016/j.cell.2024.07.027
https://doi.org/10.1073/pnas.2409026121
https://doi.org/10.1101/2023.08.31.555711
























 370
370

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










