近年来,随着测序技术的飞速发展,三代宏基因组研究成为微生物学领域的热点方向,受到国科金的重点关注与支持。借助国科金的有力支撑,科研人员在三代宏基因组研究方面不断取得新突破,从解析复杂微生物群落的组成与功能,到挖掘未培养微生物的遗传信息,为深入理解微生物世界的生态、进化和应用等方面开拓了新视野,带来了前所未有的机遇与挑战。
01.三代宏基因组
既往研究中,利用宏基因组binning有助于获得不可培养微生物的全基因组序列,但二代读长的限制仍然存在着组装Contig太短、组装完整性差、基因不完整、基因簇破裂、污染率高等问题。
三代宏基因组测序是借助三代测序技术平台,针对环境中微生物群落的多样性及其功能展开研究的技术方法。与二代宏基因组测序相比,三代宏基因组测序具备显著优势。它能够挖掘出更多的物种信息,像低丰度物种以及尚未成功培养的物种等。同时,通过三代宏基因组测序,可以为复杂微生物群体样本中的单菌组装提供又长又准的序列,大幅提升组装效果,打造宏基因组完成图Complete MAGs新概念,并可为复杂微生物组的多方向研究提供高效解决途径。
02.三代宏基因组技术优势
无论是针对环境样本还是医口样本,已有大量相关研究对二代和三代组装结果进行了比对,三代宏基因组都表现出非常显著的优势。
1. 测序结果和组装结果比对
针对14个海水样本[1]的测序结果表明,每个样本的三代 CCS测定reads的数量为6,807~1,907,840个,相应的Illumina reads数量为2,879,847~264,220,592。经比较,相比于二代或混合组装中的contigs,仅三代产生的组装中的最长contigs更长。
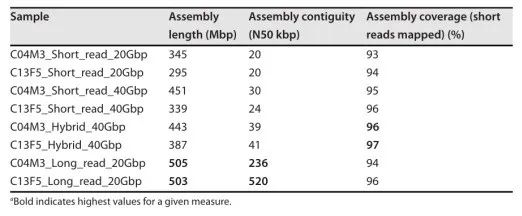
在小鼠粪便样本宏基因组研究[2]中,二代组装的N50值约为20kbp,混合组装的N50值约为~40kbp,三代组装的N50值在235-519kbp之间,三代数据组装的连续性比二代和混合组装高9-18倍。
表 小鼠粪便样本宏基因组组装数据统计
2. 注释结果比较
基于海水样本的α多样性分析表明,Illumina和混合组装的宏基因组的数值范围比三代宏基因组要小很多,Shannon多样性值差异也很大。此外,基于三代测序和二代测序的样本的β多样性结果也呈现出显著差异。

图 未组装的二代和三代和从三代、二代和混合组装中提取的开放阅读框(ORF)的α和β多样性分析。
3. 组装得到的MAG质量和数量比对
基于三种组装策略从小鼠粪便样本中重建MAG,三代组装策略恢复的MAGs的平均contig数量最小,且MAGs的N50普遍较大,混合策略次之。三代宏基因组测序在MAG连续性方面表现最佳,此外,三代宏基因组恢复的环化MAGs数量最多。
使用两种不同的binning程序比较了二代、三代和混合组装从海水样本中恢复的MAGs 的数量和质量评分。在所有三代和混合组装产生的15个高质量的MAGs中,从相应二代宏基因组中匹配到的MAG缺少(在属水平上定义)5个。
表 海水样本中来自所有三代和混合组装的15个高质量MAG的来源、质量和分类结果。


图 从小鼠粪便样本中恢复的MAG质量和数量评估。
4. 质粒、病毒和组装的环状元件
对小鼠粪便样本的研究表明,三代宏基因组在质粒研究方面具有优势,能恢复更大的质粒序列,且环形序列更易分类。二代宏基因组和三代宏基因组在恢复环状病毒基因组方面表现相当,若研究重点是病毒,添加三代测序数据可能不会显著改善结果;若关注质粒,三代测序数据可恢复二代测序数据集中缺失的大质粒序列。

图 从每个数据集类型注释为质粒、病毒等环状contig的统计。
总的来说,二代宏基因组的特点在于更倾向于恢复更多物种的宏基因组组装基因组(MAGs)。与之相比,三代宏基因组在恢复低丰度物种的MAGs方面表现欠佳。不过,三代宏基因组凭借其读长的显著优势,在组装基因组总长度、组装连续性以及MAGs的连续性这些方面,展现出了最优的性能。而二+ 三混合组装策略,在某些特定样本的处理中,也呈现出了一定的优势。所以,在实际操作时,为了实现最佳的组装效果,需要根据具体样本的实际情况,综合考量并选择合适的测序和组装策略。
03.客户文章案例
利用三代宏基因组测序改进藏盐湖沉积物中宏基因组组装基因组和病毒基因组组装

发表期刊:Microbiology Spectrum
影响因子:9.043
样本类型:表层沉积物
研究策略:三代宏基因组+宏病毒组
研究背景:随着高通量测序技术的发展和成本的降低,一些新的宏基因组组装基因组(MAGs)和病毒基因组,通过二代测序组装很容易得到。但是由于NGS短读长的限制,组装得到的MAG和病毒基因组经常存在基因组组装不连续性和关键功能元件的缺陷问题。
主要结果:
1、本研究从HiFi reads直接获得207个全长非冗余16S rRNA基因序列和19个全长非冗余18S rRNA基因,大大超过了NGS技术的检索性能。
2、仅使用HiFi reads重建了两个MAGs和29个具有环状基因组的病毒,这表明“HiFi”方法在组装高质量微生物基因组方面的巨大潜能。
3、评估了不同组装策略在宏基因组组装和病毒组装中的性能。28.9%的NGS reads可以分配到已知的分类等级,而HiFi数据的reads分配率为90.8%,大约是NGS的三倍。

图 基于HiFi reads的分类结果。(A) HiFi和NGS reads具有(已知)/不具有(未知)分类信息的百分比。(B和C)基于19个全长18S-OTUs (B)和207个全长16S-OTUs(C)的系统进化树。

图 CCL湖沉积物中代表性MAGs的系统发育。(A)基于102个代表性MAGs的PhyloPhlAn 3.0标记基因的系统发育树。3种组装策略的代表性MAGs (B)的数量和16S rRNA拷贝数分布(C)。
通过多组学分析,揭示了未培养的厌氧氨氧化群落细菌的生物合成潜力

发表期刊:Bioresource Technology
影响因子:9.7
样本类型:厌氧氨氧化污泥样本
研究策略:HiFi宏基因组+宏蛋白组
研究背景:微生物次生代谢物(SMs)及其衍生物已广泛应用于医药、农业和能源领域。对可再生能源日益增长的需求,以及抗生素耐药性、癌症和杀虫剂带来的挑战,强调了寻找新的SMs的关键。厌氧氨氧化系统含有许多未培养或未充分开发的细菌,代表了发现新的SMs的潜在资源。
主要结果:
1、通过HiFi宏基因组测序,从厌氧氨氧化微生物组中获得了1,040个生物合成基因簇(BGCs),其中58%为完整的,显示出丰富的多样性。
2、未开发的细菌门(Chloroflexota和Planctomycetota)和Proteobacteria含有大量的BGCs,显示出巨大的生物合成潜力。
3、宏蛋白质组学鉴定出128个活跃的BGCs,特别是在Planctomycetota门中。

图 中高质量MAGs的质量评估结果、系统发育分类和BGC数量。

图 Planctomycetota门中发现的完整梯形烷(Ladderane)结构的BGCs及其表达。
基于宏基因组研究,提出厌氧氨氧化群落中未培养细菌的功能分化和适应性进化新见解

发表期刊:Science of the Total Environment
影响因子:9.8
样本类型:厌氧氨氧化活性污泥样本
研究策略:HiFi宏基因组
研究背景:厌氧氨氧化(anammox)细菌在全球氮循环和废水氮去除中起着至关重要的作用,但它们与其他多种微生物具有共生关系。厌氧氨氧化菌群落中未培养细菌的功能分化和适应性进化尚不清楚。
主要结果:
1、基于HiFi宏基因组获得了30个准确和完整的宏基因组组装基因组(cMAGs)。
2、在该厌氧菌群落中发现了1个隶属于Candidatus Jettenia候选菌的新厌氧菌种和3个隶属于新科的新厌氧菌种。
3、在这些cMAGs中鉴定出63个可移动遗传元件(MGEs)和50个假定的水平基因转移(HGT)事件。
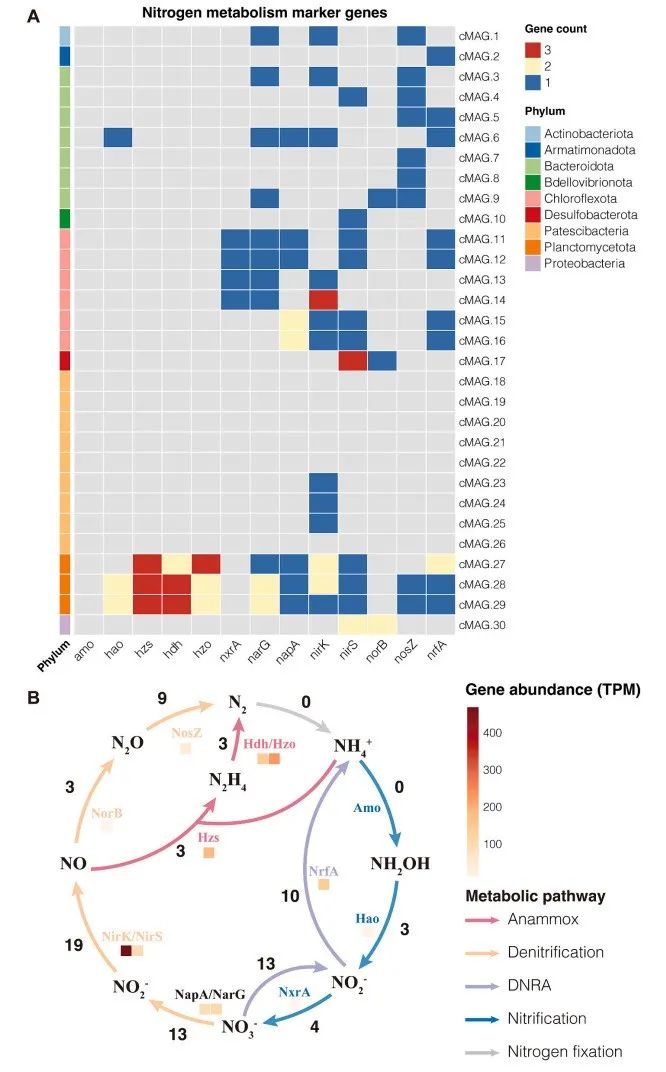
图 30个cMAGs的氮代谢潜力。(A)氮代谢功能基因。(B)提出了厌氧氨氧化群落中的氮转化途径。

图 cMAG.29中预测28个含tnpA的基因簇。

参考文献
1、Long-Read Sequencing Improves Recovery of Picoeukaryotic Genomes and Zooplankton Marker Genes from Marine Metagenomes. mSystems. 2022.
2、A comparison of short-read, HiFi long-read, and hybrid strategies for genome-resolved metagenomics. Microbiology Spectrum, 2024.
3、Improved Assembly of Metagenome-Assembled Genomes and Viruses in Tibetan Saline Lake Sediment by HiFi Metagenomic Sequencing. Microbiology Spectrum, 2022.
4、Biosynthetic potential of uncultured anammox community bacteria revealed through multi-omics analysis. Bioresource Technology, 2024.
5、New insights into functional divergence and adaptive evolution of uncultured bacteria in anammox community by complete genome-centric analysis. Science of the Total Environment, 2024.

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










