介绍FsFAST的预处理
根据官网的英文进行翻译,并在使用过程中所犯的错误进行解决的过程
介绍Tutorial Data 下载以及安装
安装
安装Tutorial Datasets 从教程数据,创建文件tutorial_data在自己的工作路径中。
curl https://surfer.nmr.mgh.harvard.edu/pub/data/tutorial_data.tar.gz -o tutorial_data.tar.gz
tar -xzvf tutorial_data.tar.gz
rm tutorial_data.tar.gz
配置
为了能够运行tutuorials用于需要设置环境变量TUTORIAL_DATA设置为 tutorial_data的根目录,文件夹中必须包含提取出来的数据(目录有’buckner_data’,‘long-tutorial’,'fsfast-functional’等),由于教程中频繁使用环境变量TUTORIAL_DATA,需要对他进行适当的设置才能正常运行
export TUTORIAL_DATA=/path/to/your/tutorial/dir
ls $TUTORIAL_DATA
buckner_data fsfast-functional
diffusion_recons fsfast-tutorial.subjects
diffusion_tutorial long-tutorial
如果出现类似上面的输出,则可以进行教程的学习了
FSFAST 预处理介绍
一旦数据存放在适当的目录结构和命名约定中,可以对数据执行预处理步骤,预处理包括:
a) Template Creation
b) Brain Mask Creation
c) Registration with FreeSurfer Anatomical
d) Motion Correction
e) Slice Timing Correction
f) Spatial Normalization
g) Masking
h) Spatial Smoothing
在FSFAST中,假设每个数据集将在三个归一化空间中进行分析:
- Left cortical Hemisphere 左半球大脑皮层
- Right cortical Hemisphere 右半球大脑皮层
- Subcortical Structures 皮层下结构(Volumed-based)
判断多大的平滑和是否需要进行Slice Timing Correction。在这个分析中使用FWHM=5mm对数据进行平滑处理,然后需要进行Slice Timing Correction。 Slice Timing对于特殊的数据集是升序的(“Ascending”),即first slice是最先得到的,second slice是第二个得到的。
执行一下命令,对数据执行预处理部分,首先切换到目标目录中.
export SUBJECTS_DIR=$TUTORIAL_DATA/fsfast-tutorial.subjects
cd $TUTORIAL_DATA/fsfast-functional
然后执行preproc-sess具体命令如下
preproc-sess -s sess01 -fsd bold -stc up -surface fsaverage lhrh -mni305 -fwhm 5 -per-run
这条命令有几个参数:
- -s sess01:进行预处理的session是哪个
- -fsd bold:表示functional subdirectory(FSD)
- -stc up:执行slice-timing correction的顺序是 ascending(‘up’)
- -surface fsaverage lhrh:数据应该被采样到 "fsaverage"受试者的左右半球
- -mni305:将数据采样到 mni305 volume(2 mm 各向同性体素大小)
- -fwhm 5:在重采样之后使用 5mm FWHM进行平滑处理
- -per-run:分别配准每个run
上面的了命令做的步骤比较多,如果是包含多个被试时,将花费比较长的时间执行。
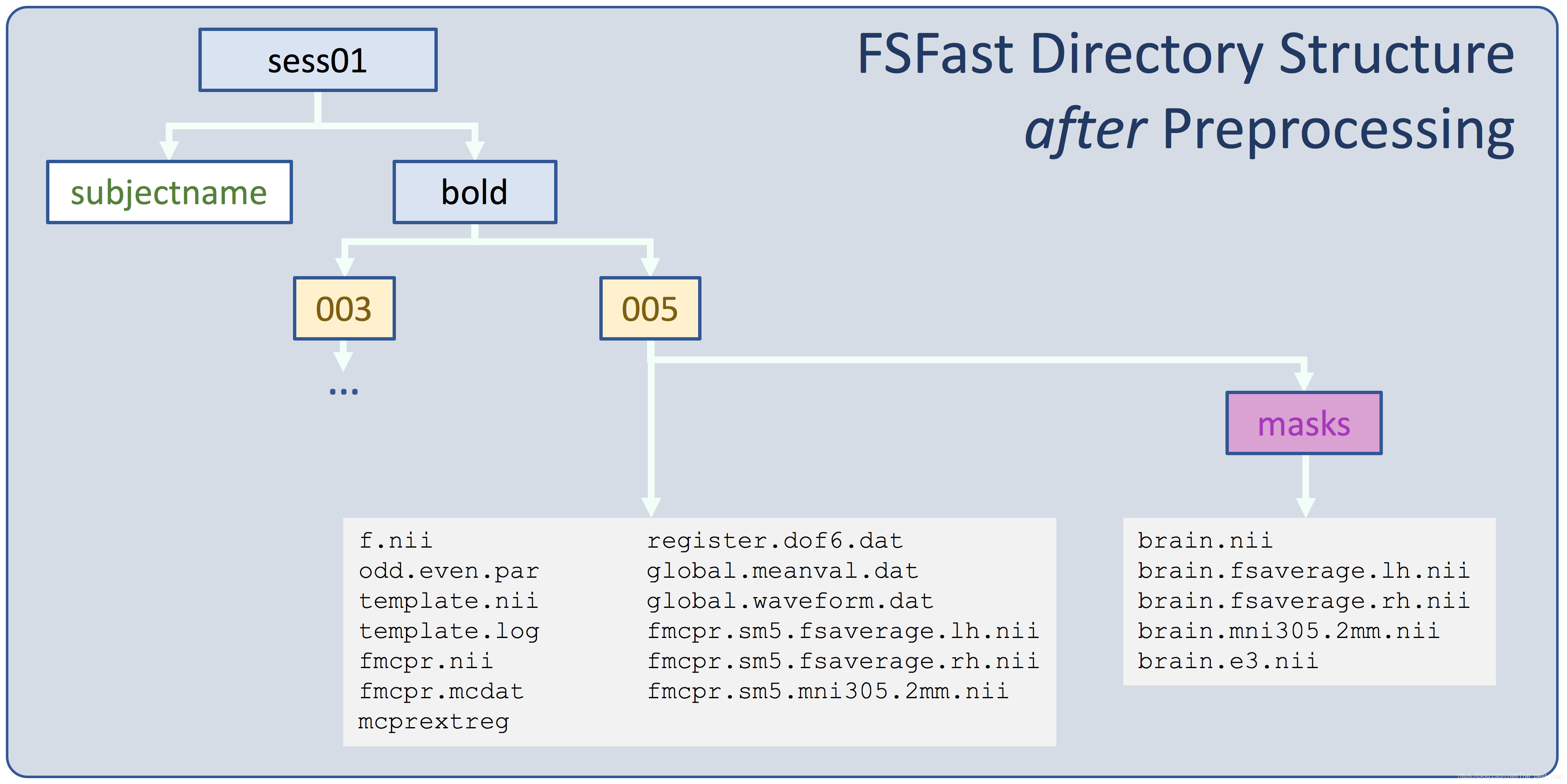
产看其中一个运行目录的内容
ls $TUTORIAL_DATA/fsfast-functional/sess01/bold/001
你将看到呈现出很多的文件,但是有三个比较重要的文件:
- fmcpr.up.sm5.fsaverage.lh.nii.gz -左半球的 fsaverage
- fmcpr.up.sm5.fsaverage.rh.nii.gz -右半球的 fsaverage
- fmcpr.sm5.mni305.2mm.nii.gz - volume的fsaverage - 用于皮层下分析
这些是时间序列的数据,并且他们的名称表明他们执行的过程
- fmcpr - 每个run的头动校正
- up - 使用升序的时间层校正(slice-timing correction)
- sm5 - 使用 5mm FWHM进行平滑处理。
- fsaverage.lh - 左侧半球的表面进行采样
- fsaverage.rh - 右侧半球的表面进行采样
- mni305.2mm - 在2mm各向同性的 fsaverage (MNI305)volume中采样
想要了解更多,看Preprocessing Details
Quality Assurance 质量保证
Functional-Anatomical Cross-modal Registration
功能-结构交叉模态配准
通过以下命令获得配准质量的总结
tkregister-sess -s sess01 -s sess02 -s sess03 -fsd bold -per-run -bbr-sum
输出为每一个session的每一个run的值
sess01 001 0.5740
sess01 002 0.5776
sess01 003 0.5813
sess01 004 0.5740
sess02 001 0.5159
sess03 001 0.6021
- 第一列表示Session的ID
- 第二列表示每个Session中的每次Run
- 第三列表示quality assurance (QA)值。他是从0到1的值,0表示完美,1表示糟糕。
实际值取决于您如何获取数据。一般来说,任何超过0.8的值都表示可能出了问题,比如:
- 功能和结构像的个体不匹配
- 功能像的左右颠倒
- 初始化失败(部分FOV(Field-of-View)时有发生)
使用以下命令查看registrations的图像
tkregister-sess -s sess02 -fsd bold -per-run
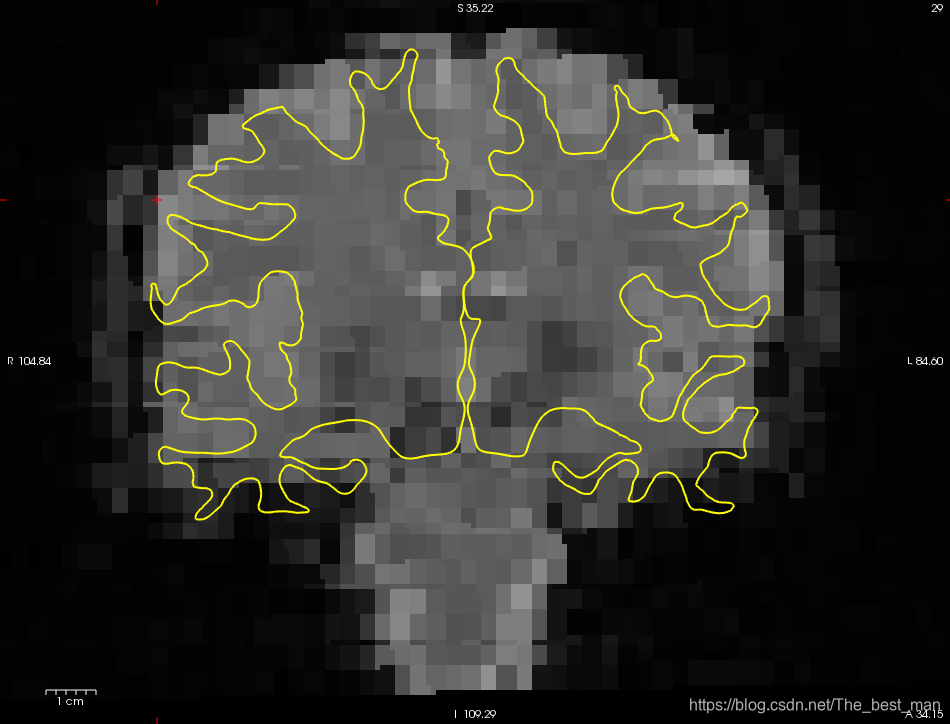
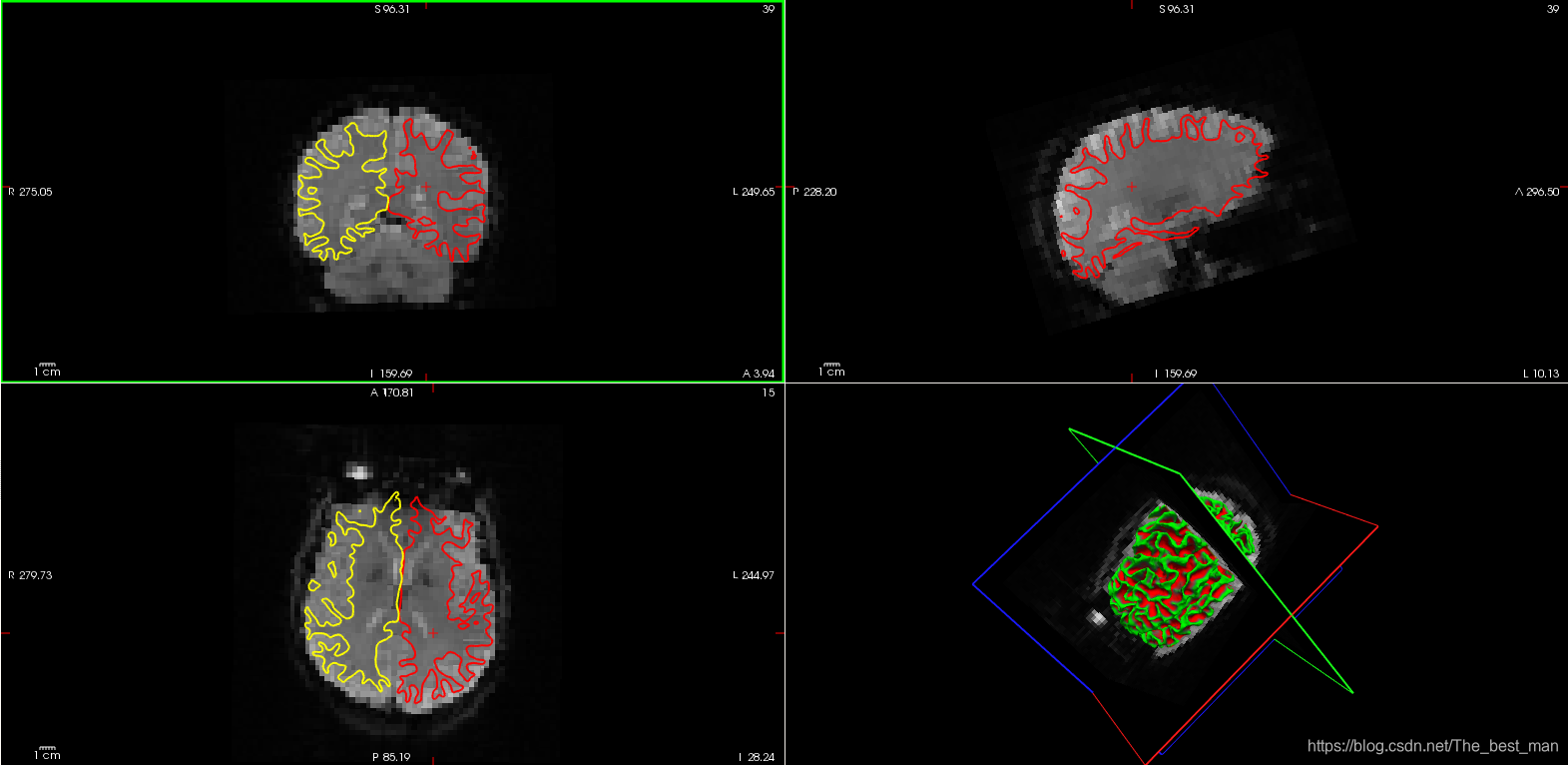
- 这将在Freeview中每次Run中显示registration。
- 现在只需要看一下配准的结果并确定他看起来结果不错
- 退出Freeview
如果你想知道更多的关于registration使用tkregister和Freeview,看多模态教程
Preprocessing Details
您不需要执行下面的步骤来理解本教程的剩余部分。这部分可以使你深入理解FS-FAST预处理的内部工作原理.
回到其中一个运行的目录,看看preprocess产生了什么
cd $TUTORIAL_DATA/fsfast-functional/sess01/bold/001
ls
在进行预处理之前,目录中只有 f.nii.gz, workmem.par和wmfir.par。现在里面有许多文件,每个文件都表示不同的预处理阶段,现在输入:
ls -ltr
这个命令将最早创建的文件放在最上面,最晚创建的文件放在最下面
Template
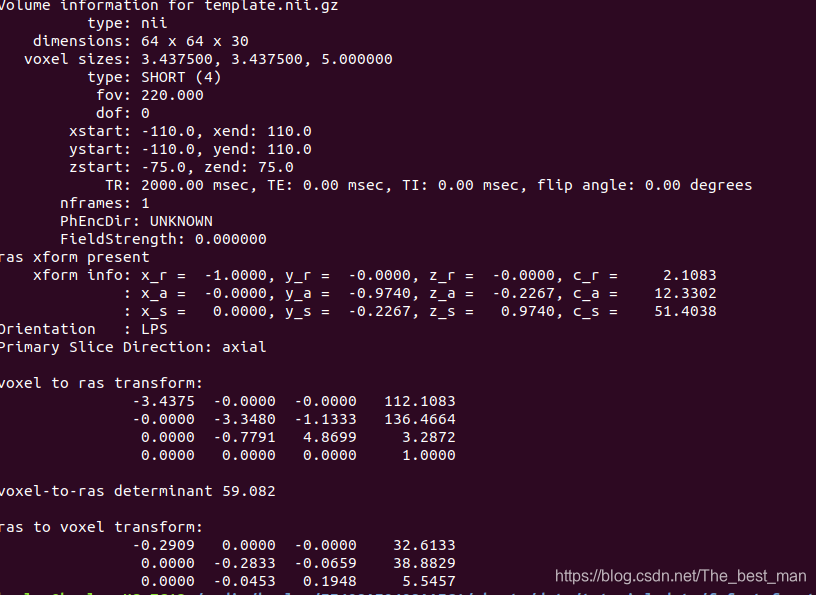
这个阶段创建 template.nii.gz 。这是原始功能数据的中间的时间点(f.nii.gz)。这是一个参考用于头动校准和配准到运行的结构图像。也被用作创建大脑的mask。
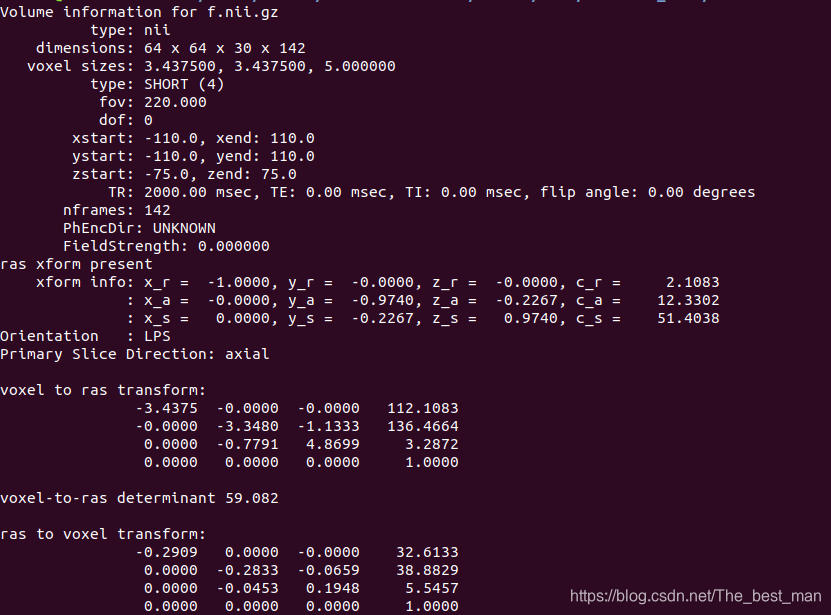
使用 mri_info验证Template与原始数据具有相同的维数和分辨率:
mri_info template.nii.gz
mri_info f.nii.gz
注意:本条命令的意思是通过mri_info之后对模板文件(template.nii.gz)与原始数据文件(f.nii.gz)进行比较,以下分别是template与f的纬度和分辨率的信息
Masking
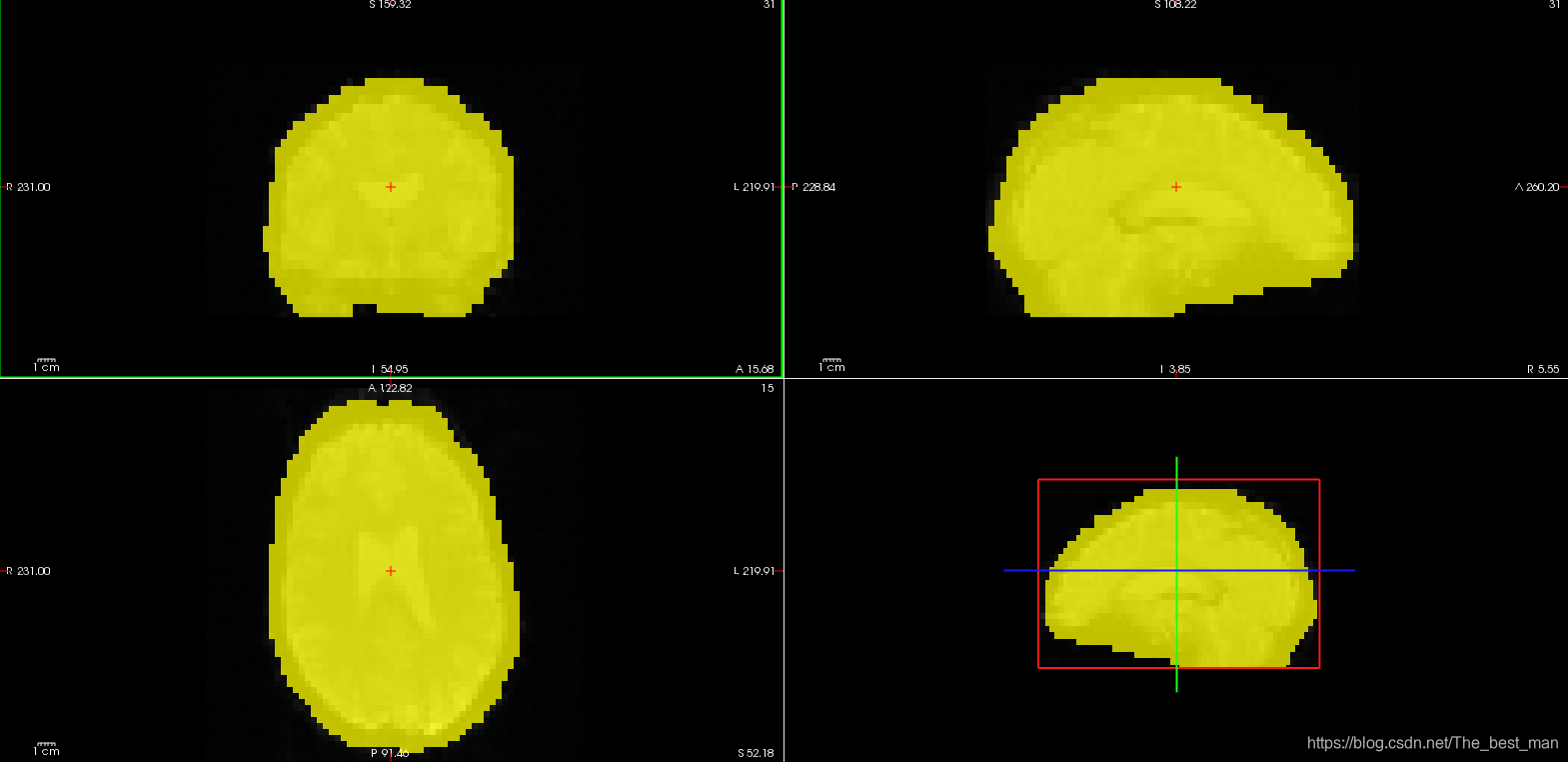
运行期间产生的Mask存放于masks目录之下。运行ls -ltr masks。(注意: ls -ltr masks 表明罗列出masks文件夹中的文件按照创建时间进行排列)您能够发现一个名称为brain.nii.gz的文件。他是通过使用FSL BET程序产生的二进制mask,还有一个由三个体素嵌入的mask名称为brain.e3.nii.gz的文件。这个eroded mask只能通过从脑组织中计算得到(减少边缘效应)。这些文件和template文件有着相同的维度。查看masks命令:
freeview -v template.nii.gz masks/brain.nii.gz:colormap=heat:opacity=0.75 -viewport cornonal
freeview -v template.nii.gz mask/.brain.e3.nii.gz:colormap=heat:opacity=0.75 -viewport coronal
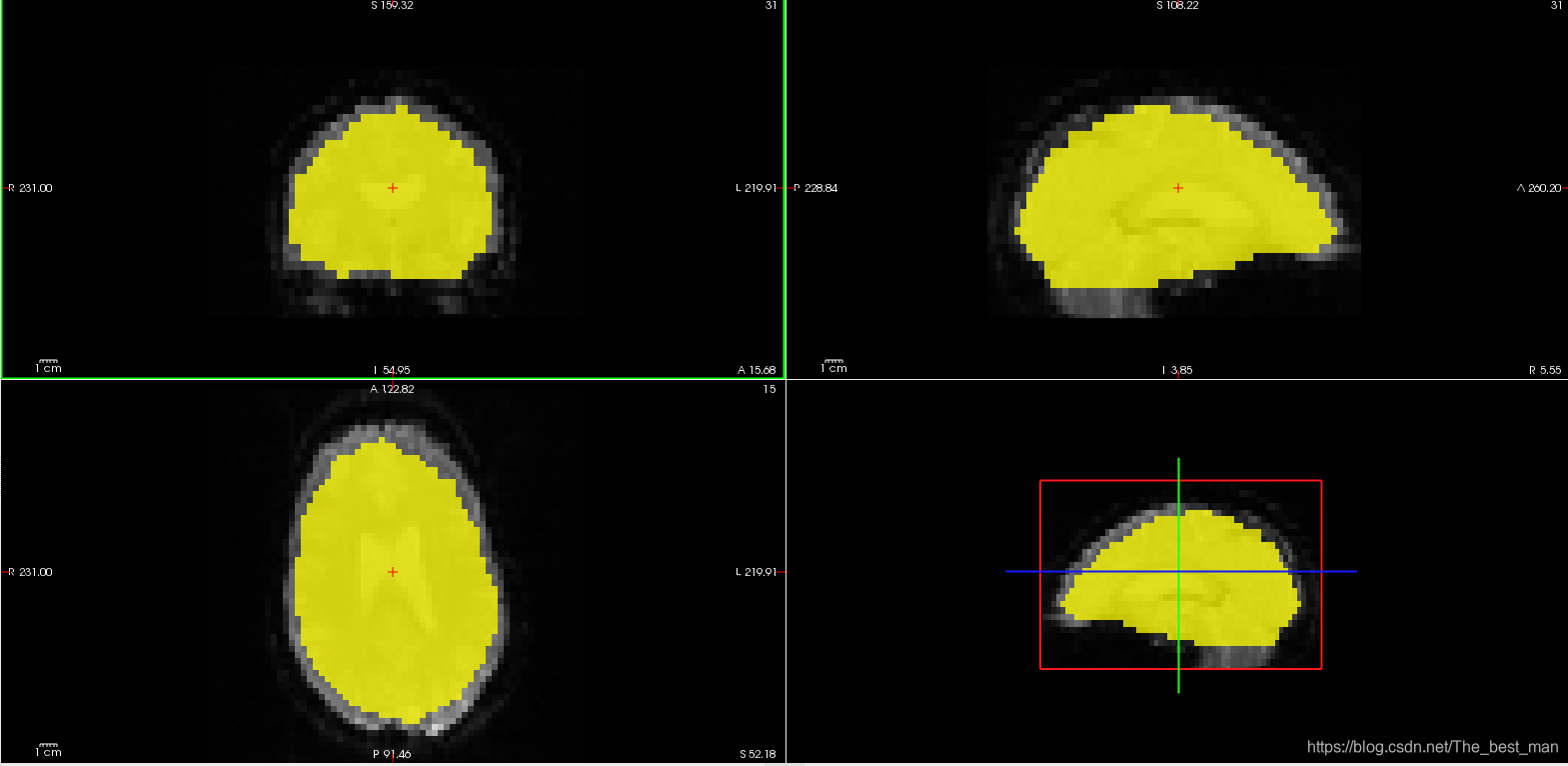
您可以通过移动不透明的滑块或者输入一个新的值来调整mask的不透明度。现在,mask的不透明度设置的是0.75(这是通过在freeview的命令行中设置 opacity=0.75),这样您就能看到哪里与template的volume重叠。brain.nii.gz是用于约束体素的操作步骤。利用eroded mask计算强度归一化和全局平均时间过程的平均功能值。其他的masks之后会讲到。
Intensity Normalization and Global Mean Time Course.
默认情况下,FS-FAST将按比例改变所有voxel的强度和时间点,以确保他们在不同的run,session,subject有相同的值。通过除以大脑中所有voxel和时间点的平均值,然后乘100,确保不同run, session, subject有相同的值。这些数据存储在globl.meanval.dat。 您可以看到这是一个简单的text文件,将值存储起来稍后再用。每个时间点的平均值也能构成waveform(text文件是 global.waveform.dat)。这个可以作为一个nuisance回归
- runs 1,2,3,4的整体意义是什么?
Functional-Anatomical Cross-modal Registration
将之后的六个文件(init.register.dof6.dat, register.dof6.lta, register.dof6.dat.sum, register.dof6.dat.param, register.dof6.dat.mincost, register.dof6.dat.log)进行配准从功能像到相同被试FreeSurfer的结构像之上。这里只有两个文件是真正重要的:register.dof6.ltaheregister.dof6.dat.mincost
- register.dof6.lta - 包含配准矩阵的text文件
- register.dof6.mincost - is a text file that contains a measure of the quality of the registration
Motion Correction
motion correction阶段产生了这些文件: fmcpr.aff12.1D, fmcpr.nii.gz, mcprextreg, mcdat2extreg.log, fmmcpr.nii.gz.mclog, fmcpr.mcdat。其中只有三个比较最重要的文件:
- fmcpr.nii.gz – 这是motion correction的功能数据。他和原始的功能数据有着相同的维度和大小。
- fmcpr.mcdat – 每个时间点的全部头动数据的text文件
- mcprextreg – 文本文件的运动校正参数组成一个正交矩阵,可以作为 nuisance回归
- 通过命令 mri_info 确认 fmcpr.nii.gz 和f.nii.gz有相同的维度
Slice-Timing Correction
Slice-Timing Correction对在2s内获得30张slice进行时间层校正。通过在时间点之间进行插入来使每个Slice与TR中间层的时间进行对齐来进行Slice-Timing Correction。通过 fmcpr.up.nii.gz进行生成.
- 使用命令
mri_info确认 fmcpr.nii.gz与f.nii.gz有着相同的维度
Resampling to Common Spaces and Spatial Smoothing
功能数据保留在native functional space。现在将其采样到公共空间。公共空间是一个geometry,即所有被试要进行体素与体素之间的配准。在FS-FAST有这样的三个空间:
- fsaverage的左半球(fmcpr.up.sm5.fsaverage.lh.nii.gz)
- fsaverage的右半球(fmcpr.up.sm5.fsaverage.rh.nii.gz)
- fsaverage的volume(MNI305 space) - 用于皮层下分析(fmcpr.sm5.mni305.2mm.nii.gz)
每一个都是将整个4D功能数据集重采样到公共空间中。进行重采样之后要进行空间平滑处理,基于2D表面的平滑处理用于表面,3D的用于Volume。查看MNI305空间volume的维度:
mri_info --dim fmcpr.up.sm5.mni305.2mm.nii.gz # 查看数据的维度和slice数量
mri_info --res fmcpr.up.sm5.mni305.2mm.nii.gz # 查看数据的分辨率

76 列76行 93slice的数量 142时间点的数量

2.000 2.000 2.000表示voxel的三个坐标轴上的大小 2000.000=TR。
这个空间的转换是基于 FreeSurfer重建过程中创建的12 DOF talairach.xfm。产看左侧大脑半球的Volume
mri_info --dim fmcpr.up.sm5.fsaverage.lh.nii.gz
mri_info --res fmcpr.up.sm5.fsaverage.lh.nii.gz
1.00 1.00 1.00 2000.000
前三个没有什么意义,因为在surface中没有行,列以及slice。最后一个2000.000表示2s。这个空间的转换时基于FreeSurfer重建过程中产生的基于表面的被试之间的配准。
First Level分析
First Level (Time Series) Analysis
这个教程描述通过 configuring 分析(设计矩阵)和对照如何分析时间序列数据。有两个不同的阶段:
- Configuring analyses and contrasts
- 通过拟合时间序列到模型上以及计算contrast的p值分析数据。
在Project的母目录下进行教程
export SUBJECTS_DIR=$TUTORIAL_DATA/fsfast-tutorial.subjects
cd $TUTORIAL_DATA/fsfast-functional




















 6609
6609











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








