数据分析服务请访问以下链接:

数据和代码获取:请查看主页个人信息
在本篇文章中,我们将使用R语言来实现一个微生物组物种丰度堆叠柱状图。下面是一步步的过程,以及与之对应的代码:首先,我们需要导入在本示例中使用到的R包。这里我们使用了tidyverse,reshape2,phyloseq和ggsci。
Step 1: 导入所需的包
rm(list=ls())
pacman::p_load(tidyverse,reshape2,phyloseq,ggsci)接下来,我们从phyloseq包中导入OTU表,并将OTU表与分类信息表合并为一个数据框。
Step 2: 读取数据
otufile <- system.file("extdata", "GP_otu_table_rand_short.txt.gz", package="phyloseq")
otu <- import_qiime(otufile)
head(otu@otu_table)[1:6, 1:10]
tax <- cbind(data.frame(otu@otu_table), data.frame(otu@tax_table)) %>%
rownames_to_column('OTU') %>%
select(OTU, Kingdom, Phylum, Class, Order, Family, Genus, Species)
# 将分类数据中的NA注释为“Unclassified”
tax[is.na(tax)] <- 'Unclassified'
head(tax)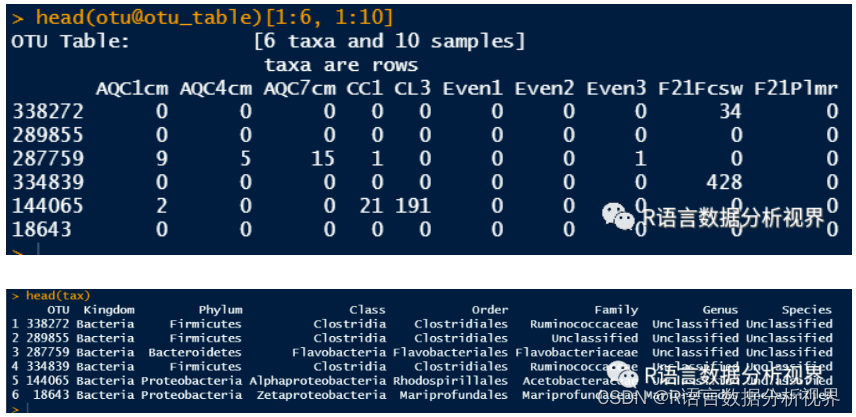
Step 3: 筛选表达量前9的OTU
top <-
data.frame(otu@otu_table) %>%
t() %>%
as.data.frame() %>%
summarise_if(is.numeric, sum, na.rm = TRUE) %>%
t() %>%
as.data.frame() %>%
set_names('SUM') %>%
arrange(desc(SUM)) %>%
rownames_to_column('OTU') %>%
head(9)
top
在此步骤中,我们从整个OTU表中筛选出表达量前9的OTU,并保存它们的信息。
Step 4: 整理数据
otu_top <-
data.frame(otu@otu_table) %>%
rownames_to_column('OTU') %>%
filter(OTU %in% top$OTU) %>%
column_to_rownames('OTU') %>%
t() %>%
as.data.frame() %>%
rownames_to_column('sample')
otu_top
# 除了前9个表达量较高的OTU之外,其他被OTU定义为“Others”
otu_others <-
data.frame(otu@otu_table) %>%
rownames_to_column('OTU') %>%
filter(! OTU %in% rownames(top)) %>%
summarise_if(is.numeric, sum, na.rm = TRUE) %>%
t() %>%
as.data.frame() %>%
set_names('Others') %>%
rownames_to_column('sample')
head(otu_others)
# 接下来这步比较关键,在分类变量数据中加入Others。
tax_top <-
tax %>%
filter(OTU %in% top$OTU) %>%
column_to_rownames('OTU') %>%
t() %>%
as.data.frame() %>%
mutate(Others = 'Others') %>%
t() %>%
as.data.frame() %>%
rownames_to_column('OTU')
tax_top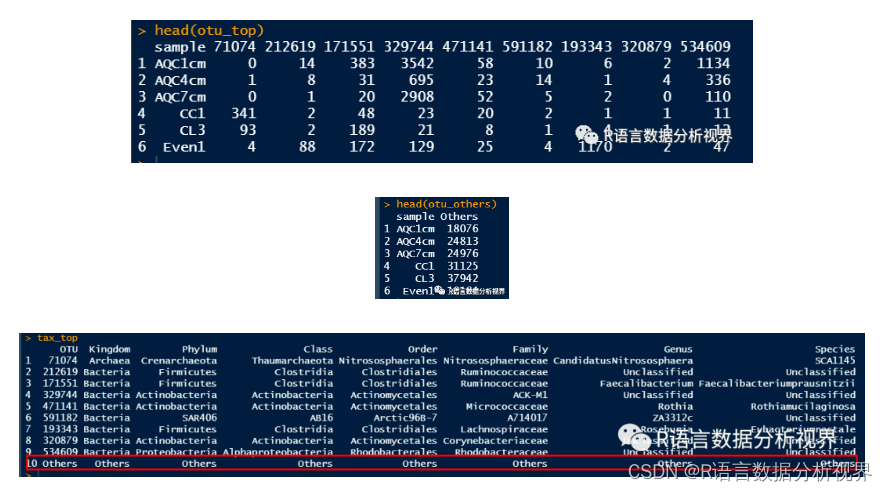
Step 5: 合并数据
# 绘图数据合并
dat <-
otu_top %>%
left_join(otu_others) %>%
melt(id.vars = 'sample') %>%
set_names(c('sample', 'OTU', 'value')) %>%
left_join(tax_top)
head(dat) 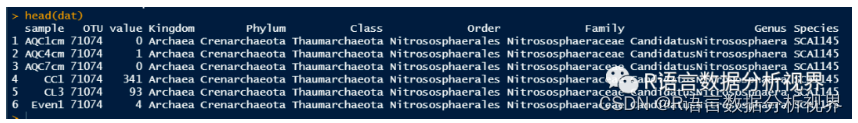
Step 6: 绘制OTU物种丰度堆叠柱状图
ggplot(dat, aes(sample, value, fill = OTU)) +
geom_bar(stat="identity", position = 'fill')+
xlab("") +
ylab("") +
theme_classic(base_size = 7) +
scale_y_continuous(expand = c(0,0)) +
ggtitle('OTU') +
guides(fill=guide_legend(title=NULL)) +
theme(axis.text.x=element_text(angle=45,vjust=1, hjust=1),
legend.key.size = unit(10, "pt")) +
ggsci::scale_fill_npg() 
为了方便后续绘制不同分类级别的物种丰度堆叠柱状图,我们定义一个通用绘图函数pic()。
Step 7: 定义绘图函数
pic <- function(name){
ggplot(dat, aes(sample, value, fill = dat[,name])) +
geom_bar(stat="identity", position = 'fill')+
xlab("") +
ylab("") +
theme_classic(base_size = 7) +
scale_y_continuous(expand = c(0,0)) +
ggtitle(name) +
guides(fill=guide_legend(title=NULL)) +
theme(axis.text.x=element_text(angle=45,vjust=1, hjust=1),
legend.key.size = unit(10, "pt")) +
ggsci::scale_fill_npg()
}Step 8: 使用绘图函数绘制各分类级别的物种丰度堆叠柱状图
# pic('OTU')
pic('Kingdom')
pic('Phylum')
pic('Class')
pic('Order')
pic('Family')
pic('Genus')
pic('Species')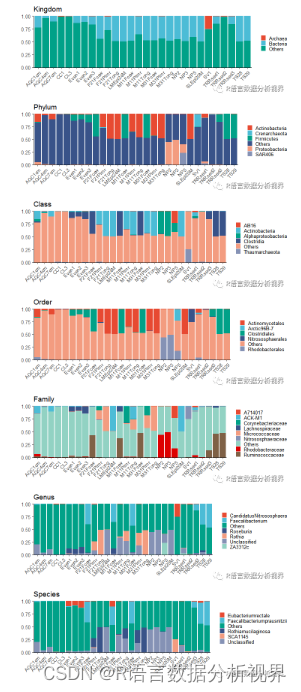
好啦!本期分享到此结束,喜欢本期内容的童鞋们请点赞、关注、转发~






















 1551
1551











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








