标题的字符数超过了博客的限制,所以进行了缩写。
Y. He, X. Chen, N. H. Tu and J. Luo, "Deep Multi-Constraint Soft Clustering Analysis for Single-Cell RNA-Seq Data via Zero-Inflated Autoencoder Embedding," in IEEE/ACM Transactions on Computational Biology and Bioinformatics, vol. 20, no. 3, pp. 2254-2265, 1 May-June 2023, doi: 10.1109/TCBB.2023.3240253.
keywords: {Data models;RNA;Clustering algorithms;Sequential analysis;Noise reduction;Noise measurement;Robustness;Cell similarity;deep clustering;single-cell RNA-seq;ZINB-model based autoencoder},
论文地址:
代码:
摘要
将细胞聚类成亚群在基于单细胞的分析中扮演着至关重要的角色,这有助于揭示细胞的异质性和多样性。由于单细胞RNA测序数据不断增加且RNA捕获率低,对高维稀疏的单细胞RNA测序数据进行聚类变得具有挑战性。在本研究中,提出了一种单细胞多约束深度软K均值聚类(scMCKC)框架。基于零膨胀负二项(ZINB)模型的自编码器,scMCKC通过考虑相似细胞之间的关联性,构建了一种新的细胞级紧密性约束,以强调簇之间的紧密性。此外,scMCKC利用由先验信息编码的成对约束来指导聚类。同时,利用加权软K均值算法确定细胞群体,根据数据和聚类中心之间的关联性分配标签。
模型

scMCKC通过将细胞相似性表达和二元加权矩阵之间的交叉熵最小化,构建了一种新颖的细胞级紧凑性约束,以调整不当的细胞标签分配。与传统的硬约束聚类方法不同,部分先验知识被编码为软成对约束,以优化潜在特征。此外,还引入了一种加权软K均值算法,并对数据进行膨胀,用于进行聚类并根据相似性确定标签。
Denoising ZINB Model-Based Autoencoder
![]()
其中 e 是高斯噪声。假设数据服从混合的零组分和负二项分布,采用基于ZINB模型的自编码器来捕获潜在分布。与传统的自编码器不同,基于ZINB的自编码器利用负二项分布来表征单细胞RNA测序,其中可学习参数为均值 μij 、离散度 θij 和权重系数 πij ,表示失活概率[28]。
与论文阅读:ZINB-Based Graph Embedding Autoencoder for Single-Cell RNA-Seq Interpretations-CSDN博客类似,
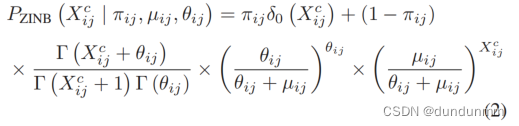
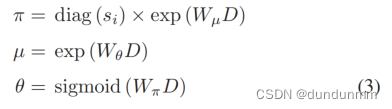
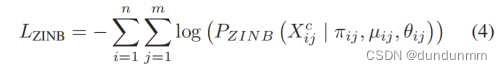
Multiple Soft Constraints
细胞级紧凑性约束
尽管单细胞RNA测序数据的表达信息嵌入在潜在层中,但在聚类层中忽略了细胞之间的相关性。由于单细胞RNA测序数据中排除了细胞之间的直接关系,因此提出了细胞级紧凑性约束,以在相似细胞之间共享信息并获得聚类友好的细胞嵌入。此外,由于噪声和高维度,细胞之间的相似性可能被错误估计。不正确的相似性可能在一定程度上误导聚类过程。
为了在细胞级别调整不正确的聚类分配,利用从潜在空间生成的判别概率向量 di 计算相似性矩阵 S。具体而言,假设 rk 是潜在空间中的可学习聚类中心,类似于 DEC [23],定义了判别概率 di 如下:
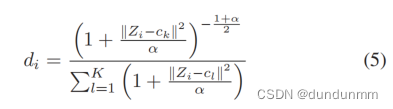
基于判别概率,相似性矩阵计算如下:

其中 \( \|\cdot\|_2 \) 表示 L2 范数[42]。与潜在表示相比,细胞相似性矩阵的构建增强了主对角线元素的表达水平。因此,引入了一个权重矩阵来减轻这种影响,其中主对角线上的值为0,其余值为1,如下所示:
\[ B_{ij} = \begin{cases} 1, & i = j \\ 0, & i \neq j \end{cases} \]
通常情况下,属于相同标签的数据点应该彼此更接近,而来自不同聚类的数据点则应保持相互远离。因此,利用权重矩阵和相似性矩阵,将这些矩阵结合起来计算以下聚类紧凑性损失:
\[ L_{\text{cell}} = -\sum_{i=1}^{n} \sum_{j=1}^{n} \left(B_{ij} \log S_{ij} + (1 - B_{ij}) \log (1 - S_{ij})\right) \]
通过这种方式,介于聚类区域之间的数据点应该更紧密,不正确的标签分配应该通过细胞相似性紧凑性约束进行调整。
成对约束
由于先验信息可用于指导细胞整合,因此利用成对约束将先验信息转化为两种类型的软约束,包括必连(ML)和禁连(CL)[32]。前者描述了细胞的联合信息,使预定义的相似实例具有较小的差异。而后者指示了分离的信息,以放大具有较少相关性的实例之间的不相似性。必连约束的损失通过分配相似的软标签来增强两个实例:
\[ L_{\text{ML}} = -\sum_{(a,b) \in \text{ML}} \log(q_a^j \times q_b^j) \]
相比之下,禁连的损失更加注重分配不同的软标签:
\[ L_{\text{CL}} = -\sum_{(a,b) \in \text{CL}} \log \left(1 - q_a^j \times q_b^j\right) \]
因此,成对损失是必连和禁连的总损失:
\[ L_{\text{pairwise}} = L_{\text{ML}} + L_{\text{CL}} \]
加权软K均值聚类
在上述小节中,原始数据被投影到低维潜在变量 Z 组成。因此,作者在潜在嵌入空间上进行聚类,而不是在去噪后的数据上进行。假设在学习空间中有 K 个聚类,其中心为 cr(1 ≤ r ≤ K)。通常情况下,K均值算法通常用于对潜在表示进行聚类。然而,数据点只能被分配到一个正确的聚类中,并且在分配的聚类中被认为是相等的。相比之下,软K均值将权重集成到K均值算法中,使数据点距离聚类中心更近或更远[35]。在这里,使用加权软K均值算法在潜在空间上实现聚类任务,其目标函数如下:
\[ L_{\text{kmeans}} = \sum_{i=1}^{n} \sum_{r=1}^{K} w_{ir} \times \|Z_i - c_r\|^2 \]
其中 $w_{ir}$ 指的是将潜在特征 $Z_i$ 分配给第 k 个类别的权重。基于上述变量,$c_k$ 表示第 k 个聚类中心如下所示:
\[ c_k = \frac{\sum_{i=1}^{n} w_{ir}Z_i}{\sum_{i=1}^{n} w_{ir}} \]
观察到聚类中心是潜在特征的加权和。一方面,对于那些距离聚类中心更近的数据点,相应的权重应增加。另一方面,对于那些远离聚类中心的数据点,相关权重应减少。因此,权重 $w_{ir}$ 应该是与潜在特征和聚类中心相关的递减函数的闭合形式[43]。基于亲和概率,将硬标签分配任务转换为加权软聚类任务。考虑到指数函数会平滑梯度下降优化过程,通过高斯核函数计算的权重如下所示:
\[ \bar{w}_{ir} = \exp \left( -\frac{\|Z_i - c_r\|^2}{\sum_{k=1}^{K} \exp \left( -\frac{\|Z_i - c_k\|^2}{\sum_{k=1}^{K} \exp \left( -\frac{\|Z_i - c_k\|^2}{2} \right)} \right)} \right) \]
为了加速算法的收敛,基于马尔可夫聚类方法对权重进行膨胀,如下所示:
\[ w_{ir} = \frac{\bar{w}_{ir}^\alpha}{\sum_{j=1}^{K} \left( \bar{w}_{ij}^\alpha \right)} \]
其中 $\alpha$ 是一个超参数,默认值为 1。加权 K 均值聚类算法的主要目的是将数据点分配到其最近的聚类中心。然而,这个过程忽略了相似细胞之间的成对距离。考虑到相似细胞更有可能属于同一聚类,引入了一种新的 Kullback-Leibler(KL)[45] 散度,以保留和增强细胞之间的相关性。采用学生 t-分布核函数[46]描述数据点之间的成对相似性,因为该分布的尾部更重,比高斯分布更具有厚重的尾部。这种特征允许潜在空间中的较大距离描述高维空间中的适度距离,从而避免将各种聚类挤在一起。
通过学生 t-分布测量的软标签分布计算如下:
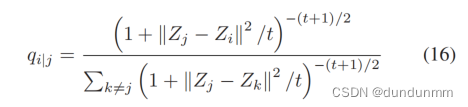
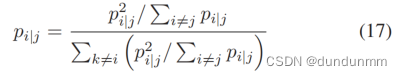
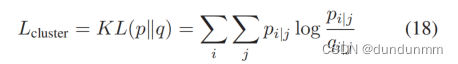
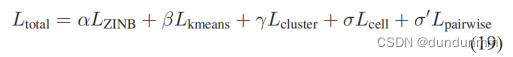
实验
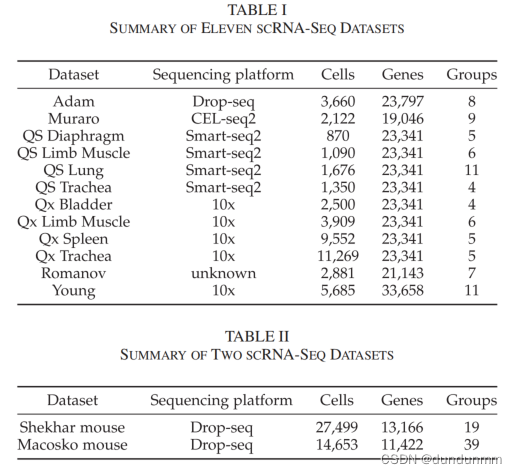
实验数据
实验结果
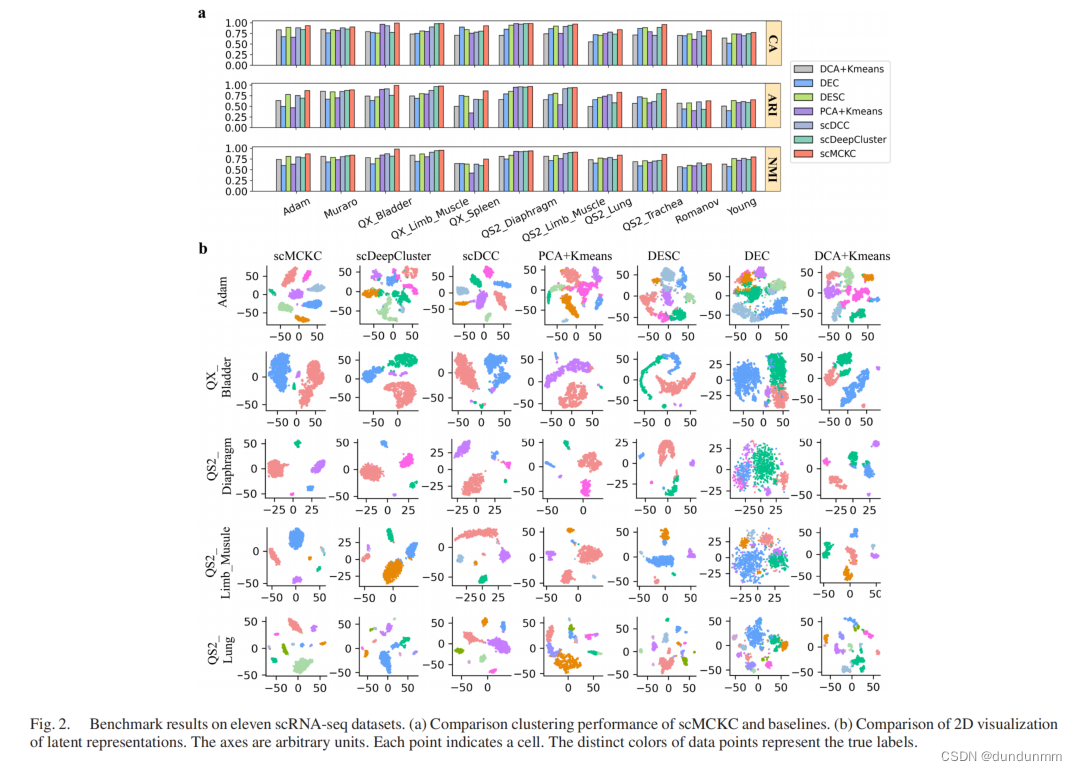
这篇文章的创新点在于多约束,在细胞级约束的基础上引入了半监督的成对约束,即先验知识。























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








