Yi Cheng, Xiuli Ma, scGAC: a graph attentional architecture for clustering single-cell RNA-seq data, Bioinformatics, Volume 38, Issue 8, March 2022, Pages 2187–2193, scGAC: a graph attentional architecture for clustering single-cell RNA-seq data | Bioinformatics | Oxford Academic
论文地址:
代码:
动机
此篇论文发表较早,动机为scRNA-seq数据的高变异性、高稀疏性和高维性给聚类分析带来了很大的挑战。虽然近年来开发了许多单细胞聚类方法,但很少有方法充分利用细胞之间的潜在关系,从而导致聚类结果不理想。
模型

模型分为三个部分:图的构建、图注意力自编码器和自优化聚类。
去噪图的构建
皮尔逊相关系数首先被计算出来以衡量细胞之间的相似性,并构建初始相似性矩阵 \(S_{n \times n}\)。然而,由于单细胞RNA测序数据具有噪声且高维度,细胞之间的相似性可能被错误估计。错误的相似性可能严重误导信息共享过程和聚类结果。
在这里,作者采用Network enhancement (NE)方法来对 \(S_{n \times n}\) 进行去噪。给定一个加权图,NE可以增加特征值间隙,同时保留其邻接矩阵的特征向量,从而更好地检测簇(Wang等,2018),使得连接不相似细胞的边的权重将被削弱。
具体来说,对 \(S_{n \times n}\) 进行NE操作,得到重新加权的相似性矩阵 \(\hat{E}_{n \times n}\),并计算去噪后的相似性矩阵 \(E_{n \times n}\) 如下:
\[ E_{ij} =
\begin{cases}
S_{ij} & \text{如果 } \hat{E}_{ij} \geq t \\
0 & \text{否则}
\end{cases}
\]
其中 \(t\) 是一个预定义的阈值。如果 \(\hat{E}_{ij}\) 太小(小于 \(t\)),则 \(S_{ij}\) 会被视为噪声并且 \(E_{ij}\) 将被设为零。
对于每个细胞,根据 \(E_{n \times n}\) 选择 \(K\) 个最相似的细胞作为它的邻居。每对邻居将在 \(A_{n \times n}\) 中通过一条边连接起来。
图注意力自编码器
这部分网络就是使用了传统的图注意自编码器。受图注意力网络(Graph Attention Networks, GAT)(Veličković等,2018)的启发,设计了一种图注意力自编码器,该自编码器由一个包含两层堆叠图注意力层的编码器和一个结构对称的解码器组成,以将拓扑信息嵌入到细胞的潜在表示中。
表示损失计算如下:
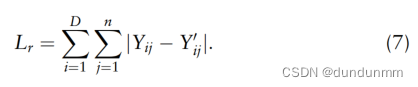
自优化聚类
在对图注意力自编码器进行预训练后,潜在表示矩阵 \(Z_{d \times n}\) 可以在细胞间关系和基因表达方面表征细胞。通过在 \(Z_{d \times n}\) 上执行 k-means(Hartigan 和 Wong,1979)可以获得一个简单的聚类结果。然而,由于聚类模块与特征学习模块没有交互,这个结果可能是次优的。因此,采用一种迭代自优化的聚类方法,使这两个模块可以互相受益,最终改进结果。
首先,基于学生 t 分布(如同在 DEC(Xie 等,2016)中)测量细胞和聚类中心(通过 k-means 初始化)之间的相似性。

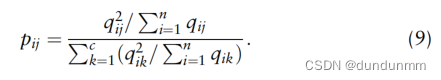
聚类中心和细胞的嵌入然后基于 \(Q_{c \times n}\) 和 \(P_{c \times n}\) 进行更新,以获得更好的聚类结果。一方面,为了更好地表征聚类,聚类中心的嵌入通过细胞的加权平均值进行更新,其中 \(Q_{c \times n}\) 作为权重,计算公式如下:

聚类标签计算如下:

聚类的损失如下:
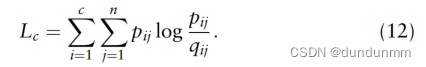
在训练过程中,基于潜在表示计算轮廓系数(Rousseeuw, 1987)来监控聚类性能,以便进行早期停止。细胞 i 的轮廓系数计算如下:
\[ s_i = \frac{b_i - a_i}{\max(a_i, b_i)}; \quad (13) \]
其中,\( a_i \) 是细胞 i 与同一簇中细胞的平均距离,\( b_i \) 是细胞 i 与不同簇中细胞的平均距离。聚类结果的轮廓系数是所有细胞的平均轮廓系数。细胞的潜在表示和簇中心将会迭代调优,直到轮廓系数收敛。
总之,总损失函数计算如下:
\[ L = L_r + c \cdot L_c; \quad (14) \]
其中,\( L_r \) 是重构损失,\( L_c \) 是聚类损失,\( c \) 是一个平衡两个损失的超参数。该损失函数将潜在表示学习和聚类结合成一个统一的框架,从而促进最终的聚类结果。
实验
实验数据
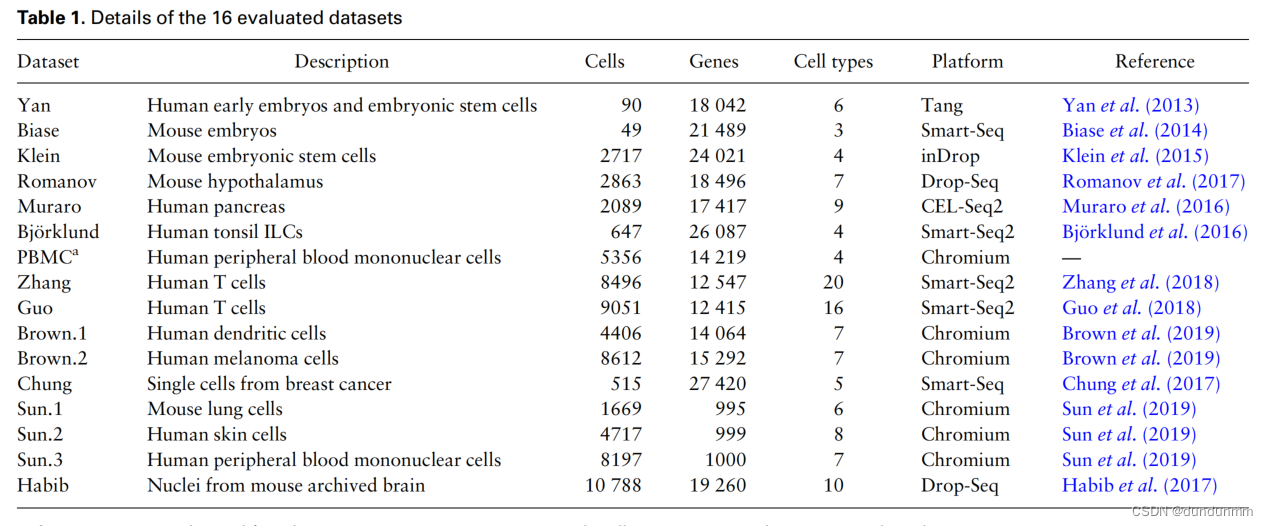
实验结果

这篇文章发表的较早,对现在来说,方法上并不新颖了,但可以作为相关工作和对比方法了解一下。






















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








