Yang, F., Wang, W., Wang, F. et al. scBERT as a large-scale pretrained deep language model for cell type annotation of single-cell RNA-seq data. Nat Mach Intell 4, 852–866 (2022). https://doi.org/10.1038/s42256-022-00534-z
论文地址:
论文代码:
TencentAILabHealthcare/scBERT (github.com)
摘要
基于单细胞RNA测序(scRNA-seq)数据对细胞类型进行注释是研究疾病进展和肿瘤微环境的前提。在此,我们展示了现有的注释方法通常存在缺乏精心整理的标记基因列表、处理批次效应不当以及难以利用潜在基因-基因交互信息的问题,削弱了这些方法的泛化性和鲁棒性。作者开发了一种基于预训练深度神经网络的模型,即单细胞双向编码表示转换器(scBERT),以克服这些挑战。遵循BERT的预训练和微调方法,scBERT通过在大量未标记的scRNA-seq数据上进行预训练,获得了对基因-基因交互的通用理解;然后将其转移到未知和用户特定的scRNA-seq数据的细胞类型注释任务中进行有监督的微调。广泛而严格的基准研究验证了scBERT在细胞类型注释、新型细胞类型发现、对批次效应的鲁棒性和模型解释性方面的卓越表现。
动机
单细胞RNA测序(scRNA-seq)已经广泛用于在单细胞水平上表征复杂组织和生物体,这彻底改变了转录组研究。准确的细胞类型注释对生物和医学研究至关重要。细胞类型注释方法可分为三类:(1) 使用标记基因进行注释,(2) 使用基于相关性的方法进行注释,(3) 通过监督分类进行注释。
"先聚类后注释"是常用的方法,其中从文献中手动整理的标记基因用于为通过无监督学习得到的聚类分配细胞类型。然而,选择标记基因依赖于研究人员的先验知识,因此容易产生偏差和错误。此外,感兴趣的细胞类型的标记基因并不总是可用的,而新型细胞类型尚未有标记基因集。而且,大多数细胞类型是由一组基因决定的,而不是单一的标记基因。如果没有适当的方法整合多个标记基因的表达信息,很难保证每个聚类的统一和准确的细胞类型分配。例如,一些自动注释方法基于标记基因应在细胞中高表达的假设。然而,即使是一些记录良好的标记基因在相应细胞类型的所有细胞中也未必高表达。这些标记基因的缺失或表达波动可能会显著影响基于标记基因的方法的准确性。
基于相关性的方法不是依赖于一组标记基因,而是测量查询样本和参考数据集之间的基因表达谱的相关性。这些方法可能受到平台和实验之间批次效应的影响。尽管存在批次效应校正方法,但区分真实的生物多样性和技术差异仍具有挑战性,因此很难保留重要的生物变异性。同时,常用的相似性测量(即余弦相似性、斯皮尔曼相关和皮尔逊相关)在测量两组高维稀疏scRNA-seq数据之间的距离时可能不够健壮或高效。
通过监督/半监督分类方法进行注释遵循机器学习中的经典范式,该范式识别基因表达谱中的模式,然后将标签从已标记数据集转移到未标记数据集。由于其对噪声和数据变异性的鲁棒性以及不依赖人工选择的标记基因,这些方法最近被广泛使用。然而,由于其有限的模型容量,大多数这些方法需要在将数据输入分类器之前执行高变异基因(HVG)选择和降维。然而,不同批次和数据集中的HVGs是可变的,这限制了它们在不同队列中的泛化能力。降维技术如主成分分析(PCA)可能会丢失高维信息以及基因水平的独立解释性。此外,这些方法中用于HVG选择和PCA的参数设置远未达成共识,并不可避免地引入人工偏差进行性能评估。由于HVGs是基于整个数据集的表达方差选择的,其中主要细胞类型占大多数方差,因此可能会忽略稀有细胞类型的关键基因。选择HVGs忽略了基因的共现和生物相互作用(特别是HVGs和非HVGs之间的相互作用),这对细胞类型注释非常有用。此外,简单的分类器如全连接网络无法有效捕捉基因间的相互作用。因此,需要一种具有改进模式识别能力的新方法来克服上述在大规模数据集上的欠拟合问题。
最近,越来越多的基于深度学习的方法被应用于scRNA-seq数据分析,并取得了优异的性能。双向编码器表示变换器(BERT)是一种最先进的基于Transformer的语言表示学习模型。由于Transformer层引入的强大自注意力机制和长距离信息整合能力,它在自然语言处理(NLP)领域取得了突破性进展。BERT的预训练和微调范式使得利用大规模未标记数据来提高AI模型的泛化能力成为可能。受此令人兴奋的进展启发,作者开发了单细胞BERT(scBERT)模型,用于scRNA-seq数据的细胞注释。遵循预训练和微调范式,作者验证了在大规模未标记scRNA-seq数据上应用自监督学习的强大效果,以提高模型的泛化能力并克服批次效应。广泛的基准测试表明,scBERT可以提供具有基因水平解释性的鲁棒且准确的细胞类型注释。据作者所知,scBERT首创了Transformer架构在scRNA-seq数据分析中的应用,设计了创新的基因嵌入。
scBERT算法--部分实验结果
原始的BERT模型提出了一种革命性的技术,通过预训练生成通用语言知识,然后通过微调将知识转移到不同配置的下游任务中。借鉴BERT的思想和范式,作者开发了一种名为scBERT的新型统一架构(见图1),该架构通过在数百万个来自不同来源的未标记scRNA-seq数据上进行预训练,学习通用的scRNA-seq知识,并通过插入分类器并利用参考数据集监督微调参数来分配细胞类型。预训练使模型能够学习基因间相互作用的一般句法,有助于消除数据集间的批次效应并提高泛化能力(见扩展数据图1a)。微调则确保每个基因的输出嵌入编码与参考数据集的转录谱更相关的上下文信息。为了注释查询细胞,scBERT通过挖掘高级隐含模式,计算细胞属于参考数据集中标记的任何细胞类型的概率(见扩展数据图1b)。如果没有高置信度分配的细胞类型,查询细胞将被标记为未分配,以防止错误分配并允许发现新的细胞类型。与原始BERT模型相比,scBERT在细胞类型注释任务中有一些创新设计。
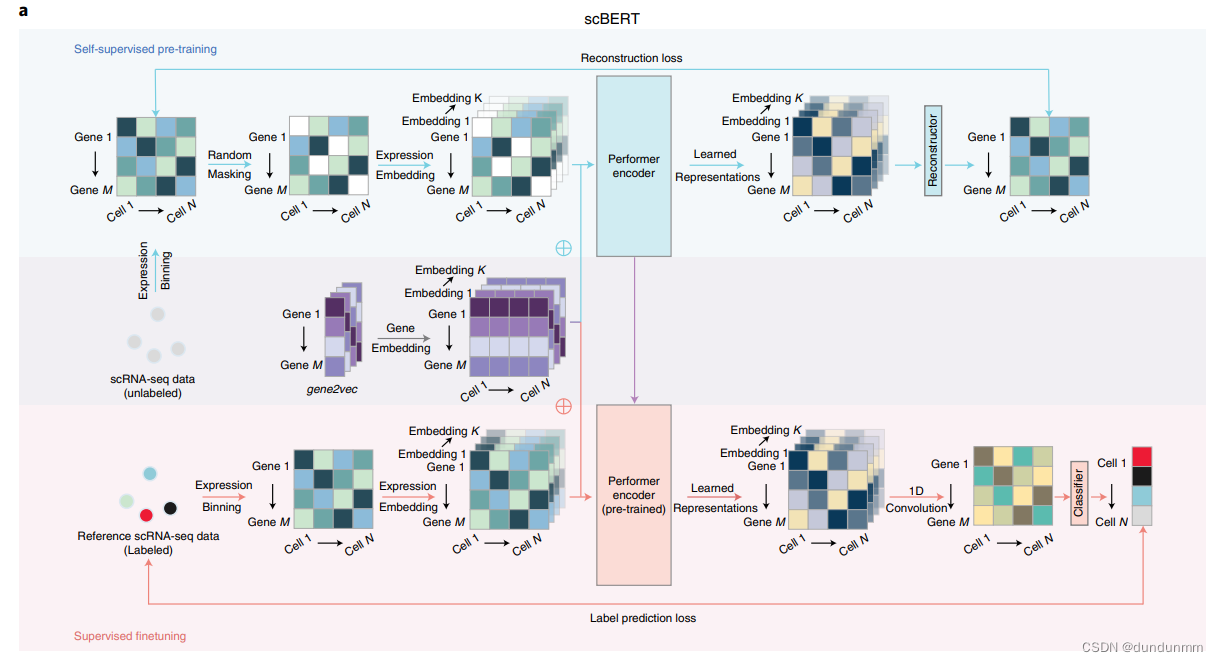

首先,BERT的嵌入包括token和位置嵌入。作者的嵌入设计在某些方面与BERT相似,同时具有独特的特征以利用基因知识。原始BERT的token嵌入是离散变量(代表一个单词),而作者模型的原始表达输入是连续变量(代表单个细胞中基因的表达),伴随着生物和技术噪声。作者借鉴了NLP领域的词袋技术,将基因的表达分箱(可以看作是每个细胞中基因转录的频率),从而将它们转换为离散值,同时还能在一定程度上减少数据噪声。由于作者输入的列顺序的打乱不会改变其含义(类似于扩展BERT理解表格数据的TaBERT),绝对位置对于基因是没有意义的。相反,基因嵌入来自于gene2vec,以表示基因身份(每个基因都有唯一的gene2vec嵌入),可以被视为相对嵌入,从一般共表达的角度捕捉语义相似性。共表达的基因保持更近的表示,基因的分布式表示已被证明在捕捉基因间相互作用方面很有用。通过这种方式,scBERT有效地形式化了基因表达的信息,为Transformer生成了特定于单细胞的嵌入(scBERT嵌入),在预训练后代表了细胞特定的表达(见扩展数据图1c)。

其次,现有的单细胞方法由于其有限的能力,必须通过选择或操作基因(如HVG选择、手动选择标记基因和PCA)来预处理原始数据,以有效地处理高维数据;这些步骤不可避免地带来人工偏差和过拟合问题,从而可能严重损害其泛化能力。相反,具有大感受野的Transformer能够有效利用scRNA-seq数据中的全局信息,通过无偏捕捉长程基因间相互作用,学习每个细胞的综合全局表示。由于计算复杂性,Transformer的输入序列长度限制为512,而大多数scRNA-seq数据包含超过10,000个基因。因此,作者用Performer替代BERT中使用的Transformer编码器,以提高模型的可扩展性,容忍超过16,000个基因输入。通过Performer,scBERT保持了完整的基因级解释,放弃了HVGs和降维的使用,让有辨别力的基因和有用的相互作用自行浮现出来(见扩展数据图1d)。scBERT因此能够在无偏数据驱动的方式中发现基因表达模式和更长的依赖关系,用于细胞类型注释。scBERT是稳定且鲁棒的,而不是严重依赖超参数选择(见扩展数据图1e)。


scBERT模型
scBERT模型采用了BERT的先进范式,并对其架构进行了调整,以解决单细胞数据分析问题。模型与BERT的关联如下所述。首先,scBERT遵循BERT的革命性方法,进行自监督预训练,并使用Transformer作为模型的骨干。其次,作者的嵌入设计在某些方面与BERT相似,同时具有利用基因知识的独特功能。从这个角度来看,作者的表达嵌入可以被视为BERT的token嵌入。由于打乱输入的列顺序不会改变其含义(类似于BERT扩展到理解表格数据的TaBERT),绝对位置对于基因是无意义的。作者使用gene2vec生成基因嵌入,这可以被视为相对嵌入,捕捉任何两个基因之间的语义相似性。第三,具有全局感受野的Transformer可以在没有绝对位置信息的情况下有效地学习全局表示和长程依赖性,在非序列数据(如图像、表格)上表现出色。
基因嵌入
在NLP中,BERT模型的输入是词嵌入,一组在预定义向量空间中的实值向量,表示单个词。词嵌入技术帮助更好地表示文本,确保具有相似意义的词具有相似的表示。然而,从scRNA-seq的角度来看,输入由单个基因构成,并且需要预定义向量空间来表示它们之间的相似性。因此,作者采用gene2vec来专门编码基因嵌入。这种方式利用过去的知识提供的基因间关系,减少了模型训练的难度。
表达嵌入
除了基因嵌入,还有一个挑战是如何利用每个基因的转录水平,这是一个连续变量。值得注意的是,词在文本中出现的频率是文本分析中的有价值信息,通常通过术语频率统计分析转换为词袋,用于下游任务。基因表达也可以被视为在生物系统中已经记录好的每个基因的出现。从这一见解出发,作者应用了传统的术语频率分析方法,将连续的表达变量离散化,并转换为200维向量,然后作为scBERT模型的token嵌入。
模型构建
BERT模型中使用Transformer作为基本单元的二次计算复杂性对于长序列扩展不太好,而scRNA-seq的基因数可以超过20,000。为此,作者采用了变换器的矩阵分解版本(即Performer)来扩大序列长度。变换器中的常规点积注意力是Q、K、V的映射,它们分别是输入查询、键和值的编码表示。双向注意力矩阵的公式如下:
\[ \text{Att}(Q, K, V) = D^{-1} (Q K^T) V, D = \text{diag} (Q K^T 1_L) \]
其中,Q = WqX, K = WkX, V = WvX是输入X的线性变换;Wq, Wk, Wv是权重矩阵作为参数;1L是长度为L的全1向量;\(\text{diag}(⋅)\)是以输入向量为对角线的对角矩阵。
Performer中的注意力矩阵描述如下:
\[ \hat{\text{Att}}(Q, K, V) = \hat{D}^{-1} (Q' ((K')^T V)) , \hat{D} = \text{diag} (Q' ((K')^T 1_L)) \]
其中,Q' = ∅(Q), K' = ∅(K),函数∅(x)定义为:

其中,c是一个正常数,ω是随机特征矩阵,m是矩阵的维数。作者构建了六层Performer编码器和每层十个头的模型。
模型训练过程包含两个阶段:在未标记数据上的自监督学习以获得预训练模型,以及在特定细胞类型注释任务上的监督学习以获得微调模型。
在未标记数据上的自监督学习
在本研究中,作者遵循BERT模型在NLP任务中的传统自学习策略,通过随机屏蔽输入数据值,并基于剩余输入进行预测。考虑到dropout现象,作者随机屏蔽了非零基因表达,然后使用剩余基因通过模型预测重构原始输入。使用交叉熵损失作为重构损失,公式如下:
\[ L_{\text{Rec}} = - \sum_{i=1}^{M} \sum_{j=1}^{N} y_{i,j} \log (p_{i,j}) \]
其中,M是细胞数,N是被屏蔽的基因表达值数;yi,j和pi,j分别是细胞i中基因j的真实和预测表达。通过这种自监督策略,模型可以在大量未标记数据上学习基因表达模式的通用深度表示,可能减轻下游微调过程的努力。
在特定任务上的监督学习
scBERT的输出是对应于每个基因的200维特征,并应用一维卷积来提取每个基因特征的抽象信息。然后,应用三层神经网络作为分类头,将基因特征转换为每个细胞类型的概率。交叉熵损失也被用于细胞类型标签预测损失,计算公式为:
\[ L_{\text{Pred}} = - \sum_{i=1}^{M} z_i \log (q_i) \]
其中zi和qi分别表示细胞i的真实细胞类型标签和预测标签。
原文是实验内容详实丰富,大量的实验数据支撑。
学习。。。
























 531
531

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








