PyMOL中常用的命令列表
- PyMOL中常用的命令列表,包括了加载文件、去除水分子、改变颜色、显示样式和图形优化等操作,可以帮助你完成全方位的分子展示设置。
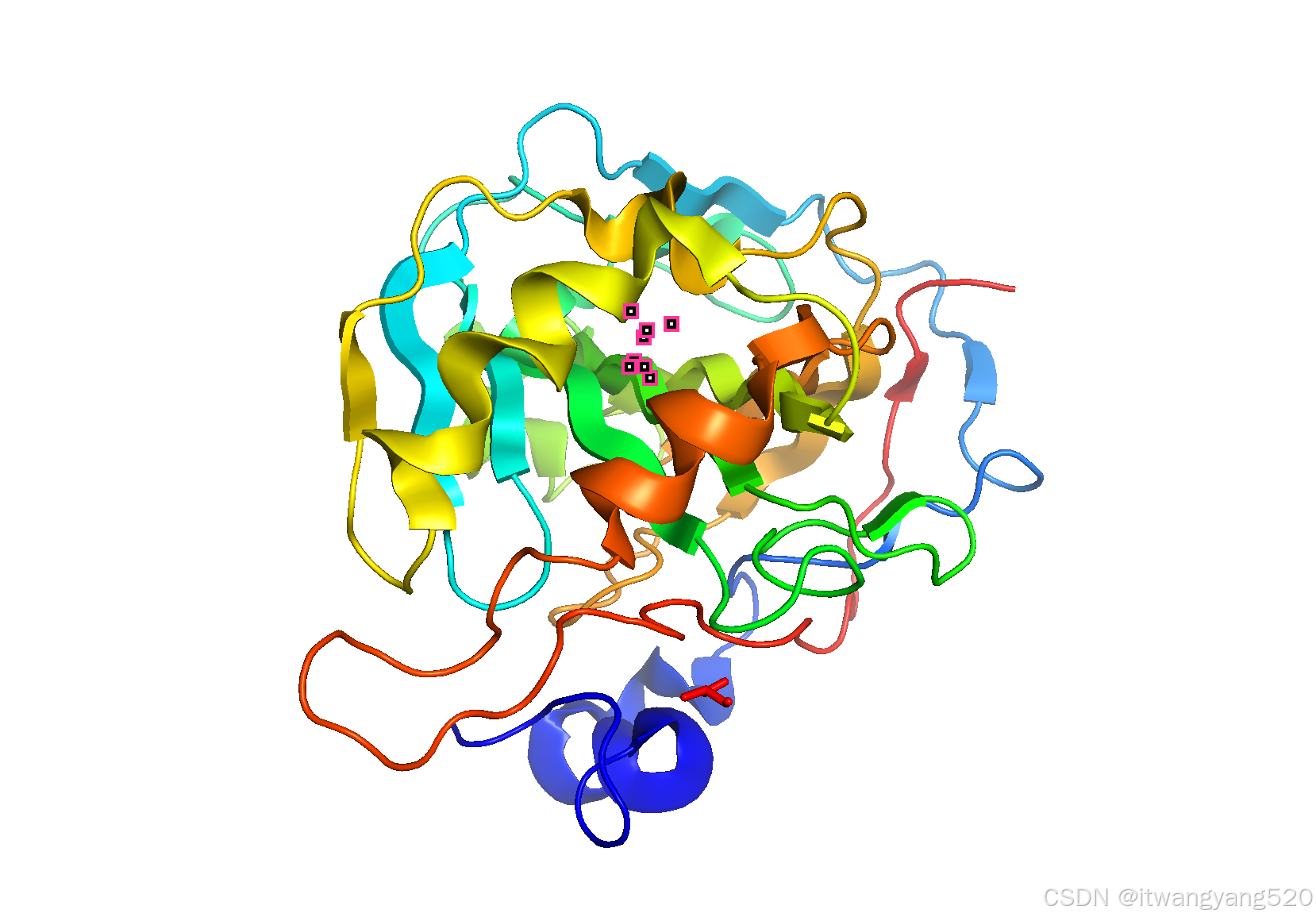
基础命令流程
-
加载分子结构
load your_file.pdb # 加载PDB文件 -
去除水分子
remove solvent # 移除所有溶剂分子,包括水 # 或者 remove resn HOH # 仅移除水分子(PDB文件中水分子通常标记为 HOH) -
分子显示样式:选择分子结构的展示样式
show cartoon # 显示为卡通样式(适用于蛋白质) show sticks # 显示为棒状模型 show spheres # 显示为球状模型 hide everything # 隐藏所有显示 -
变换颜色:为分子各部分赋予五颜六色的效果
spectrum count, rainbow # 使用彩虹色渐变效果 color blue, chain A # 为链 A 着蓝色 color red, chain B # 为链 B 着红色 util.cbc # 随机为各链分配颜色 spectrum b, blue_white_red, minimum=10, maximum=50 # 使用温度因子渐变 -
分子选择:对分子结构的不同区域进行选择
select backbone, name CA+C+N+O # 选择骨架原子 select sidechain, not backbone # 选择侧链 -
调整分子显示:对选定的分子区域应用不同的显示样式
show sticks, sidechain # 侧链显示为棒状 show spheres, backbone # 骨架显示为球状 -
优化图像显示:改善图像质量和显示效果
set cartoon_fancy_helices, 1 # 优化卡通模式中的α螺旋显示 set ray_trace_mode, 1 # 启用光线追踪模式 set antialias, 2 # 抗锯齿处理,提高图像质量 -
保存图片或场景:将生成的图像导出为图片文件
ray 800, 600 # 生成高分辨率的图片(光线追踪渲染) png my_image.png, dpi=300 # 导出图片,设置为300dpi
示例:完整操作流程
以下是一个典型的完整操作流程示例,用于生成一个五颜六色、去除水分子的蛋白质分子结构图:
load your_file.pdb # 加载PDB文件
remove resn HOH # 去掉水分子
spectrum count, rainbow # 设置五颜六色的渐变
show cartoon # 选择卡通样式
util.cbc # 随机分配颜色
set cartoon_fancy_helices, 1 # 优化卡通模式中的螺旋显示
set ray_trace_mode, 1 # 启用光线追踪模式
set antialias, 2 # 开启抗锯齿
ray 1200, 800 # 渲染高分辨率图像
png my_image.png, dpi=300 # 保存图片,设置分辨率为300dpi
常用的其他命令
-
旋转和移动:手动旋转或移动分子
rotate x, 90 # 绕X轴旋转90度 translate [10, 0, 0] # 沿X轴移动10单位 -
测量距离:测量两个原子之间的距离
distance my_dist, name CA, resi 1 and name CB # 计算特定原子间的距离
在PyMOL中,可以将小分子和蛋白质的相互作用(例如氢键、疏水作用)清晰地展示出来,并且标记相关的名称和距离。以下是实现这一效果的完整命令流程:
基础流程
-
加载蛋白质和小分子结构文件:首先加载PDB文件。这里假设蛋白质和小分子分别存储在不同的PDB文件中。
load protein.pdb, protein # 加载蛋白质文件,并命名为“protein” load ligand.pdb, ligand # 加载小分子文件,并命名为“ligand” -
显示小分子和蛋白质的不同样式:可以用不同样式展示蛋白质和小分子,使其更易识别。
show cartoon, protein # 蛋白质显示为卡通模式 color cyan, protein # 为蛋白质设置颜色为青色 show sticks, ligand # 小分子显示为棒状模型 color yellow, ligand # 为小分子设置颜色为黄色 -
选择关键残基或与小分子相互作用的区域:选择小分子与蛋白质之间的相互作用区域(例如,5 Å内的原子)。
select binding_site, br. protein within 5 of ligand # 选择与小分子5 Å内的蛋白质原子 show sticks, binding_site # 绑定位点显示为棒状 color magenta, binding_site # 将绑定位点的颜色设置为紫色 -
添加相互作用连线:检测氢键和疏水作用,并自动添加连线。
distance hbonds, protein, ligand, mode=2 # 计算氢键,显示为连线 set dash_width, 3 # 设置连线宽度 set dash_color, red # 设置连线颜色为红色 -
标记小分子和残基名称:标记小分子名称和关键残基,以便更好地了解相互作用。
label ligand, resn # 为小分子标记名称(通常为其残基名) label binding_site, resn + resi # 为关键残基标记名称和序号 -
测量并显示距离:标记小分子和蛋白质之间的距离,以量化相互作用强度。
distance interaction_distances, binding_site, ligand set dash_gap, 0.5 # 调整连线间隔 set dash_color, green # 设置距离标记的连线颜色为绿色
完整操作流程
以下是一个完整的PyMOL命令序列示例,将小分子与蛋白质对接的相互作用展示出来,包括连线、颜色和名称标记:
load protein.pdb, protein # 加载蛋白质
load ligand.pdb, ligand # 加载小分子
show cartoon, protein # 蛋白质为卡通模式
color cyan, protein # 蛋白质颜色设为青色
show sticks, ligand # 小分子为棒状模型
color yellow, ligand # 小分子颜色设为黄色
select binding_site, br. protein within 5 of ligand # 选择绑定位点
show sticks, binding_site # 显示绑定位点为棒状
color magenta, binding_site # 绑定位点颜色设为紫色
distance hbonds, protein, ligand, mode=2 # 添加氢键连线
set dash_width, 3 # 设置连线宽度
set dash_color, red # 氢键连线颜色设为红色
label ligand, resn # 标记小分子名称
label binding_site, resn + resi # 标记绑定位点的残基名称和序号
distance interaction_distances, binding_site, ligand # 显示距离
set dash_gap, 0.5 # 调整连线间隔
set dash_color, green # 距离连线颜色设为绿色
提示
-
手动添加相互作用连线:如果自动计算连线不够准确,可以手动添加。选择两个原子并计算它们的距离,如:
distance manual_dist, (ligand and name C1), (protein and resi 50 and name O)
以下是一个完整的PyMOL操作流程,包含加载文件、去除水分子、设置颜色、展示小分子和蛋白质的相互作用、标记名称、添加透明背景以及保存超清4K图像的所有步骤。所有操作步骤将以命令行的方式写出,方便直接复制到PyMOL中执行。
完整PyMOL操作命令
# 1. 加载文件
load protein.pdb, protein # 加载蛋白质文件
load ligand.pdb, ligand # 加载小分子文件
# 2. 移除水分子
remove solvent # 移除所有溶剂分子,包括水
# 或者仅移除水分子
remove resn HOH
# 3. 设置显示样式和颜色
show cartoon, protein # 将蛋白质显示为卡通模式
color cyan, protein # 蛋白质颜色设置为青色
show sticks, ligand # 将小分子显示为棒状
color yellow, ligand # 小分子颜色设置为黄色
# 4. 选择与小分子有相互作用的区域
select binding_site, br. protein within 5 of ligand # 选择与小分子5 Å内的蛋白质原子
show sticks, binding_site # 将绑定位点显示为棒状
color magenta, binding_site # 设置绑定位点的颜色为紫色
# 5. 添加相互作用连线
distance hbonds, protein, ligand, mode=2 # 自动添加氢键连线
set dash_width, 3 # 设置连线宽度
set dash_color, red # 氢键连线颜色设置为红色
# 6. 标记小分子和关键残基的名称
label ligand, resn # 标记小分子名称
label binding_site, resn + resi # 标记关键残基的名称和序号
# 7. 显示并标记距离
distance interaction_distances, binding_site, ligand # 计算并显示距离
set dash_gap, 0.5 # 调整距离连线间隔
set dash_color, green # 距离连线颜色设置为绿色
# 8. 设置透明背景
bg_color white # 设置背景颜色为白色
set ray_opaque_background, 0 # 设置背景为透明
# 9. 优化图像质量
set cartoon_fancy_helices, 1 # 优化卡通模式中的α螺旋显示
set ray_trace_mode, 1 # 启用光线追踪模式
set antialias, 2 # 开启抗锯齿处理,提升图像质量
# 10. 保存超清4K图像
ray 3840, 2160 # 渲染4K分辨率(3840x2160)的图像
png output_image.png, dpi=300 # 保存渲染结果为PNG格式,分辨率为300dpi
步骤解析
- 加载文件:使用
load命令分别加载蛋白质和小分子文件,并命名为protein和ligand。 - 去除水分子:使用
remove solvent或remove resn HOH以去除水分子。 - 设置显示样式和颜色:设置蛋白质为卡通样式,小分子为棒状模型,并为两者选择不同的颜色。
- 选择相互作用位点:选择与小分子距离5 Å内的蛋白质原子区域,显示为棒状并设为紫色。
- 添加相互作用连线:添加氢键连线,使用红色表示,设置连线宽度为3。
- 标记名称:标记小分子和蛋白质关键残基的名称,以便查看相互作用的具体信息。
- 显示距离:计算小分子与蛋白质间的距离,并用绿色标记距离连线。
- 设置透明背景:将背景设置为白色并设为透明,以便导出图像适应各种背景。
- 优化图像质量:启用光线追踪模式、抗锯齿和高级卡通样式,使图像更美观。
- 保存4K图像:渲染4K分辨率图像,并导出为PNG格式,设置为300dpi,确保图像清晰度适合打印或展示。
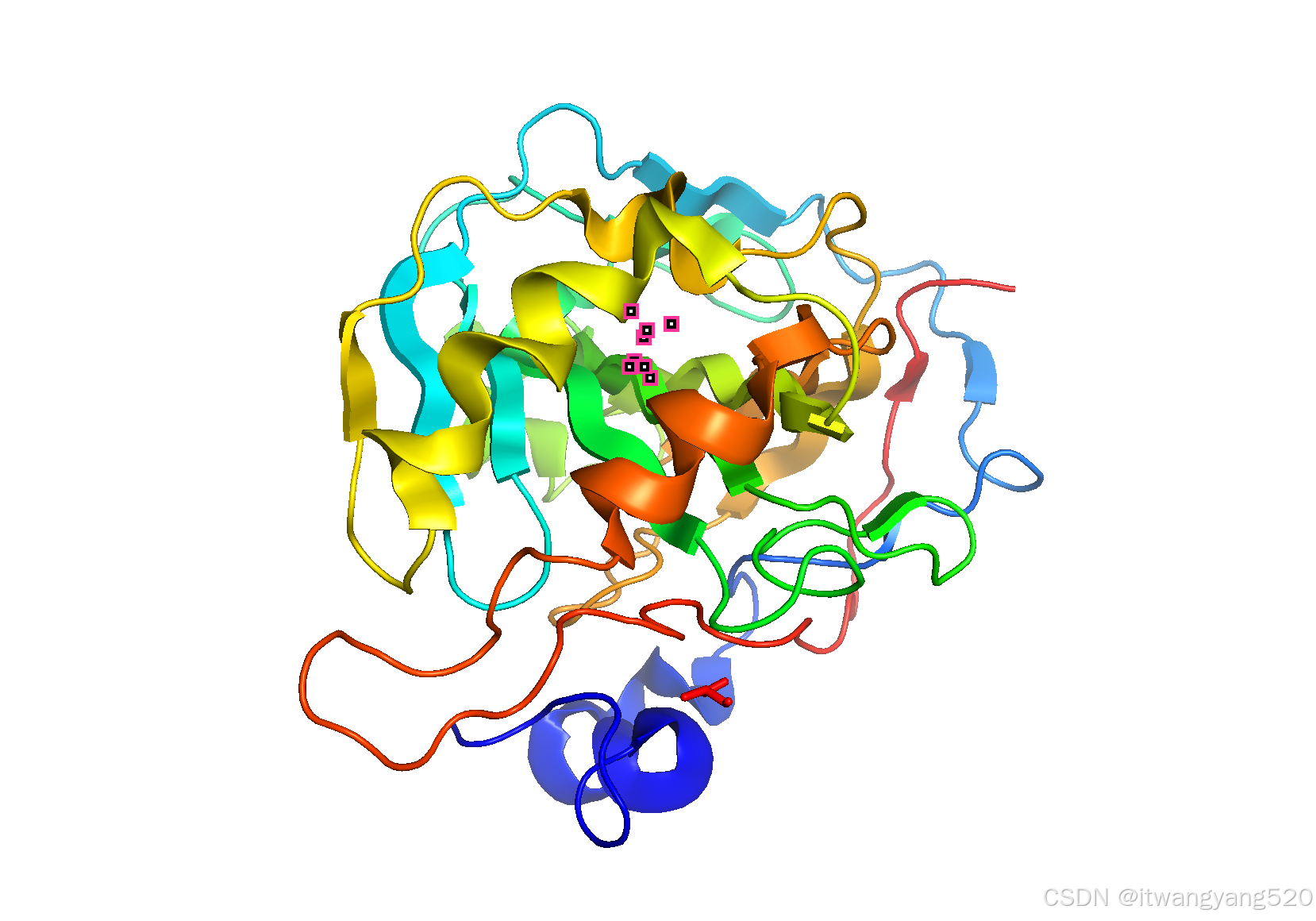
在PyMOL中要显示小分子与蛋白质之间的连接虚线以及标记名字(标签),需要确保首先定义好小分子配体的名称。例如,如果小分子加载时未自动定义为"ligand",可以将其手动命名为“ligand”。假设您的小分子是“UNK”并且已正确加载,可以尝试以下步骤:
步骤 1:重命名小分子为"ligand"
首先,通过以下命令选择并重命名小分子为“ligand”:
select ligand, resn UNK
步骤 2:定义结合位点并创建虚线显示
接着,可以通过以下命令显示蛋白质与小分子之间5Å内的相互作用,并用虚线展示:
select binding_site, br. protein within 5 of ligand
distance hbonds, binding_site, ligand, mode=2
这将计算并显示5Å内的氢键或相互作用,并用虚线表示。
步骤 3:设置虚线颜色
如果您希望虚线为红色,可以设置如下:
set dash_color, red
步骤 4:显示小分子名称标签
可以在小分子“ligand”上显示名称标签,通过以下命令:
label ligand, resn
总结完整命令
综合所有步骤,可以按以下顺序输入命令:
select ligand, resn UNK
select binding_site, br. protein within 5 of ligand
distance hbonds, binding_site, ligand, mode=2
set dash_color, red
label ligand, resn
如果小分子标签密密麻麻、遮挡视图,可以通过以下几个方式来优化显示,使得标签清晰且不影响整体视觉效果。
优化标签显示步骤:
-
只标记中心原子或配体名称
只在小分子的中心或重要原子上显示标签,避免所有原子都被标记。label ligand and name CA, resn这里将只在小分子的“CA”原子上显示标签,您可以根据实际情况选择适合的中心原子名称。
-
减少标签的字体大小
可以通过调整字体大小来减小标签,从而避免遮挡:set label_size, -0.5负值会缩小字体大小,您可以调整该值以获得理想的标签大小。
-
移动标签位置
如果希望将标签稍微移开,可以使用以下命令:set label_position, [x_offset, y_offset, z_offset]例如:
set label_position, [2, 2, 0]这将标签偏移到不同的位置,具体数值可以根据屏幕显示情况进行调整。
-
只在关键原子间显示虚线
选择一些关键的蛋白质与小分子之间的相互作用位置,这样虚线和标签就更清晰。select binding_site, br. protein within 5 of ligand distance hbonds, binding_site and name CA, ligand and name N -
隐藏其他标签
如果不需要显示所有标签,可以先清除其他不必要的标签:hide labels
完整示例
综合以上设置:
select ligand, resn UNK
select binding_site, br. protein within 5 of ligand
distance hbonds, binding_site and name CA, ligand and name N
set dash_color, red
label ligand and name CA, resn
set label_size, -0.5
这样将仅在小分子的中心原子上显示标签,同时调整字体大小,并显示蛋白与小分子间的关键相互作用,从而使得显示更清晰、直观。
要让小分子呈现五颜六色的效果,可以使用PyMOL中的color或spectrum命令来为小分子分配渐变颜色,产生丰富的视觉效果。以下是一些具体的步骤:
1. 使用spectrum命令为小分子添加渐变颜色
spectrum命令可以为小分子中的每个原子设置不同颜色,创造出渐变的彩虹效果:
spectrum count, rainbow, ligand
在这条命令中:
count是用于指定渐变的依据(可以是其他依据,如b-factor等)。rainbow表示使用彩虹色调渐变。ligand是小分子的选择名称。如果小分子不是默认命名为“ligand”,可以替换成小分子的选择名,例如“resn UNK”。
2. 使用颜色组来随机设置颜色
如果希望小分子的每个部分呈现不同的颜色,可以尝试将小分子的每一部分手动分配颜色:
color red, ligand and elem C
color yellow, ligand and elem O
color green, ligand and elem N
color cyan, ligand and elem S
color magenta, ligand and elem P
这样可以为小分子的不同原子类型分配特定颜色,使小分子呈现五颜六色的效果。
3. 混合spectrum和其他设置
还可以同时使用渐变效果和元素特定的颜色,以让小分子更具层次感。例如:
spectrum b, blue_white_red, ligand
set cartoon_color, orange, ligand
show sticks, ligand
完整示例命令
将小分子渲染为渐变的彩虹色并显示为“sticks”模式:
select ligand, resn UNK
spectrum count, rainbow, ligand
show sticks, ligand
这样,小分子就会呈现出五颜六色的效果,并在三维视图中更具吸引力。您可以根据实际视觉效果调整这些命令,达到理想的渲染效果。
在PyMOL中,为了旋转小分子并以5K分辨率保存图片,可以按照以下步骤进行:
1. 调整视角
手动旋转小分子,确保从所需角度清晰地查看它。可以使用鼠标操作或使用以下命令来微调视角:
orient ligand
这将自动调整视角,使“小分子”(配体)中心位置对齐,从而可以获得清晰的显示。
2. 设置分辨率
为了获得5K分辨率的图像(例如,5000 x 5000像素),可以使用ray命令来设置渲染分辨率。在PyMOL中,输入:
ray 5000, 5000
此命令将生成高质量的5K渲染图像。
3. 保存图像
在渲染完成后,可以保存图片。指定文件名(例如,output.png),并输入以下命令来保存渲染的图像:
png output.png, dpi=300
这样将以300 dpi分辨率保存图片为5K画质。
完整步骤示例
确保小分子在画面中心并以5K分辨率保存图片的完整命令:
orient ligand
ray 5000, 5000
png output.png, dpi=300
执行这些命令后,PyMOL会渲染并保存5K高分辨率的图像。您可以在指定的目录中找到保存的文件,并用于进一步分析或展示。
如果不想在PyMOL中显示文字标签和虚线(如氢键或其他相互作用的连接线),可以通过以下命令隐藏它们:
1. 隐藏文字标签
要隐藏所有标签,可以使用以下命令:
hide labels
2. 隐藏虚线
如果添加了虚线(如氢键),可以使用以下命令来隐藏虚线:
hide dashes
3. 移除多余的选择框
为了避免干扰视图,可以确保所有的临时选择框(如选择的残基或原子)都被取消显示:
hide selections
4. 最终命令清理界面
完整的命令集如下,可确保界面干净、无标签和线条干扰:
hide labels
hide dashes
hide selections
在执行以上命令后,您的视图将只显示小分子和蛋白质的三维结构,没有任何标签或虚线,从而获得简洁的图像。然后可以按照之前的步骤进行旋转、设置分辨率并保存高质量的图片。
























 893
893

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








