
The DECIMER 2.0:https://github.com/Kohulan/DECIMER-Image_Transformer
光学化学结构识别(OCSR)工具,其中性能最好的OCSR工具大多是基于规则。DECIMER算法的灵感主要来自于由谷歌的DeepMind开发的成功的AlphaGo Zero算法。AlphaGo Zero的成功,非常具有挑战性的问题可以通过拥有足够的数据量和使用适当的神经网络架构来充分解决。
目标是建立一个准确率约为90%的系统,可以通过在5000 - 1亿个分子数据集上训练网络来实现
Materials and methods
完全数据驱动的化学图像识别解决方案,PubChem,使用CDK去除所有显式氢,生成了SMILES,继承了规范化并保留了立体化学信息。在生成SMILES之后,将使用以下一组规则对数据集进行过滤,以获得一个平衡的数据集。
SMILES筛选:

主数据集包含3900万个分子。同样的规则集被用来生成第二个数据集,但是带有带电基团的分子包括两性离子形式和立体化学被保留了下来。此外,包含数据集中罕见标记的分子被删除,结果是一个包含大约3700万个分子的数据集。添加额外的信息导致SMILES字符的长度变长。
每个分子Image被随机旋转,并描述为8位PNG图像,分辨率为299 × 299
使用来自第二个数据集的图像集,并引入图像增强,生成第三个数据集。图像增强是使用imgaug python包应用的。对图像随机应用下列增强之一:

训练后,在预测中经常出现无效smiles,导致整体精度显著降低。为了解决这个问题,使用DeepSMILES或SELFIES。与标准的SMILES相比,DeepSMILES表现出更好的结果,但无效的DeepSMILES再次引起了类似的问题。最后,使用了SELFIES,因为通过在右括号(“]”)和左括号(“[”)中分割SELFIES,可以很容易地分割成token。不需要应用进一步的规则将其分割成一个token集。
3个数据集中的所有smile字符串都使用Python转换为SELFIES,
the datasets:
Dataset 1: PNG images + SELFIES, without stereochemical information and charged groups
Dataset 2: PNG images + SELFIES, with stereochemical information and charged groups.
Dataset 3: Augmented PNG images + SELFIES, with stereochemical information and charged groups
数据集从每个数据集的10%中选取。为了保证测试和训练数据的化学多样性相似,使用RDKIT MaxMin算法选取10%的SMILES作为测试数据集

Image feature extraction:评估了InceptionV3和EfficientNet-B3,像素值归一化为区间−1到1,利用在InceptionV3(ImageNet预训练)和EfficientNet-b3(Noisy-student预训练),提取出来是InceptionV3 feature map(8 × 8 × 2048),EfficientNet-b3 feature map(10 × 10 × 1536)
Tokenization:以 [ _ ] 规则分割,类似于如下:

encoder-decoder结构:编码器为CNN的两个网络(InceptionV3/EfficientNet-B3),解码器基于RNN(GRU)和两个全连接层。该解码器由1024个单元组成,embed_dim为512。Adam优化器进行训练,整个学习阶段的learning_rate为0.0005,损失为预测SELFIES和SELFIES ground truth的Cross_entropy
Transformer network: 4层和8 heads。attention_embed_dim为512,forward_mlp_size为2048,dropout 0.1,adam

Training the models:
V100 Tesla with 32 GB, 384 GB of RAM and two Intel(R) Xeon(R) Gold 6230 CPUs,batch_size 512,一个epoch 30min,最终跑1d5h48m,
TPU v3- node 8,batch_size 1024,每epoch 8 min 41 s,最终跑8 h 41 min 4 s
Testing the models:预测的SELFIES被解码回SMILES,然后使用包括在CDK中的PubChem指纹计算原始的和预测的SMILES的Tanimoto相似度。除了对于Tanimoto相似指数为1.0的预测,还使用CDK生成了inchi来执行同构检查,并确定Tanimoto 1.0预测是否为结构标识的良好proxy。
Results and discussion:
不同设备运行时间(壕无人性),100万数据集:

90W训练,10W测试
Image feature extraction test(InceptionV3 vs EfficentNet-B3):

EfficentNet-B3效果更好。
Encoder–decoder model vs. transformer model:

transformer效果更好。
Image feature extraction comparison using EfficientNet‑B3 and B7:
EfficientNet-B7比EfficientNet-B3 更好 2.7%。对B7,图像必须600 × 600,化学结构描述必须放大两倍正常比例。B3 299*299就可以了。大多数化学结构描述可以很容易地适应299 × 299的比例,因此要使用600 × 600的比例,图像应该放大。放大会导致信息丢失

The performance measure with increasing dataset size:为评估训练和测试数据的分割百分比如何影响训练效率
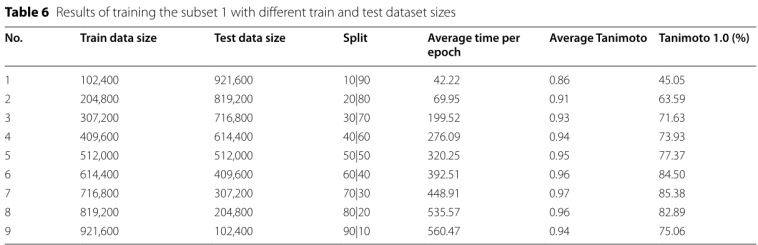
测试数据性能提高到7:3,略有下降,对此作者无法解释,
不同数据集大小的性能:

Analysis of the predictions with low Tanimoto similarity indices
语义上的小错误,如环闭合的缺失,将导致化学家眼中看起来很大的错误:

克服策略是在训练集中使用不同旋转的同一化学结构的多个描述,网络可看到同一组输入数据的更多例子。此外,采用不同/更多的图像增强方法,可以使网络更清楚地看到化学结构。


Performance of the network with training data using stereochemistry information—Dataset 2(相同的数据集,但包含了立体化学和离子信息)
通过包含这个信息,令牌的唯一数量增加了,在计算令牌分布后,令牌数量最少的分子被删除。使用RDKit MaxMin算法创建了包含37个Mio分子的新数据集,并将其拆分为训练数据集和测试数据集。
添加立体化学信息和离子,唯一的SELFIES token_dim从27个增加到61个。立体化学信息的加入增加了token的数量,也在化学结构描述(image)中引入了新的信息,如楔形键和虚线键。

数据集2的两个子集,一个包含1500万个训练分子加上150万个测试分子,另一个包含3300万个训练分子加上370万个测试分子。

平均Tanimoto较低。Tanimoto 1.0的计数也更低。



























 572
572

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








