纳入数据
差异分析的数据
保证差异分析的数据包含p值,foldchange数据,以及根据前两者区分上下调基因的分类
导入基因
set.seed(123)
logFC <- rnorm(100, mean=0, sd=1)
pvalue <- runif(100, 0, 0.05)
gene_names <- paste("Gene", 1:100, sep="")
data <- data.frame(logFC=logFC, pvalue=pvalue, gene_names=gene_names)
对基因进行上下调分类
data$group <- ifelse(data$pvalue > 0.05, "NS",
ifelse(data$pvalue < 0.05 & data$logFC > 0.7, "Up",
ifelse(data$pvalue < 0.05 & data$logFC < -0.7, "Down", "NS")))
绘制图形
初步绘制
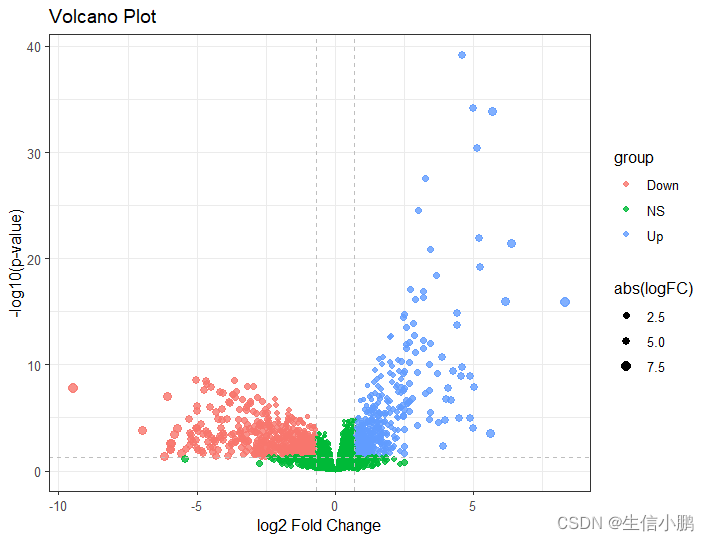
ggplot(data, aes(x=logFC, y=-log10(PValue))) +
geom_point(aes(color=group, size=abs(logFC)), alpha=0.8) +
labs(title="Volcano Plot", x="log2 Fold Change", y="-log10(p-value)") +
geom_hline(yintercept=-log10(0.05), linetype="dashed", color="black") +
geom_vline(xintercept=c(-0.7, 0.7), linetype="dashed", color="black") +
scale_size(range = c(0.5, 3)) +
guides(size = guide_legend(override.aes = list(alpha = 1)))+
theme_bw()
添加注释
sig_genes <- subset(data, pvalue < 0.01 & abs(logFC) > 1)
ggplot(data, aes(x=logFC, y=-log10(pvalue))) +
geom_point(aes(color=pvalue), alpha=0.8) +
scale_color_gradient(low="black", high="red") +
labs(title="Volcano Plot", x="log2 Fold Change", y="-log10(p-value)") +
annotate("text", x=sig_genes$logFC, y=-log10(sig_genes$pvalue),
label=sig_genes$gene_names, size=3, color="blue")























 6584
6584











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










