前言
偶然搜到这个包,不同于ggplot的强大,这个包是专门绘制火山图的,优化做得比较好,少量参数就可以做出不错的差异表达火山图。另外还是用了另一个r包的示例文件,所以大家完全可以重现这个图哈。我的大部分代码可能都会找r包里的示例文件作图,方便大家学习和重现。
- 安装并激活r包EnhancedVolcano
if (!requireNamespace('BiocManager', quietly = TRUE))
install.packages('BiocManager')
library(EnhancedVolcano)
需要注意的是,这个包可能不是特别好安装,一般的解决方法是,安装的时候哪个包报错就重装哪个包就行,不行再在语句后面加一个force=true
- 使用一下另一个r包的示例文件
BiocManager::install('GOplot')
library(GOplot)
2.1 查看一下r包的示例文件
head(EC$genelist)
data1 = EC$genelist
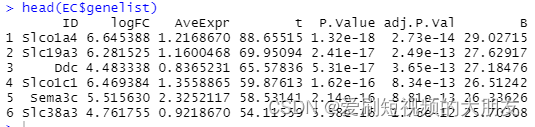 这里面就有绘制差异表达基因火山图的所有要素:基因名、logFC和p.value(注,在使用自己的数据时不需要对p值做对数计算,因为EnhancedVolcano包会自动计算)
这里面就有绘制差异表达基因火山图的所有要素:基因名、logFC和p.value(注,在使用自己的数据时不需要对p值做对数计算,因为EnhancedVolcano包会自动计算)
- 绘制火山图
EnhancedVolcano(data1,
# 基因名字
lab = data1$ID,#使用第一列作为图中点的标签
x = 'logFC',#设置以logFC为x轴
y = 'adj.P.Val',#设置以adj.P.Val为y轴(默认进行对数计算-log10(adj.P.Val)也可以指定为p值)
xlim = c(-10,10),#设置x轴的宽度
ylim = c(0,15),#设置y轴的高度
pCutoff = 0.05,#设置y轴的阈值线
FCcutoff = 1)#设置x轴阈值线
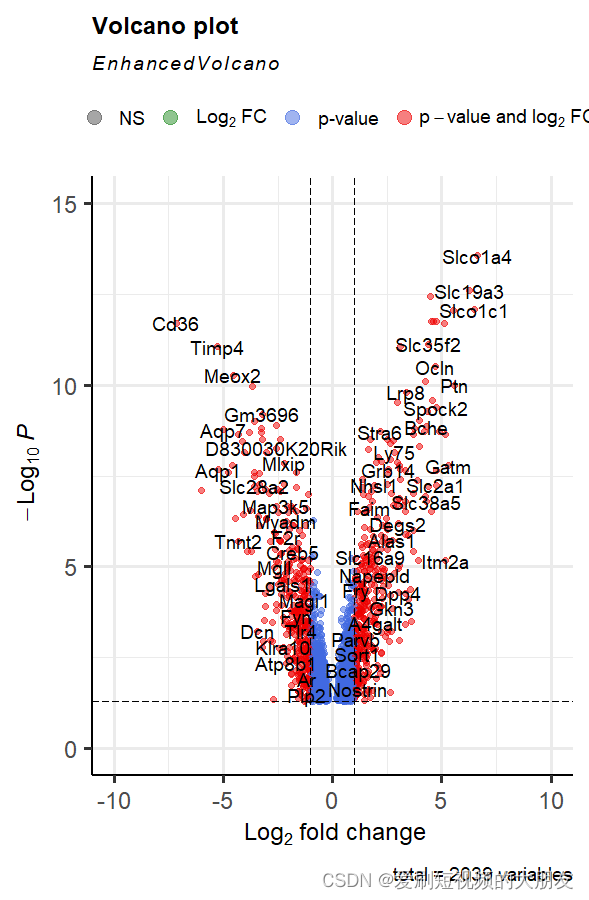
因为这个goplot包的数据只给了显著后的数据,不过也可以通过调整阈值做一个与正常火山图相似的图
#调整阈值
EnhancedVolcano(data1,
# 基因名字
lab = data1$ID,#使用第一列作为图中点的标签
x = 'logFC',#设置以logFC为x轴
y = 'adj.P.Val',#设置以adj.P.Val为y轴(默认进行对数计算-log10(adj.P.Val)也可以指定为p值)
xlim = c(-10,10),#设置x轴的宽度
ylim = c(0,15),#设置y轴的高度
pCutoff = 0.001,#设置y轴的阈值线
FCcutoff = 1.5)#设置x轴阈值线

- 查看更多功能
help(package = EnhancedVolcano)
 万能的帮助文档,掌握这个帮助文档的调出与查阅可以看懂很多代码的含义。
万能的帮助文档,掌握这个帮助文档的调出与查阅可以看懂很多代码的含义。

这个是一般的用法,下划即可看到每个参数的说明。






















 3126
3126











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








