GLM(General Linear Model),指一般线性模型,其直接将基因型X和表型数据Y进行线性拟合,使用计算公式表示为:Y = Xα + Zβ + e。公式各组分含义如下:
y:表型性状;
Xα:群体结构,作为固定效应(Fix Effect);
Zβ:标记效应(Marker Effect);
e:残差。
本文使用TASSEL5软件中GLM模型进行关联分析,使用三个示例文件,基因型文件-mdp_genotype.hmp.txt,群体结构文件-mdp_population_structure.txt和表型文件(mdp_traits.txt)。以开花期为例,使用的GLM统计模型为:
开花期=群体结构+标记效应+残差
使用TASSEL5分析流程如下:
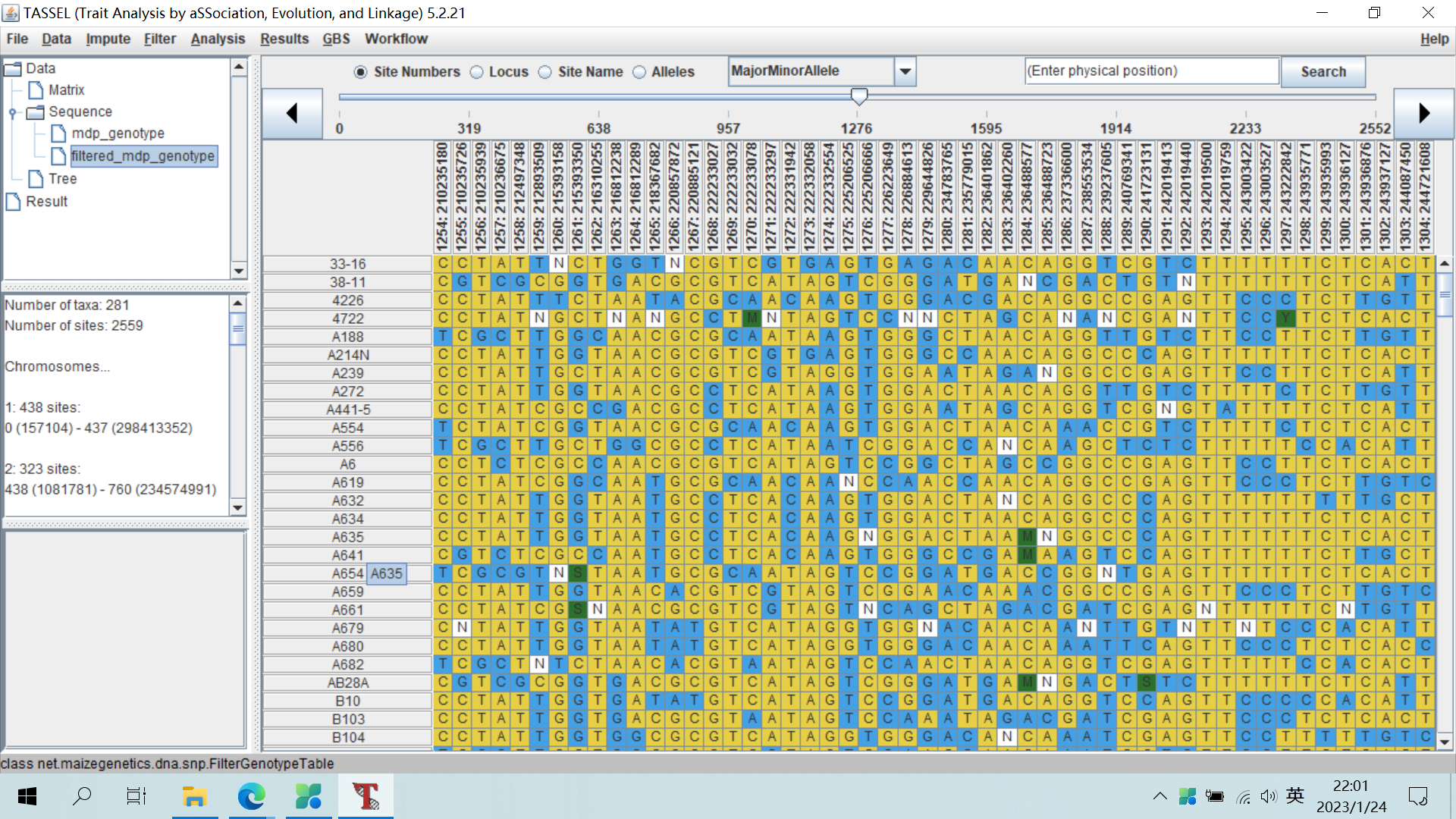
一、基因型文件过滤
主要包括删除单态性位点和低频率的位点。选择基因型文件mdp_genotype,点击Filter-Sites选项;设置Minimum Frequency为0.05,Maximum Frequency为1.0,Minimum Count为150;勾选Removeminor SNP states选项,单击Filter产生过滤后的基因型文件。
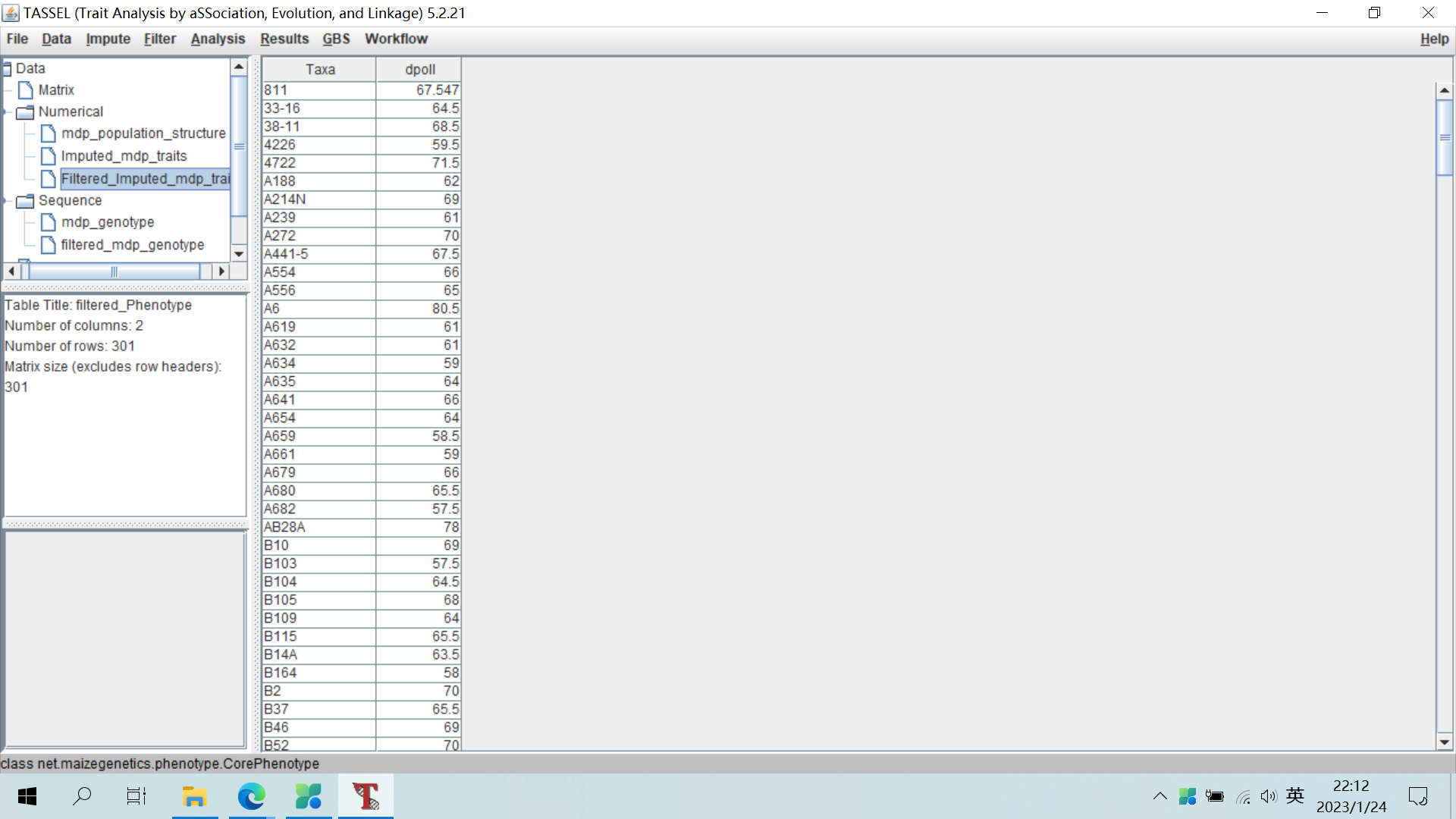
二、选择性状
选择表型文件mdp_traits.txt,点击Filter-Traits选项,只对开花期dpoll进行选择。

点击OK,产生开花期的表型文件。
三、协变量的选择
群体结构是每个群体的比例输出,本案例包含Q1+Q2+Q3三个群体,将其全部作为协变量会产生线性相依关系,GLM模型可以正确处理这种相依性,而MLM(混合线性模型)则不适用于这种选择。因此,本文使用三个群体作为协变量进行GLM关联分析。
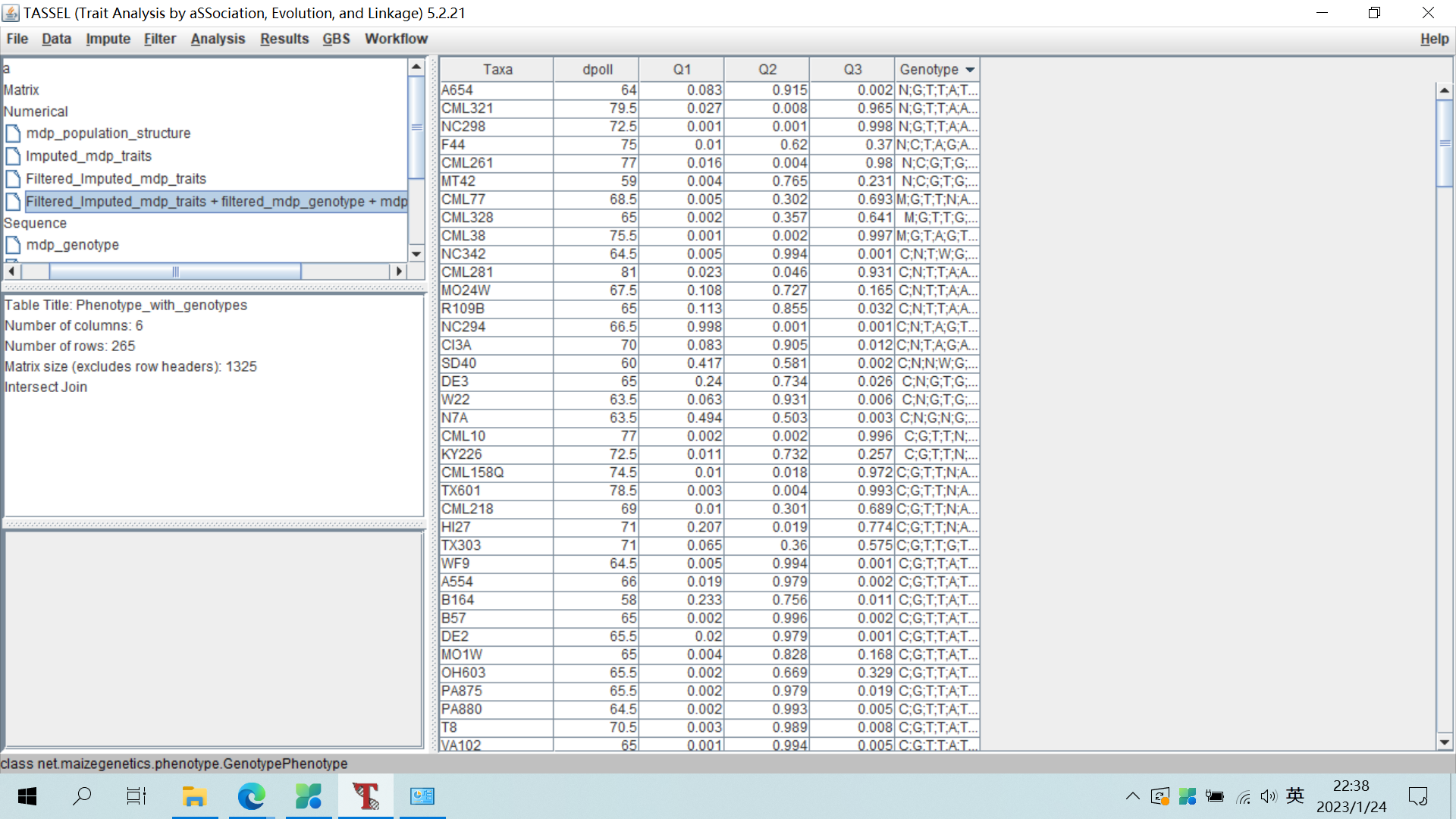
四、数据的合并
按住Ctrl键,同时选择过滤后的基因型文件、开花期表型文件以及群体结构文件,点击Data-Intersect Join选项,产生合并后的文件。
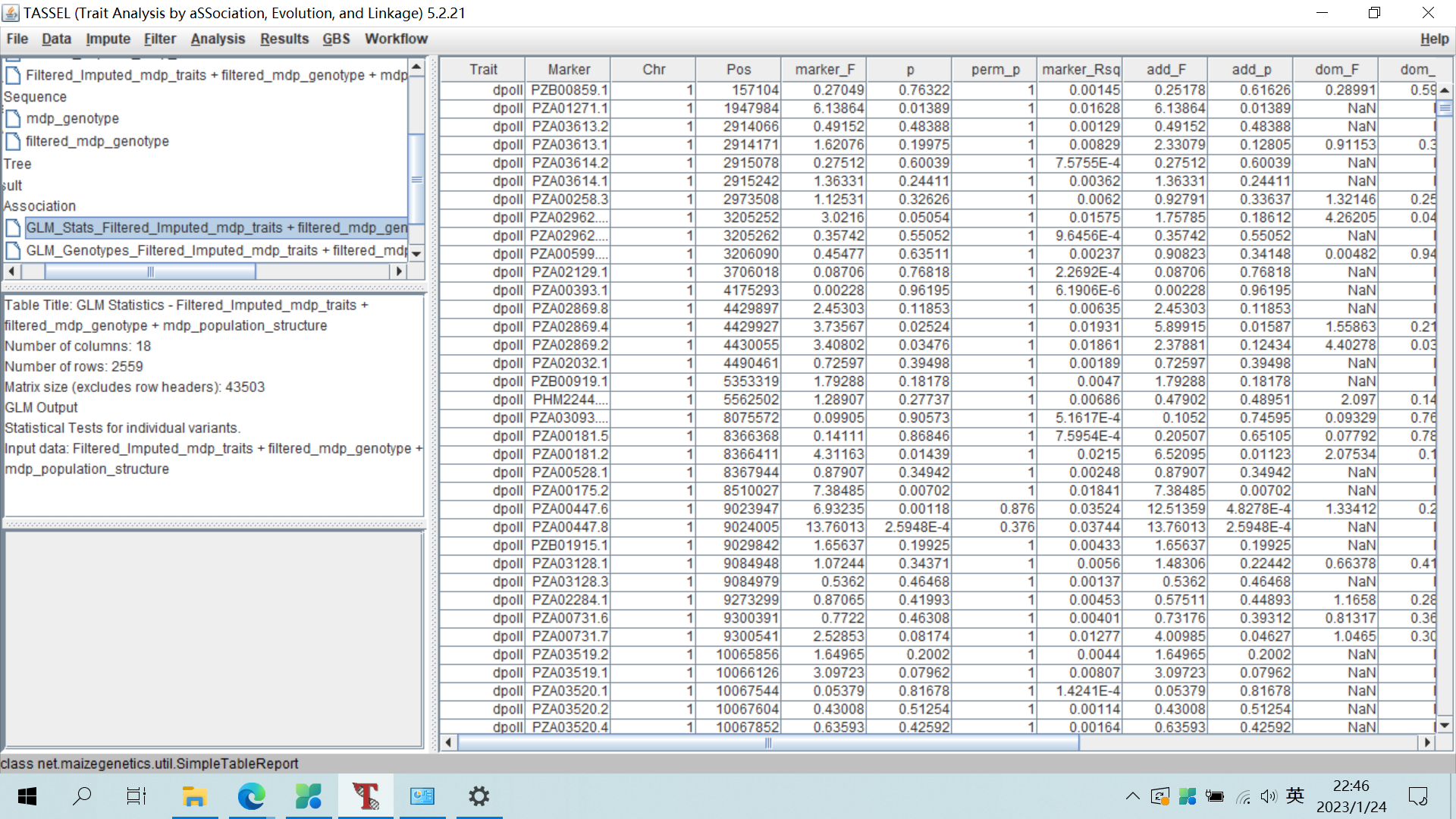
五、GLM关联分析
选择合并后的文件,点击Analysis-GLM选项进行关联分析。
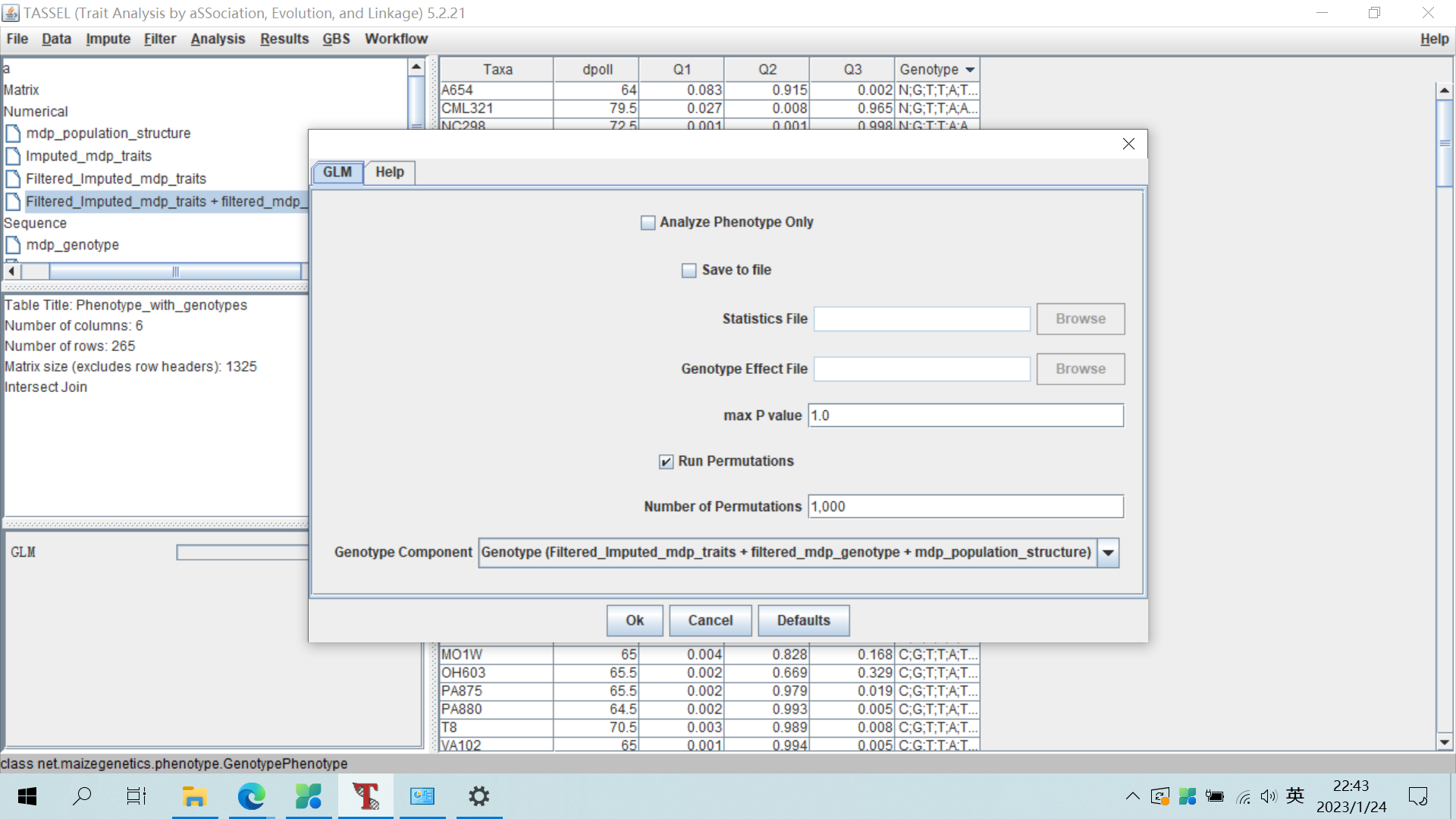
可设置Run Permutations为1000,产生分析后的结果文件。
六、关联分析结果可视化
1、曼哈顿图可视化
选择关联分析后的结果文件,点击Results-Manhattan Plot选项,即产生曼哈顿图。

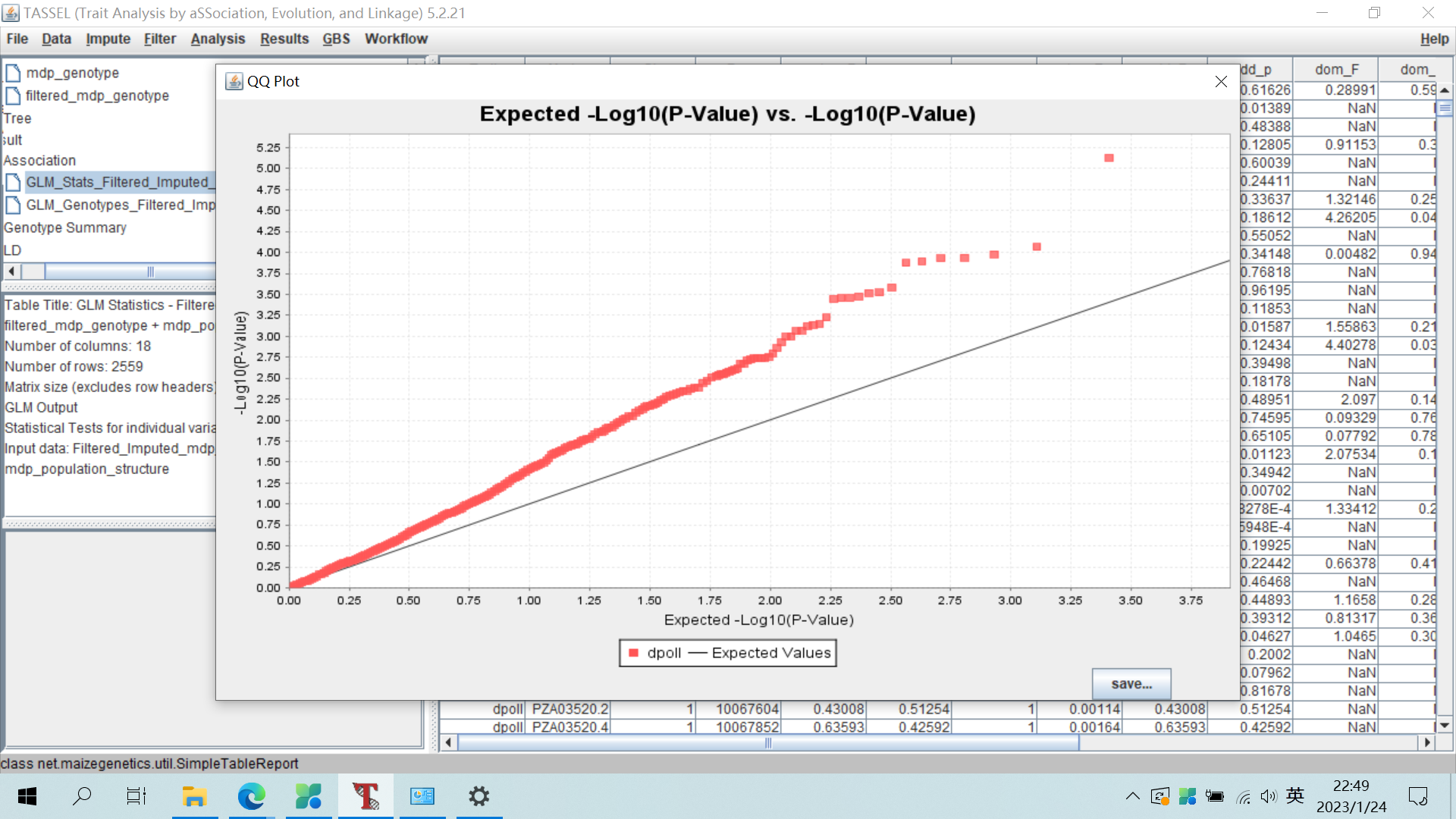
2、QQ图可视化
选择关联分析后的结果文件,点击Results-QQ Plot选项,即产生QQ图。
也可以输出关联分析结果文件,使用其他软件进行可视化。






















 1388
1388











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










