
1写在前面
最近在画热图(heatmap)时,遇到一个问题,就是如果画热图时导入的基因过多,基因名就会重叠在一起,根本没法看,非常影响颜值。😭
这里提供一种基于ComplexHeatmap的解决方案,大家往下看吧。👇
2用到的包
rm(list = ls())
library(tidyverse)
library(circlize)
library(ComplexHeatmap)
3示例数据
这里我们随机生成一个矩阵,200行,10列。😘
expr <- matrix(rnorm(2000), nrow = 200)
rownames(expr) <- paste0("gene", 1:200)
colnames(expr) <- paste0("sample",1:10)

4数据预处理
我们先做个归一化处理,如果你的数据已经做过了,就不用做了。🤗
expr_scaled <- t(scale(t(expr)))

5简单绘图
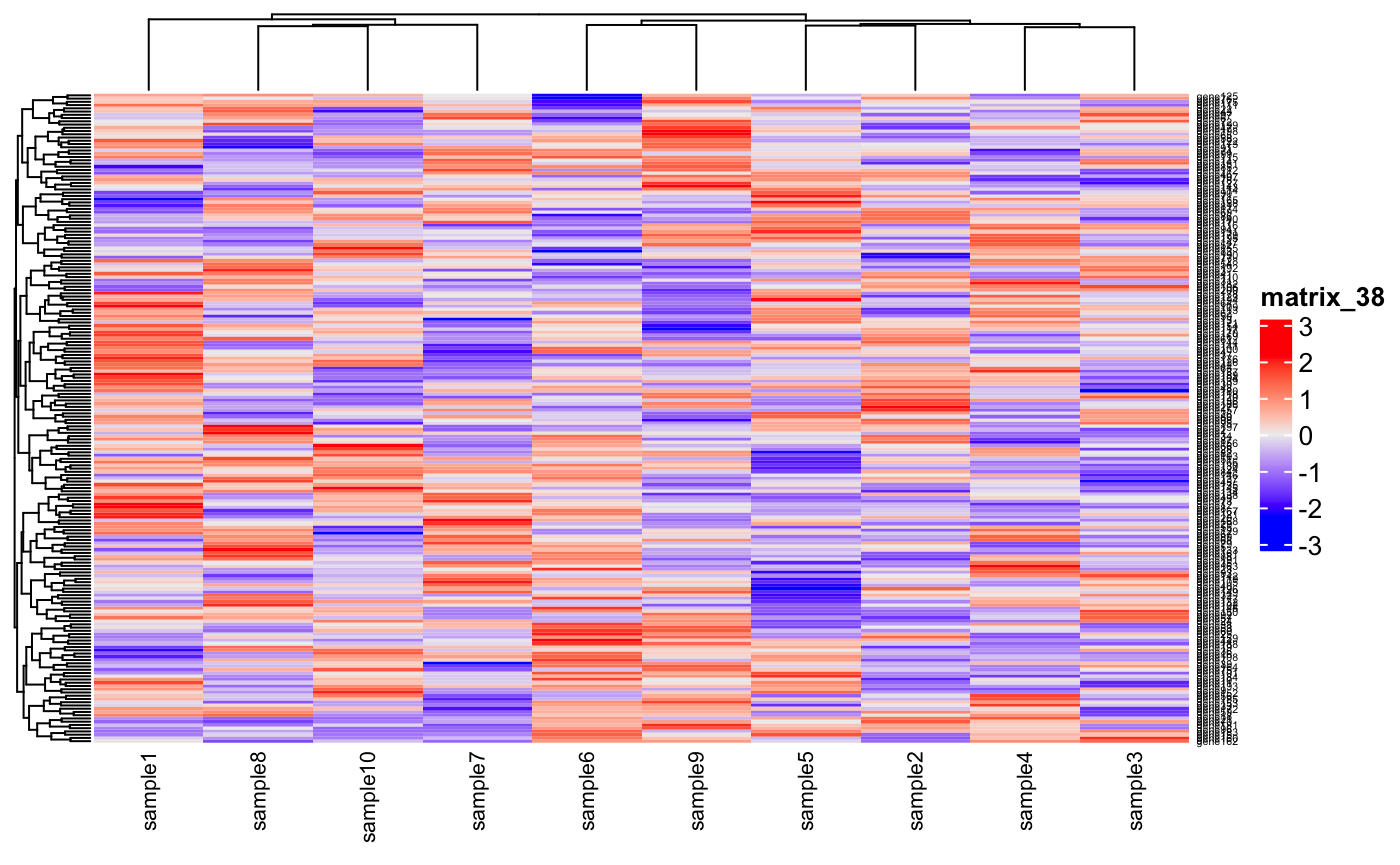
基因名(行名)全都堆在一起了,根本没法看。😭
当然你可以选择扩大画布,来显示出基因名。🤨
Heatmap(expr_scaled,
row_names_gp = gpar(fontsize = 4),
column_names_gp= gpar(fontsize = 8)
)
6标注基因
6.1 需要标注的基因名
假设我们需要标注gene1,gene10,gene100到gene106,以及gene180。👀
gene <- paste0("gene", c(1,1, 10, 100:106, 180))
6.2 提取目的基因位置
这里我们需要找到这些基因在原始矩阵的位置。🤒
为了保险起见,我们把对应的基因名也从中提取出来。🤜
genemark <- which(rownames(expr_scaled) %in% gene)
labs <- rownames(expr_scaled)[genemark]
6.3 生成行注释数据
我们把上面搞定的位置和相应文本整理成注释文件,作为行注释。🤨
ha <- rowAnnotation(
foo = anno_mark(at = genemark,
labels = labs,
labels_gp = gpar(fontsize = 8)
))
6.4 可视化
解决啦! 现在这些基因名就没有再“挤”在一起啦,嘿嘿。
Heatmap(expr_scaled,
cluster_rows = F,
right_annotation = ha,
show_row_names = F,
row_names_gp = gpar(fontsize = 4)
)

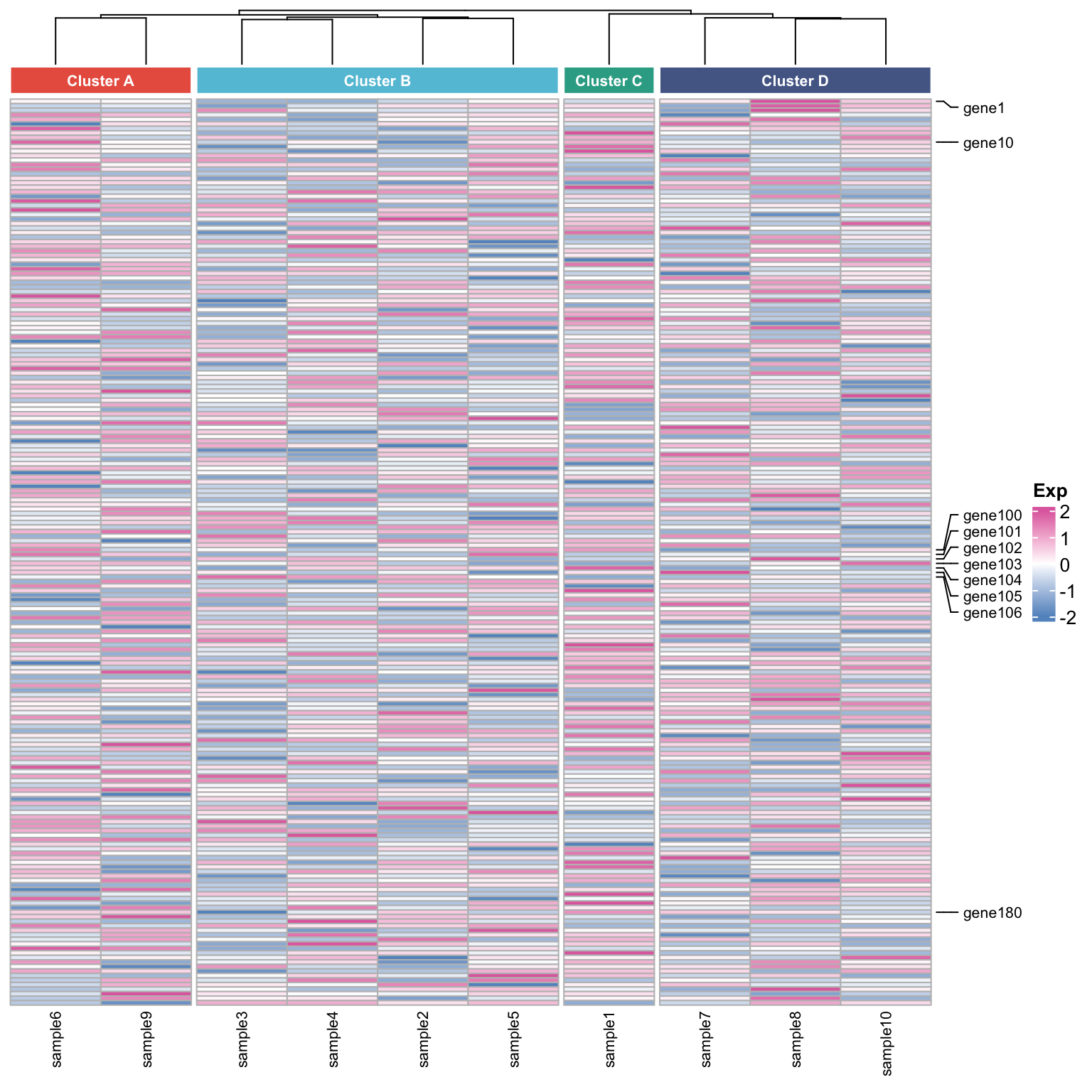
7美化一下
这里换一个我个人常用的配色,并对样本进行聚类注释。🥰
7.1 列聚类注释
class <- anno_block(
gp = gpar(fill = c("#E64B35E5", "#4DBBD5E5", "#00A087E5", "#3C5488E5"),
col="white"),
height = unit(5, "mm"),
labels = c("Cluster A", "Cluster B", "Cluster C","Cluster D"),
labels_gp = gpar(col = "white", fontsize = 8,fontface="bold")
)
clusters <- HeatmapAnnotation(group = class)
7.2 可视化
哈哈哈哈哈哈哈,美不胜收呀!~🤩
妈妈再也不用担心我的热图注释“挤”在一起啦!!!🤜
Heatmap(expr_scaled,
## row parameters
cluster_rows = F,
right_annotation = ha,
show_row_names = F,
show_row_dend = F,
row_names_gp = gpar(fontsize = 4),
## column parameters
column_split= 4,
column_title = NULL,
column_names_gp = gpar(fontsize = 8),
top_annotation = clusters,
## color
col = colorRamp2(c(-2,0,2), c("#6395C7", "white", "#E06EAD")),
## legend
name = "Exp",
rect_gp = gpar(col="grey")
)

点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰
📍 🤨 Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案)
📍 🤩 scRNA-seq | 吐血整理的单细胞入门教程
📍 🤔 Reticulate | 如何在Rstudio中优雅地调用Python!?
📍 🤣 NetworkD3 | 让我们一起画个动态的桑基图吧~
📍 🤩 RColorBrewer | 再多的配色也能轻松搞定!~
📍 🧐 rms | 批量完成你的线性回归
📍 🤒 CMplot | 连Nature上的曼哈顿图都卷起来啦
📍 🤩 CMplot | 完美复刻Nature上的曼哈顿图
📍 🤠 Network | 高颜值动态网络可视化工具
📍 🤗 boxjitter | 完美复刻Nature上的高颜值统计图
📍 🤫 linkET | 完美解决ggcor安装失败方案(附教程)
📍 ......

本文由 mdnice 多平台发布





















 2万+
2万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








