在地球生命历史的大部分时间里,基因信息都通过指定的20种氨基酸的密码来传递。氨基酸是蛋白质的基本组成部分,蛋白质在细胞中承担了大部分繁重的工作;它们的侧链决定了蛋白质的折叠、相互作用和化学活性。通过限制可用的侧链,可自然有效地限制蛋白质的反应类型。
作为1980年代的博士生Peter Schultz不禁想知道,为什么自然界要以这种方式限制自己,并着手试图规避这一局限。几年后,作为加州大学伯克利分校的教授,Schultz和他的团队通过调整蛋白质合成的机制成功地实现了这一目标。尽管这项工作仅限于试管内,但它标志着破解遗传密码的努力取得了关键的早期成功。
从那时起,许多研究人员都追随Schultz的脚步,调整细胞内制造蛋白质的机制,既改变现有的大分子,也利用全新的基本组成部分创建聚合物。由此产生的分子可以用于研究以及治疗方法和材料的开发。但这是一项艰巨的任务,因为蛋白质合成是一项至关重要的细胞功能,难以轻易改变。
英国剑桥MRC分子生物学实验室的合成与化学生物学家Jason Chin表示:“这是一个超级令人兴奋的领域。从一开始,我认为我们正在做一些非常特别的事情。”
从体外到体内
要理解为什么,我们必须考虑蛋白质是如何合成的(参见“蛋白质破解”)。
在转录过程中,DNA被复制成RNA,然后在被称为核糖体的分子机器中被翻译成蛋白质。RNA中的每个碱基三联体(腺嘌呤、胞嘧啶、鸟嘌呤和尿嘧啶)代表遗传密码的一个词或密码子。总共有64个密码子——其中61个编码氨基酸,3个表示核糖体终止信号。

氨基酸通过转运RNA(tRNA)被带到核糖体,每个tRNA与一个互补密码子相对应。氨酰-tRNA合成酶将tRNA与其对应的氨基酸耦合。
要让这个系统在试管中接受非天然的,或者说“非典型”的氨基酸,需要进行几个关键调整。首先,Schultz的团队设计了一种方法,通过化学方式将非天然氨基酸与能识别三个终止密码子之一的tRNA相结合。然后,他们在青霉素抗性蛋白β-内酰胺酶的基因中引入一个终止密码子。利用这些修改,研究人员探索了不同非典型蛋白质变体如何影响酶活性[1]。
但这仅限于试管内。为了将非典型氨基酸嵌入活细胞,团队必须在不改变它的情况下补充翻译装置。他们通过识别和修改对宿主细胞具有生物正交性的tRNA和氨酰-tRNA合成酶来实现这一目标,也就是说,它们无法识别(和被识别)细胞内翻译机制的其他部分。
现任斯克里普斯研究所(Scripps Research)总裁兼首席执行官的Schultz说:“人们认为这将是非常非常困难的。幸运的是,我们当时足够天真地认为我们可以做到。”(Schultz创建了几家公司,其中包括位于加州拉荷亚的Ambrx,该公司利用非典型氨基酸开发生物治疗方法。他目前是Ambrx科学顾问委员会的成员。)
最终,Schultz的团队确定了来自一种古细菌Methanococcus jannaschii的tRNA-合成酶对,这些酶不容易识别以及他们计划修改的大肠杆菌的tRNA和合成酶。该团队优化了这些分子,使其对新氨基酸具有选择性。2001年,研究人员将这个系统植入大肠杆菌,使细胞能够使用其自身的翻译机器将非标准氨基酸嵌入蛋白质中[2]。
曾在Schultz实验室做博士后研究的Chin说:“或许这是遗传密码在体内扩展的开始。”
Schultz的团队和其他研究人员使用这种方法将200多种非标准氨基酸遗传编码到蛋白质中,为研究蛋白质结构和功能提供了强大的工具。科学家们可以引入荧光标记或其他标记到蛋白质中,也可以进行光笼实验(其主要原理是使用光笼(photocaged)非天然氨基酸替换目标蛋白的关键活性位点,令蛋白在无光照激活的条件下处于失活状态;随后,经光照激活,光笼小分子离去,蛋白质的功能得以恢复。),其中蛋白质通过可以用光去除的化学基团被失活("囚禁")。
科学家们还超越了大肠杆菌,改变线虫、果蝇、植物甚至小鼠的遗传密码。例如,Chin与剑桥大学的神经科学家Michael Hastings及其同事,改变了编码氨基酸吡咯赖氨酸的tRNA和tRNA合成酶,将一种称为炔基赖氨酸N6-[(2-丙炔氧基)甲酰基]-L-赖氨酸(AlkK)的氨基酸结合到蛋白质中。他们将其用作在小鼠脑细胞中打开和关闭基因的开关。研究人员删除了调节昼夜节律(由身体内部时钟控制的日常循环)的一个关键蛋白质,并将一种只能在AlkK存在的情况下翻译的新基因引入细胞中。由于小鼠无法自行合成该氨基酸,研究人员可以通过向啮齿动物的饮水中添加或去除氨基酸来控制其昼夜节律[3]。
Schultz一直在利用扩展的遗传密码来探究如果有超过20种氨基酸供其使用,生物体会是什么样子。在一项研究中,他的团队创建了一个大肠杆菌O-琥珀酰基转移酶的变体库,该酶参与了蛋氨酸的生物合成,对温度特别敏感,其中酶的催化中心外的每个密码子都被非典型的氨基酸(对苯二甲酰基)丙氨酸(pBzF)所替代。该团队发现了一个变体,在高达比典型范围高21°C的温度下仍然稳定——这一壮举被归因于pBzF和另一种氨基酸之间形成的化学键[4]。
Chin、Schultz等研究人员也开始将这项技术应用于治疗学的研发。例如,加州大学旧金山分校的化学生物学家Lei Wang正在开发基于蛋白质的药物,可以与其他生物分子形成共价键。
蛋白质通常通过相对较弱的非共价相互作用与其他分子相互作用,但共价键可以增强它们的效力,Wang说。2020年,他的团队将非典型氨基酸氟硫酸盐-L-酪氨酸(FSY)整合到PD-1(一种免疫检查点蛋白,有助于控制身体的免疫反应)中,以创造抗肿瘤药物。通常,T细胞上的PD-1和肿瘤细胞上的PD-L1之间的相互作用会抑制免疫反应,使肿瘤能够逃脱免疫监视。当Wang的团队将含有FSY的PD-1注射到移植有人类癌细胞的小鼠中时,该蛋白质与PD-L1形成了不可逆的共价键,导致肿瘤缩小[5]。Wang是拉霍亚Enlaza Therapeutics公司的一位科学顾问,该公司开发了这种治疗策略。
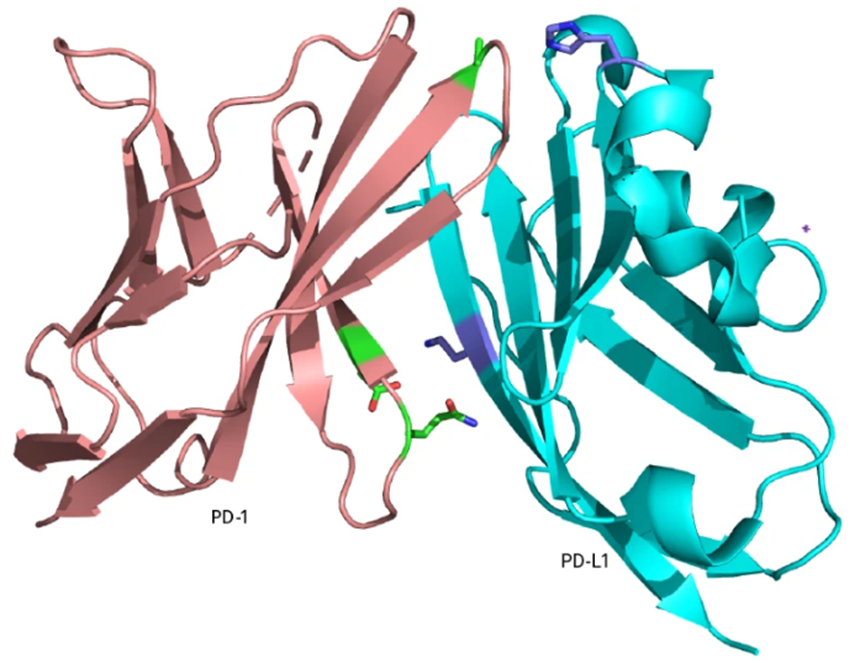
PD-1 (左)与配体PD-L1共价结合。图片来源: Q. Li et al. Cell 182, 85–97 (2020)
与此同时,北京大学的化学和合成生物学家Tao Liu和他的团队一直在将遗传扩展应用于细胞和基因治疗。在 2021 年发表的一项研究中,Liu和他的团队报告了工程细胞,在合成氨基酸 O-甲基酪氨酸存在下,可表达胰岛素[6]。当植入糖尿病小鼠体内时,研究人员可以通过控制它们在动物食物中分配多少O-甲基酪氨酸来控制动物的血糖水平。
扩展应用
除了编码新的氨基酸外,扩展的遗传密码还可以应用于遗传隔离。由于20种氨基酸具有61个密码子,遗传密码是冗余的,这意味着多个密码子编码相同的氨基酸。遗传密码几乎是普遍适用的。通过将给定密码子的所有实例替换为具有相同意义的密码子,并去除使用原始密码子的机制,研究人员可以使细胞对外来DNA(包括病原体)产生有效的免疫力。
Chin及其团队在2022年的一项研究中展示了这一概念;他们创造了一种大肠杆菌突变体,其中六个丝氨酸密码子中的两个被重新分配为编码其他氨基酸的密码子,然后删除了识别原始丝氨酸密码子的tRNA。他们进一步调整了系统,以确保病毒无法使用它们自己的tRNA(如果有的话)。结果发现,这些细胞对来自其他细菌的水平基因转移以及病毒感染具有抵抗性[7]。哈佛大学的合成生物学家George Church领导的另一个团队在今年早些时候发表的一篇论文中应用了类似的“遗传防火墙”方法,创造出抗噬菌体的细菌[8]。
研究人员还可以利用改变过的遗传密码来创建聚合物。2021年,Chin的团队破解了遗传密码,在大肠杆菌中合成了短聚合物,甚至是一种称为大环的人工环状结构[9]。现在,Chin希望进一步推动这项技术,创建能够大规模合成全新聚合物(如塑料)的细胞工厂。与蛋白质一样,塑料由长链的单体组成。然而,虽然遗传密码规定了蛋白质中氨基酸的顺序,但对于人工聚合物来说,并不存在类似的系统。
Chin表示:“如果我们能够以与蛋白质相同的方式编码扩展的建构单元,那么我们就可以将细胞转化为用于合成从新药到材料等各种聚合物的生产的活体工厂。”(Chin在英国Saffron Walden创立了一家名为Constructive Bio的公司,以推动这一目标。)
突破边界
除了密码子重新分配外,研究人员还可以通过将核酸密码子表从四个碱基扩展到六个碱基来增加可用的蛋白质构建单元的数量,从而将可能的三联密码子数量增加到216个。2014年,当时在斯克里普斯研究所的生物化学家Floyd Romesberg领导的一个团队报告称,他们创造了一种带有六碱基遗传密码的细菌株,且能够成功复制[10]。随后,该团队证明了这些细胞可以利用扩展的DNA产生含有非标准氨基酸的蛋白质[11]。
另一种方法是将密码子的长度从三个碱基扩展到四个碱基,从而将可能的密码子数量增加到256个。这需要修改多个翻译机制的组成部分,包括核糖体。Chin及其团队利用这种策略,在包含传统的三碱基密码子的转录本中,将四种非标准氨基酸整合至大肠杆菌中[12]。其他研究人员一直在探索是否可能创建一个全四碱基的遗传密码。
一些研究人员正在尝试更极端的改变。这些改变包括骨架修饰——创造所谓的β-或γ-氨基酸(而不是自然界中的α-氨基酸),或者是标准氨基酸的镜像异构体。使用这两种类型的构建单元构建的聚合物可能非常稳定,因为典型的蛋白质降解机制无法识别它们。
但是现有的翻译机制包括将氨基酸连接到tRNA上的氨酰-tRNA合成酶并不识别外源氨基酸。2006年,东京大学的化学生物学家Hiroaki Suga利用一种基于RNA的催化剂“flexizyme”(可催化蛋白质的合成)开发了一项解决方案:将氨基酸与tRNA连接起来[13]。
Suga专注于体外合成聚合物,因为它在他能够进行修改方面提供了最大的灵活性。他的团队利用flexizyme将多达11种非标准氨基酸与12种标准氨基酸结合在一个大环中。Suga共同创立了位于日本川崎的生物技术公司PeptiDream,使用相同的技术进行药物研发。
然而,这项工作同样也是体外实验;将修饰的氨基酸应用于细胞中的蛋白质合成仍然是一个挑战。尽管在少数情况下,科学家们已经能够将这些独特的氨基酸整合到蛋白质中,但需要采取额外的步骤来提高该过程的效率。除了找到可以在体内与这些氨基酸一起工作的合成酶外,多数情况下,科学家们还必须设计一种可以加工这些新氨基酸的核糖体 - 同时仍然履行其常规的职责。
在最近的两项研究中,加州大学伯克利分校的化学和合成生物学家Alanna Schepartz和她的团队报告了解决这些问题的步骤。在其中一篇论文中,他们描述了一种来自古细菌嗜甲烷菌的合成酶,它可以接受非α氨基酸,并且与大肠杆菌具有生物正交性(bio-orthogonal)。在另一项研究中,研究小组报告了一种计算技术,用于筛选可以有效加工独特骨架单体的大肠杆菌核糖体。研究人员表示,这将有助于识别最有可能与现有核糖体相容的非α氨基酸[15,16]。
其他小组正在努力开发一种核糖体,可以从头开始产生具有外源氨基酸的蛋白质。去年十月,中国杭州西湖大学的研究人员报道了一种镜像RNA聚合酶,可以合成产生镜像核糖体所需的所有RNA分子[17]。尽管要使镜像核糖体成为现实还需要更多步骤,但创建这样的分子将是使用翻译机制制造镜像蛋白质的重要一步。
其他研究人员正在努力重新编程核糖体以产生具有碳 - 碳键的聚合物,而不是他们通常锻造的氮 - 碳酰胺键来连接氨基酸。
如果这些策略得以实现,它们将赋予科学家对新聚合物的巨大合成能力。没有人知道这种具有与蛋白质相同长度和序列的合成聚合物会有什么特性,“因为从未制造过这样的分子”,Schepartz说。但她和其他人希望尽快实现这一目标。“对于这个领域来说,这是一个非常激动人心的时刻。
阅读原文内容:
https://doi.org/10.1038/d41586-023-01980-4
参考文献:
[1] Noren, C. J. Anthony-Cahill, S. J., Griffith, M. C. & Schultz, P. G. Science 244, 182–188 (1989).
[2] Wang, L. Brock, A., Herberich, B. & Schultz, P. G. Science 292, 498–500 (2001).
[3] Maywood, E. S. et al. Proc. Natl Acad. Sci. USA 115, E12388–E12397 (2018).
[4] Li, J. C., Liu, T., Wang, Y., Mehta, A. P. & Schultz, P. G. J. Am. Chem. Soc. 140, 15997–16000 (2018).
[5] Li, Q. et al. Cell 182, 85–97 (2020).
[6] Chen, C. et al. Nature Chem. Biol. 18, 47–55 (2022).
[7] Zürcher, J. F. et al. Science 378, 516–523 (2022).
[8] Nyerges, A. et al. Nature 615, 720–727 (2023).
[9] Nyerges, A. et al. Nature 615, 720–727 (2023).
[10] Malyshev, D. A. et al. Nature 509, 385–388 (2014).
[11] Zhang, Y. et al. Nature 551, 644–647 (2017).
[12] Dunkelmann, D. L., Oehm, S. B., Beattie, A. T. & Chin, J. W. Nature Chem. 13, 1110–1117 (2021).
[13] Murakami, H., Ohta, A., Ashigai, H. & Suga, H. Nature Methods 3, 357–359 (2006).
[14] Murakami, H., Ohta, A., Ashigai, H. & Suga, H. Nature Methods 3, 357–359 (2006).
[15] Fricke, R. et al. Nature Chem. https://doi.org/10.1038/s41557-023-01224-y (2023).
[16] Watson, Z. L. et al. Nature Chem. https://doi.org/10.1038/s41557-023-01226-w (2023).
[17] Xu, Y. & Zhu, T. F. Science 378, 405–412 (2022).
往期精品(点击图片直达文字对应教程)










 研究人员通过改变遗传密码,成功在细胞中引入非天然氨基酸,扩展了蛋白质的构建单元,这在药物开发和材料科学中有广泛应用。这种方法允许科学家操控蛋白质功能,创建更稳定的酶,甚至控制细胞行为。此外,这些进展也可能导致新型聚合物的合成,推动生物技术的革新。
研究人员通过改变遗传密码,成功在细胞中引入非天然氨基酸,扩展了蛋白质的构建单元,这在药物开发和材料科学中有广泛应用。这种方法允许科学家操控蛋白质功能,创建更稳定的酶,甚至控制细胞行为。此外,这些进展也可能导致新型聚合物的合成,推动生物技术的革新。














































 1343
1343

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










