通讯点击蓝字 关注我们
最全可视化集合工具EVenn使用手册

iMeta主页:http://www.imeta.science
研究论文
● 原文链接DOI: https://doi.org/10.1002/imt2.184
● 2024年4月11日,中国中医科学院陈同、黄璐琦和中国农科院深圳基因组所刘永鑫等团队在iMeta在线联合发表了题为 “Visualizing set relationships: EVenn's comprehensive approach to Venn diagrams” 的文章。
● 本文概述了EVenn的各种应用,包括韦恩图的代表性结果解读、数据中心管理数据,多个案例演示。通过这些功能,使EVenn成为一个用户友好的集合关系探索工具,为深入探索多组学数据提供强大的支持。
● 第一作者:杨莓
● 通讯作者:陈同(chent@nrc.ac.cn)、刘永鑫(liuyongxin@caas.cn)、黄璐琦(huangluqi01@126.com)
● 主要单位:天津中医药大学中医药研究院、中国中医科学院中药资源中心道地药材品质保障与资源持续利用全国重点实验室、中国农业科学院深圳农业基因组研究所
亮 点
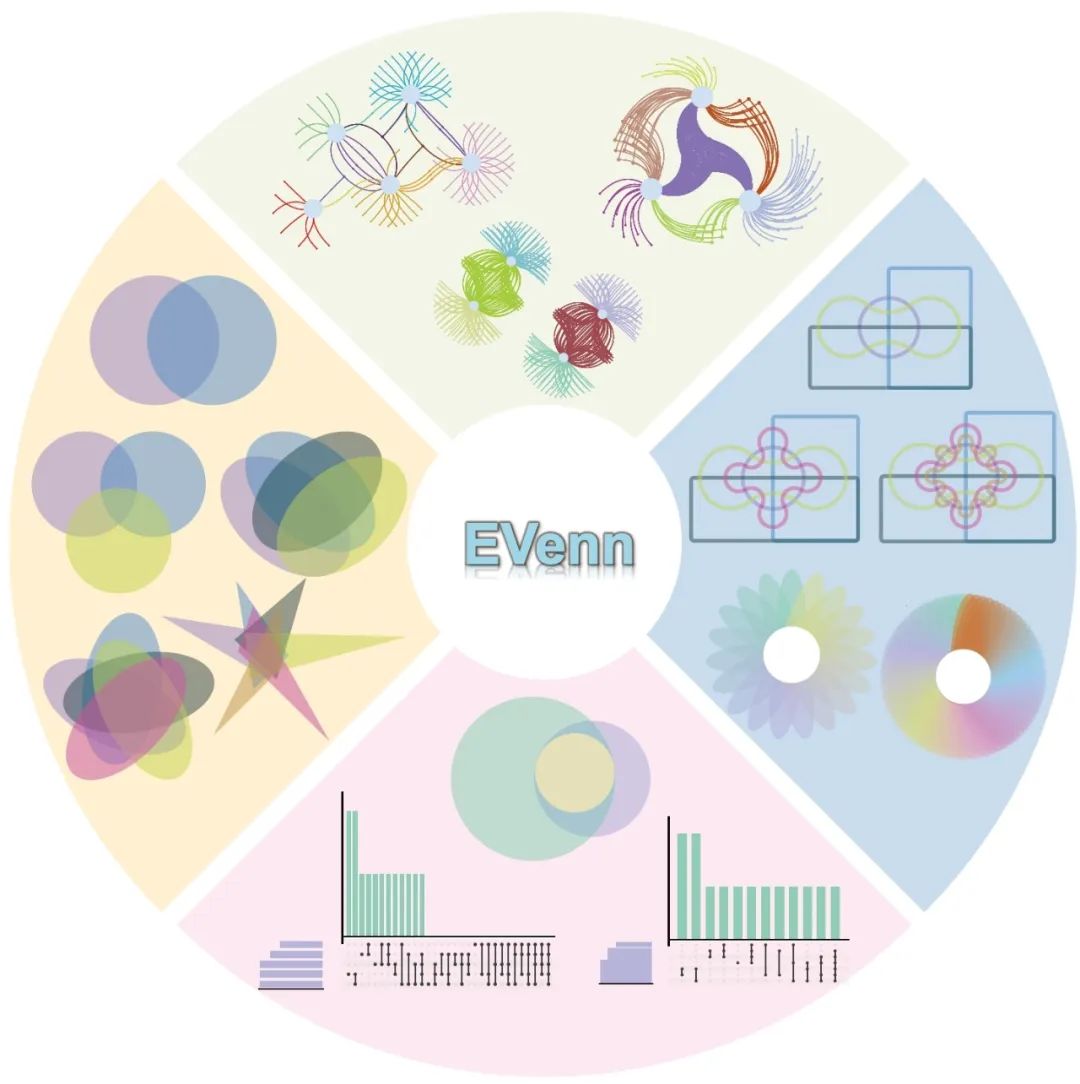
● 全面的韦恩功能:EVenn提供了一个统一的平台,包含多种韦恩图工具,从交互式图到网络图,满足了代谢组学、基因组学、转录组学和蛋白质组学等多组学数据分析的复杂需求;
● 高效的数据探索:EVenn的数据中心支持标准化的数据格式,简化数据上传和分析过程。本文通过代表性结果和详细案例展示其实用性;
● 用户友好的界面:EVenn拥有易用的界面,简化了各种韦恩图、欧拉图、UpSet图的生成过程。EVenn是研究人员解读多组学数据的好用工具。
摘 要
韦恩图作为一种展现集合关系的可视化方式,凭借其易于解读的特性,被广泛应用于代谢组学、基因组学、转录组学和蛋白组学等多个学科领域,其实用性毋庸置疑。然而,由于缺乏标准化的数据格式以及需要在不同平台间切换以生成适合不同数据的多种韦恩图,这增加了操作的复杂性。为了解决这些挑战,我们推出了多功能EVenn平台,提供了一个统一的界面,以高效地探索和可视化各种的韦恩图及其变体展示。EVenn(http://www.ehbio.com/test/venn)提供了标准化的数据上传格式,简化了数据上传过程,增强了多模块分析的能力。本文概述了EVenn的各种应用,包括韦恩图的代表性结果解读、数据中心管理数据,多个案例演示。通过这些功能,使EVenn成为一个用户友好的集合关系探索工具,为深入探索多组学数据提供强大的支持。
视频解读
Bilibili:https://www.bilibili.com/video/BV13M4m1X7gf
Youtube:https://youtu.be/ZZXQiCYFvDM
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
近年来,随着多组学技术的迅速发展,生成的数据日益增加。研究人员面临着如何处理和可视化如此庞大的多组学数据以获得对生物过程有意义的见解。复杂数据集的整合与可视化已成为多组学研究中日益重要的方面。分析生成的数据集包括差异代谢物、中草药、传统中医证候、差异表达基因、蛋白质与DNA/RNA的结合区域、微生物群落的特异性以及基因本体(GO)相似性。韦恩图被用来展示数据集之间的交集和差集,常用工具包括ImageGP、ComplexHeatmap、ggVennDiagram、Wekemo Bioincloud等。
本文全面介绍了EVenn的多种韦恩图和各种工具使用方法。EVenn平台提供了数据中心、交互式韦恩图、交互式爱德华图、欧拉图、UpSet图、花瓣图、交互式花瓣图、韦恩网络、韦恩差异显著性分析和韦恩计算等功能。这些功能被设计在不同的选项页面中,每个选项页面都包含了示例数据集,以便快速了解工具。EVenn允许跨多个数据集进行分析和可视化,有效地探索和解释复杂的多组学数据蕴含的关系。我们展示了EVenn中每个工具对特定数据集分析的适用性,阐明了各种韦恩图的含义,并概述了数据的格式和上传方式。此外,我们通过描述不同组学的案例展示了EVenn的多功能性和其在多数据分析中的实用性。EVenn平台为从事多组学分析的研究人员提供了易用的工具,促进了新见解的探索。
EVenn界面和代表性结果
EVenn平台的主界面提供了十个模块:数据中心、交互式韦恩图、交互式爱德华图、欧拉图、UpSet图、花瓣图、交互式花瓣图、韦恩网络、韦恩差异显著性分析以及韦恩计算模块。其中交互式韦恩图、交互式爱德华图、欧拉图、UpSet图、花瓣图及交互式花瓣图,均可用于比较不同组织的差异代谢物、不同组织或条件下的基因表达谱、不同样本中的蛋白质组成、不同疾病或条件下的生物标志物、不同药物处理组与对照组之间的筛选结果,以及不同样本中的菌群共存(OTUs)。韦恩网络图对于分析基因互作、蛋白质-蛋白质互作和代谢物互作网络特别有价值。例如,它能在网络图中展示富集的KEGG通路或GO条目中基因的共存情况。此模块同样适用于转录组、基因组及其他组学数据的分析。以下,我们提供了详细解释,以阐明每个模块生成的代表性结果图及其适用条件。
交互式韦恩图/爱德华图
EVenn的交互式韦恩图具有多重用途,允许用户在标准韦恩图和爱德华图之间无缝切换,同时便于探索和绘制多达6组数据。标准韦恩图通常用于比较2到6组实验数据(图1A),这里使用颜色编码来表示每组的特有和共有元素。例如,紫色(a,b,c)代表每组特有元素,蓝色(d,e,f)表示两组之间的共有元素,黄色区域(g)表示三组共同存在的元素。
相比之下,交互式爱德华图在集合多时能更清晰地展示各个部分,特别是在涉及5到6组的情况下(图1B)。以三组为例,其中a、b和c表示特有元素的区域,d、e和f代表两组中的共有元素区域,g代表三组中的共有元素。
增强可视化的欧拉图
相较于韦恩图,欧拉图在数据允许的情况下省略了空交集区域,从而提升了多个集合可视化的精确性,有更好地视觉效果和呈现力。欧拉图通常用于2至3组实验数据,生成圆圈的大小同集合中元素数目成正相关,交集区域的大小与共享元素的数量成正比。这一特性清晰地描述集合之间的部分包含、完全包含及完全排除关系(如图1C所示)。
在图1C所展示的三个集合例子中,每个圆圈的大小表示其各自集合中元素的数量,圆圈越大表示元素数量越多。绿色(a)与紫色(b)代表各自集合的特有元素,而重叠区域(c)代表两个集合之间共有的元素。此外,黄色区域(d)表示了三个集合共有元素。欧拉图有效地展示出集合之间的包含关系,为集合交互提供了更直观地视觉化表现。
UpSet图:一个强大的集合交集分析工具
UpSet图作为一个创新的可视化技术,专为集合交集的定量分析而设计,能够处理包含3至40个集合的复杂数据集。它通过两种展示模式,巧妙地可视化了不同的集合组合关系。如图1D所示,该图有三个主要组成部分:一个水平条形图表示每个集合中的总元素数量,一个垂直条形图指示对应交集中的元素数量,以及一个带有连接点的矩阵,勾勒出集合之间所有的关系类型。
在这个例子中,单独一个黑色点代表每个集合中的特有元素,而有线连接的黑色点象征着集合之间共有的元素。UpSet图全面展示了集合之间的关系,其中两个黑点的连接表示两个集合共有的元素,三个黑点的连接表示三个集合共有的元素,四个黑点的连接则表示四个集合共有的元素,以此类推。
花瓣图:揭示多组数据集的复杂关系
当处理超过十组的复杂数据集时,传统的可视化方法如韦恩图、欧拉图和UpSet图会难以应对。花瓣图是平衡解读性与信息丰富度的一种可视化方式。图1E的左侧展示了一个包含19个集合的花瓣图,而右侧是包含70个集合的花瓣图。
在图1E中,中心圆圈(a)代表所有集合共有的元素。围绕它的每个椭圆(b)表示每个集合相比于其它所有集合中特有元素的数量,或者是每个集合减去共有元素后的元素,这取决于具体的参数设置。
韦恩网络:揭示集合间的相互联系
韦恩网络拓展了标准韦恩图的展示边界,不仅是展现交集和并集的关系,而且可以展示出具体的元素。它巧妙地将每个集合指定为一个大的父节点,每个元素指定为一个小的子节点,并通过边将各个元素也就是子节点连接至其相应的父节点,来描绘集合内和集合间的关系。韦恩网络布局的灵活性在图1F中得到展示,韦恩网络会根据不同的数据类型呈现出相应的结构形态,已更好展现数据内集合之间的固有关系。
在图1F中,4个集合和5个集合的网络图揭示了它们元素之间的复杂关系。每个元素,由一条边连接表示其是各集合的特有元素。两条连接边线表示两个集合的交集,而三条边线代表三个集合的交集。韦恩网络展示了数据集内外相互连接的视觉层次。
韦恩图、UpSet图与韦恩网络三者之间的对应关系
在图1G中,我们借助一个三集合场景,展示了韦恩网络、韦恩图和UpSet图之间的对应关系以更好地理解这三种展示形式。每张图通过相应的字母标记一致的元素区域,确保了对这三种可视化之间关系的清晰理解。字母和颜色的一致性便于轻松识别同类型的关系组合:黄色(g)表示三个集合共有的元素,而蓝色(d、e、f)表示了两个集合共有的元素。与此形成对比的是,紫色(a、b、c)表示每个集合独有的元素。

图 1. EVenn的代表性结果示意图
(A)2至6组集合的交互式标准韦恩图;(B)2至6组集合的交互式爱德华图;(C)2至3组集合的欧拉图;(D)显示空交集和不显示空交集的UpSet图;(E)19组和70组集合的花瓣图;(F)4至5组集合的韦恩网络图;(G)韦恩图、UpSet图和韦恩网络图三者之间的对应关系图。
EVenn数据中心简化输入数据
EVenn的数据中心使用标准化的两列矩阵格式来简化输入过程,适用于韦恩图、欧拉图、UpSet图、花瓣图、交互式花瓣图、韦恩网络和韦恩计算工具。这种用户友好的格式,确保所有工具都可兼容,以第一行作为标题行,第一列是元素(例如,基因或OTUs),第二列对应各自的集合,一个TAB键用作列分隔符。准备这种输入矩阵非常简单,可以通过Excel、其他兼容的文本编辑器或简单程序完成。数据中心提供了两种文件上传模式—本地文件上传和直接粘贴数据上传。这种灵活的数据输入方式和标准化的数据格式,提升了用户体验,并加速了从数据准备到深入分析的过程。
上传文件是个很简单的过程,虽说是 5 步,但实际不复杂。首先拖动或点击文件以启动上传(图S1,步骤1)。其次为文件赋予独一无二的名称,以便轻松识别(图S1,步骤2)。所上传文件的内容将自动充填至指定文本区域(图S1,步骤3),用户可浏览及修改数据。最后点击“提交”按钮完成上传过程(图S1,步骤4)。一旦完成,文件将在文件选择界面中显示(图S1,步骤5),在打开每个工具时可以直接选择这里上传的文件进行分析和展示。
另一种数据输入的方法是将数据直接粘贴至数据中心的文本域。首先输入文件名以便标识(图S2,步骤1),然后将数据矩阵粘贴至指定文本区(图S2,步骤2)。点击“提交”完成保存过程(图S2,步骤3)。上传的文件也会在文件选择器中显示(图S2,步骤4)。
为有效管理上传文件,数据中心允许最多保存五个文件。为了加入新文件,可在“提交”按钮下方的下拉菜单中有选择地删除旧文件后再保存(图S2,步骤5)。需留意,这些文件存储在您本地的网页浏览器,并未上传至我们的服务器。因此,关闭您的网页浏览器有可能导致这些上传文件的丢失,且无法从您电脑的其他网页浏览器访问这些文件。
数据智能参数限制
为了提升用户体验并防止因输入不准确或操作失误引起的错误,EVenn引入了数据智能参数限制。例如,欧拉图支持两种输入矩阵类型,并根据数据类型的不同,条件性地启用或禁用某些可设置参数。在双列模式下,调整仅限于包含所有元素的列和包含所有集合信息的列。此外,在让用户确定列名时采用一个用户友好的下拉框取代开放式输入框,以减少打字错误并通过限制用户的选择范围更好地提示用户的操作。这些优化显著提高了EVenn界面的易用性,确保了更流畅的使用体验。
案例展示
案例一:交互式韦恩图展示差异代谢物
韦恩图以直观的方式比较和分析不同条件或组织中的代谢物、基因和OTUs的重叠情况。例如,以研究茯苓处理前后不同部位的代谢物变化为例(表S1)。交互式韦恩图支持三种不同的数据输入方法:使用两列格式上传或粘贴数据(默认模式)(图S3,步骤1),直接粘贴每个集合的元素(输入元素)(图S3,步骤2),以及手动输入每个交集的计数(输入数字)(图S3,步骤3)。在此,我们展示了使用粘贴两列矩阵输入方法生成交互式韦恩图的简便过程,有效地展示了茯苓5个部位的差异代谢物。该方法包括下面一系列步骤:
1. 数据矩阵输入:将差异代谢物数据矩阵以两列格式粘贴至文本区域(图S4,步骤1)。第一列是代谢物名称,第二列是其检测部位信息。
2. 集合选择:按照顺序选择FS1、FS2、FS3、FS4及FS5集合进行分析(图S4,步骤2)。
3. 生成交互式韦恩图:上述两个步骤完成后(其实每选择一步,都可以看到韦恩图在当前状态下的展示),将生成一个交互式韦恩图,展示茯苓5个部位差异代谢物的交集情况(图S4,步骤3)。
4. 重排集合顺序:通过点击并拖动集合(FS1、FS2、FS3、FS4及FS5),将集合调整到理想位置(这里也可以删除集合)(图S4,步骤4)。
5. 自定义集合颜色:使用位于每个集合名称旁的颜色选择器(图S4,步骤5),为5个集合自定义颜色,分别为它们指定黄色、粉色、绿色、紫色及蓝色。
6. 全局配置参数:点击配置按钮激活全局配置参数(图S4,步骤6)。相应选择“经典模式”或“爱德华模式”。字体、字号、输入列表统计、切换按钮及交集计数是否展示都保持默认参数。
7. 访问特定交集数据:点击韦恩图中的计数数字,下方文本区域显示该交集的差异代谢物名称(图S4,步骤7)。标题显示差异代谢物的所属的特定集合区域。
8. 下载选项:通过点击下载按钮,获取差异代谢物PNG和SVG格式韦恩图,以及差异代谢物交集列表CSV格式文件(图S4,步骤8)。
图2A和B分别展示了茯苓5个部位的标准韦恩图和爱德华图。可见茯苓5个部位(FS1、FS2、FS3、FS4及FS5)共有119个处理后出现一致变化的差异代谢物。此外,FS1、FS2、FS3、FS4及FS5分别有2、5、1、0、4个处理后独特变化的差异代谢物。进一步地分析发现:FS1和FS2、以及FS2和FS3之间无相同的差异代谢物;FS3和FS4共有1个差异代谢物,而FS4和FS5共有7个差异代谢物。韦恩图清晰地描绘了茯苓处理前后5个部位差异代谢物的变化关系。
案例二:欧拉图展示差异表达基因关系
通过分析文献中抗癌药物处理后不同类型细胞中差异基因关系,来展示欧拉图工具的使用。欧拉图工具支持两种类型数据输入方法:两列模式矩阵和计数矩阵。利用该工具生成差异表达基因欧拉图的具体步骤如下:
1. 数据整理:整理文献中每种类型细胞的差异表达基因数量并呈现在表S2中。
2. 选择输入类型:选择交集-计数矩阵方法(图S5,步骤1)。
3. 数据输入:将差异表达基因的两列计数矩阵粘贴到交集-计数矩阵框中(图S5,步骤2)。第一行是标题行,第一列是所有集合的交集部分,第二列是每个交集的元素数量。此外,第一列中的“&”符号代表交集,TAB键作为列分隔符。
4. 基本参数选择:参数“Intersection Column containing all intersections”选择“交集”,参数“Column containing counts for each intersection”参数选择“计数”(图S5,步骤3)。
5. 其他类型参数:此处,布局和颜色以及图片属性参数使用默认设置(图S5,步骤4)。
我们对复杂的颜色参数设置进一步说明。这里提供两种定义颜色的方法:为每个集合手动设置颜色和手动设置颜色向量(颜色集)(图S6A)。
为每个集合手动设置颜色:设置每个集合的颜色。选择颜色时,请确保点击颜色选择器中的“确定”按钮(图S6B)。此参数的使用与手动设置颜色向量(颜色集)参数互斥。启用此参数前,请确保清除手动颜色设置向量(颜色集)的值。如果您想为每个集合指定颜色,应选择相同数量的颜色(图S6B)。如果选取的颜色数量少于集合数,程序将自动生成中间色以使其与集合的数量相等。
手动设置颜色向量(颜色集):为所有集合选择颜色向量。此参数与为每个集合手动设置颜色参数互斥(图S6C)。首先请清除为每个集合手动设置颜色参数的值,以启用此参数。
6. 生成欧拉图:点击“提交”按钮(图S5,步骤5),在绘图区显示差异表达基因的欧拉图(图S5,步骤6)。右击图片可以保存成PNG格式,也可以通过点击“下载PDF”按钮,将图片保存成PDF格式(图S5,步骤7)。
通过上述步骤生成了图2C,可以看出NCCIT细胞特有的差异表达基因最多为666个,在图中占据了最大的面积。相比之下,NCCIT、Erms和GBM之间的共有差异基因数量最少,仅有15个基因,因此占据了图中最小的面积。采用欧拉图,与文献中原有的可视化方式相比提高了差异表达基因数量展示的精准性。
案例三:UpSet图展示ChIP-seq数据
ChIP-seq是研究表观组学中广泛应用的技术,用于探索蛋白质与DNA之间互作和了解基因组的功能元件。在本案例研究中,我们对小鼠胚胎干细胞(ESC)中5个转录因子(Esrrb、Klf4、Med1、Nanog、Sox2)和2个组蛋白修饰标记(H3K27ac与H3K4me1)的ChIP-seq数据进行了分析,旨在确定它们共有和特有的结合位点。这些ChIP-seq数据源自Whyte等人的研究 (表S3)。UpSet图的生成接受两种输入格式:两列矩阵和二进制矩阵格式。生成ChIP-seq UpSet图的步骤如下:
1. 数据集预处理:利用ImageGP平台将表S3中Sox2、Nanog、Klf4、Esrrb、H3K27ac、Med1、H3K4me1及其结合基因数据转换成两列格式,如表S4所示(https://www.bic.ac.cn/BIC,图S7,步骤1)。
2. 选择数据输入类型:选择两列矩阵格式输入数据。
3. 数据输入:以两列格式粘贴转录调控因子和靶向基因数据,第一列是靶向基因,第二列是转录调控因子(图S8,步骤1)。
4. 基本参数选择:参数“将第一行作为标题行”,设置为“是”,而参数“保留空相交”设置为“假”(图S8,步骤2)。
5. 生成UpSet图:点击“提交”按钮(图S8步骤3)即可得到UpSet图。然后,点击“下载PDF”按钮(图S8步骤4)即可下载PDF格式的图片。
如图2D所示,UpSet图展示了7个调控因子之间的共有和特有结合或修饰的基因,其中有3411个基因受到7个转录调控因子的共同调控,此外,还有475个基因仅与Sox2、Nanog、Klf4、Esrrb、Med1和H3K4me1结合。值得注意的是,这七个转录调控因子各自并没有特有的结合基因。通过UpSet图可以有效地可视化它们与结合位点之间的复杂关系。
案例四:交互式花瓣图展示微生物群落的特异性
探究微生物群落特异性对于理解生态角色、疾病诊断、揭示进化过程以及探索关键菌群至关重要。交互式花瓣图作为一种宝贵的工具,能够展示超过十个微生物群落组中的共有和特有的元素。通过EasyAmplicon流程获取不同组(敲除组、野生型及过表达组)的操作分类单元(OTU)数据。生成微生物组交互式花瓣图,请遵循以下步骤:
1. 数据预处理:使用ImageGP平台将表S5的数据转换为两列格式(转换后的数据见表S6)。
2. 两列模式数据矩阵输入:将微生物OTUs数据粘贴到数据框的指定文本区域(第一列是元素,即OTUs或ASVs,第二列是集合名称,即样本组)。
3. 选择参数:为元素选择“OTUID”列,为集合信息选择“Group”列。对于椭圆中展示的数值的参数,分别选择两种模式。其余参数保持默认设置。
4. 生成交互式花瓣图:点击“提交”按钮生成交互式花瓣图。通过点击图片中的数字可以获取各个组的共有和特有元素。
5. 下载SVG:将图片以SVG格式导出。
图2E展示了18个样本组共有413个相同OTUs。其中,WT3、OE1和KO3集合包含独特的OTUs,分别是ASV_942、ASV_1022和ASV_1201。相反,其余15个组缺乏独特OTUs。图2F也展示了18个微生物组中413个相同OTUs元素的存在,但是围绕中心圈的18个花瓣则代表在减去共有元素后每组的OTUs元素计数。运用两种不同花瓣图展示方法对于从多个角度分析OTUs在较多样品组中的相似性和差异性具有优势。
案例五:韦恩网络显示GO富集结果
为了展示韦恩网络可视化功能,我们通过IMP平台(https://www.bic.ac.cn/IMP)提供的GO/KEGG富集分析工具,获取了穿心莲的基因及GO数据。所获得的数据集,如图S10所示,包含了生物过程条目与其关联的基因。我们的目标是构建一个韦恩网络图,展示富集到的生物通路与基因的网络关系。具体操作如下:
1. 数据预处理:筛选所需的基因和GO数据,使用 ImageGP平台(https://www.bic.ac.cn/BIC)将数据转换成韦恩网络所需的两列格式(图S7,步骤2,表S7)。
2. 数据矩阵输入:将上面转换出的 2 列矩阵粘贴到文本域中,注意目标基因需要在第一列,GO条目需要在第二列(图S11,步骤1)。
3. 选择参数:设置颜色和展示模式两个参数可以自由选择。例如,我们选择了粉色、绿色、紫色和蓝色作为颜色参数(图S11,步骤2)。此外,先选择“显示所有元素”模式以全面展示数据,也可以选择“仅显示共有元素及特有元素”模式展示类似花瓣图中的数据统计(图S11,步骤3)。
4. 生成韦恩网络图:点击“提交”按钮后,生成韦恩网络图(图S11,步骤4)。
5. 优化韦恩网络图布局:为了优化韦恩网络图的清晰度和美观性,访问“工具菜单”(图S11,步骤5)并使用“首选布局”按钮(图S11,步骤6)。随后,点击工具按钮以显示左侧工具栏(图S11,步骤7),修改节点、边、布局参数。其中,边类型参数设置为“diagonalCross”,布局模式中布局计算算法设置为“barnesHut”,而其他设置保持默认值(图S11,步骤8-9)。
6. 韦恩网络的节点和边参数:我们提供了一个“编辑X节点”按钮,用于更改多个选定节点的属性,以及一个“编辑边”按钮,用于更改边的宽度和颜色(图S11,步骤10-11)。节点和边的颜色方案如图2G所示。
7. 下载韦恩网络图:点击“导出SVG”按钮,以SVG格式导出韦恩网络图(图S11,步骤 12)。
如图2G所示,IMPTAPA2N19692_1基因参与到四个关键生物过程:monocarboxylic acid biosynthesis、cellular amino acid metabolism、organitogen compound biosynthesis和multiple organismal enrichment process。基因IMPTAPA2N21328_1、IMPTAPA2N31674_1、IMPTAPA2N40577_1、IMPTAPA2N11277_1、IMPTAPA2N33800_1和IMPTAPA2N22367_1与monocarboxylic acid biosynthesis和multiple organismal enrichment process相关。此外,IMPTAPA2N33400_1特异性从属于organitogen compound biosynthesis过程。图2H描绘了四个不同生物过程中共有和特有基因的存在。

图2. EVenn案例展示
(A)茯苓5个部位处理前后差异代谢物的交互式韦恩图;(B)茯苓5个部位处理前后差异代谢物的交互式爱德华图;(C)抗癌药物治疗后三种细胞差异表达基因的欧拉图;(D)7个转录调控因子及其靶向基因ChIP-seq的UpSet图;(E)交互式花瓣图展示18个微生物组共有和特有OTUs数量;(F)交互式花瓣图展示18个微生物组共有及减去共有后的OTUs数量;(G)韦恩网络图展示穿心莲基因GO富集分析结果;(H)韦恩网络图展示穿心莲四个生物过程富集分析结果(仅展示共有和特有基因)。
结 论
韦恩图数据分析的复杂性源于数据格式的不一致和缺乏统一的数据分析平台。本文全面概述了EVenn平台内各个模块的功能与操作,包括数据中心、交互式韦恩图、欧拉图、UpSet图、花瓣图、交互式花瓣图以及韦恩网络图。
在不同实验条件下比较代谢物、基因、基因组区域和微生物类群等数据,常需借助专门的可视化技术,包括交互式韦恩图、欧拉图、UpSet图、花瓣图和交互式花瓣图。其中,交互式韦恩图特别适用于比较多达6组实验数据,允许直观识别共性与差异。相比之下,欧拉图更适合比较2到3组实验数据,通过面积比例表示每组的数据大小。UpSet图可以比较3组至40组复杂的数据集。而花瓣图在评估10组以上数据的共有与特有元素时更常用,可以清晰地展示多组数据关系。此外,韦恩网络图是用网络形式展示集合关系的有效工具。它可以揭示多组实验之间的关系,展示复杂的交互模式。总之,选择哪种可视化方法取决于数据和研究目的。通过理解每个工具的功能,研究人员可以有效地解释和展示他们的结果。
代码和数据可用性
数据和脚本已在GitHub (https://github.com/Tong-Chen/venn-doc)和Gitee(https://gitee.com/ct5869/venn-doc/)发布。所有的补充材料(文本、图、表、中文翻译版本或视频)也可从线上获取。
引文格式:
Mei Yang, Tong Chen, Yong-Xin Liu, Luqi Huang. 2024. Visualizing set relationships: EVenn’s comprehensive approach to Venn diagrams. iMeta 3: e184. https://doi.org/10.1002/imt2.184
作者简介

杨莓(第一作者)
● 天津中医药大学,中药学在读博士生。
● 目前研究方向为中药基因组,转录组等数据分析和多组学工具开发。以第一作者或共同第一作者在iMeta,Nucleic Acids Research,Frontiers in Plant Science,Phytochemical Analysis等期刊发表论文5篇,副主编著作1部,参编著作1部。

陈同(通讯作者)
● 博士,中国中医科学院副教授。
● 研究方向涉及高通量数据分析、生物信息工具开发、合成生物学、表观组学等,在Cell Stem Cell (封面文章),Nucleic Acids Research,Nature communications, Protein & Cell, iMeta等高水平杂志以第一或通讯作者发表文章十余篇;开发在线绘图和分析平台 ImageGP、BIC、EVenn, 在线数据库平台IMP、CRAMdb、ETCM1/2、CMNPD、iLABdb、SoFDA 等,总计使用超 150 万人次;运营有十四万人关注的微信公众号和视频号《生信宝典》,分享有1400 多篇生物信息分析原创文章、教程和视频,阅读播放千万次。联合创办iMeta期刊,现为执行主编,致力于打造微生物和生物信息领域的国产高水平杂志。

刘永鑫(通讯作者)
● 中国农科院深圳基因组所研究员,iMeta期刊执行主编,宏基因组公众号创始人。
● 主要研究方向为微生物组方法开发、功能挖掘和科学传播,在Science、iMeta、Nature Biotechnology、Nature Microbiology等期刊发表论文60余篇,被引18000+次,入选全球Top 2%高被引科学家。主编《微生物组实验手册》专著,由300多位同行参与,共同打造本领域长期更新的中文百科全书。创办宏基因组公众号,16万+同行关注,累计阅读量超4千万,打造本领域最具影响的科学传播平台。为Nature Communications、Microbiome、ISME、NAR等70种期刊审稿200次。2022年联合发起iMeta期刊(ESCI收录),打造微生物组/生物信息领域国际顶刊,解决我国本领域期刊出版卡脖子问题,建立国际学术话语权体系,2024年6月即将获得本刊首个影响因子,预计IF~20。

黄璐琦(通讯作者)
● 中共党员,中国工程院院士。现任国家中医药管理局副局长、党组成员,中国中医科学院院长。
● 任中国科协第十届全国委员会副主席,国家监察委员会第一届特约监察员,第十五届、十六届北京市人民代表大会代表。政协第十三届全国委员会常务委员。荣获全国优秀共产党员称号。从事中药资源学与分子生药学研究。提出和发展了“分子生药学”学科,揭示和阐明了道地药材形成理论,建立珍稀濒危常用中药资源五种保护模式,组织实施第四次全国中药资源普查,摸清我国中药资源本底。获国家科学技术进步二等奖5项、全国创新争先奖章、国家杰出青年科学基金资助、中国标准创新突出贡献奖、全国优秀博士学位论文指导教师等荣誉。主持研发我国首个具有完全自主知识产权的治疗新冠肺炎的中药新药,荣获全国抗击新冠肺炎疫情先进个人称号。以第一作者或通讯作者发表论文580余篇,包括BMJ,NAT COMMUN,PNAS,JACS等SCI论文300余篇。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底

3卷2期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、Scopus等数据库收录。
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science
高颜值免费 SCI 在线绘图(点击图片直达)
最全植物基因组数据库IMP (点击图片直达)
往期精品(点击图片直达文字对应教程)





































































 6317
6317

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










