做差异基因分析时经常会遇到有老师纠结是样品组A比样品组B还是样品组B比样品组A。每次我都是很诧异,这有区别吗?
这是一个典型的DESeq2输出结果,我们怎么知道他计算的是trt/untrt还是untrt/trt?
ID trt untrt baseMean log2FoldChange pvalue padj
ENSG00000152583 1885.248 80.835 983.042 4.546 1.219e-91 2.149e-87
ENSG00000189221 4366.392 416.725 2391.559 3.387 9.955e-61 8.779e-57
ENSG00000179094 1353.139 161.359 757.249 3.065 2.435e-54 1.432e-50
ENSG00000116584 1450.876 3033.977 2242.427 -1.064 3.957e-49 1.745e-45选2个基因做个例子就可以,ENSG00000152583的log2FoldChange是正值,这个基因在trt样本组表达高;ENSG00000116584的log2FoldChange是负值,这个基因在untrt样本组表达高;结果很明显了,是trt/untrt。log2FoldChange为正时表示基因在处理后上调,为负时表示基因在处理后下调。
如果反过来,如下(给log2FoldChange列都乘以了-1),log2FoldChange为正时表示基因在处理后下调,为负时表示基因在处理后上调。
ID trt untrt baseMean log2FoldChange pvalue padj
ENSG00000152583 1885.248 80.835 983.042 -4.546 1.219e-91 2.149e-87
ENSG00000189221 4366.392 416.725 2391.559 -3.387 9.955e-61 8.779e-57
ENSG00000179094 1353.139 161.359 757.249 -3.065 2.435e-54 1.432e-50
ENSG00000116584 1450.876 3033.977 2242.427 1.064 3.957e-49 1.745e-45其实就是描述方式不同。你可以在差异基因分析之前制定以哪个组做参考;如果没有指定或忘记了指定,结果正好又是反过来的,直接对log2FoldChange取反就可以。
另外还有一个问题,在之前几期课程也是常常被问起,log2 Fold change (有时简写为log2FC)是什么?初次不知道这个单词的含义没问题,如果不知道差异倍数就有点不好理解了。
首先看Fold change是什么?是差异倍数。怎么算的呢?正常计算是两个组的平均值的商,具体到上面的例子就是trt/untrt,如ENSG00000152583的fold change是1885.248/80.835=23.32217。
为什么会取log2呢?我们看下下面这张图。所有算出的小于0的Fold change转为了负数,大于0的Fold change还是正数。且上调两倍可转为log2FC=1,下调两倍可转为log2FC=-1,转换后的值上下调存在对称关系,更有利于查看、筛选和绘图。如常用筛选标准abs(log2FC)>=1可以获得差异倍数2倍的上下调基因 (log(2)==1; log(0.5)=-1)。
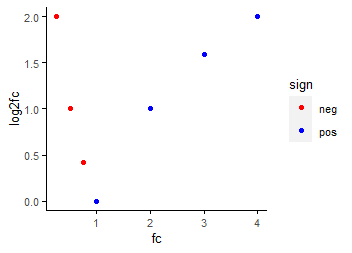
fc=c(seq(0.25,1,length.out=4), seq(1,4,length.out=4))
data = data.frame(fc=fc, log2fc=abs(log2(fc)))
data$sign = ifelse(data$fc<1,'neg','pos')
data
# devtools::install_git("https://gitee.com/ct5869/ImageGP")
library(ImageGP)
sp_scatterplot(data, xvariable = "fc", yvariable = "log2fc", color_variable = "sign", manual_color_vector = c("red","blue"))当然我们算出的log2FC跟DESeq2给出的不完全一致,是因为DESEq2做了进一步校正,但通常差别不大。
























 8209
8209

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










