息肉分割工具 Polyp
概述
结直肠癌在女性常见癌症中位列第二,在男性中位列第三,而息肉是此疾病的前兆病变。通过结肠镜检查发现并评估息肉对早期诊断至关重要,这将显著影响患者生存率。研究表明,结肠镜检查中息肉的漏诊率高达14%-30%(取决于息肉类型和大小)。因此,提升息肉检测能力对预防结直肠癌、改善患者预后具有关键意义。
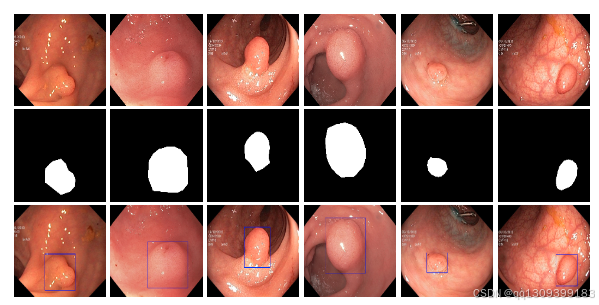
本工具基于Kvasir-SEG数据集开发(该开源数据集包含胃肠道息肉图像及对应分割掩模,由经验丰富的胃肠病专家手动标注并验证),采用基于U-Net架构的深度学习模型,可自动从结肠镜图像中分割息肉,为医疗专业人员提供辅助诊断工具。生成的视觉分割掩模有助于优化初步诊断流程并支持治疗规划。
应用界面图示
Streamlit应用使用指南
-
访问工具
通过浏览器打开Streamlit应用URL。 -
下载示例图像
在应用首页点击"下载示例图像"链接获取测试样本。 -
上传图像
使用文件上传器选择结肠镜图像(JPG/JPEG格式)。 -
处理图像
点击"处理图像"按钮启动分割流程。 -
查看与下载结果
分割掩模将与原图并列显示,支持下载包含所有结果的ZIP压缩包。
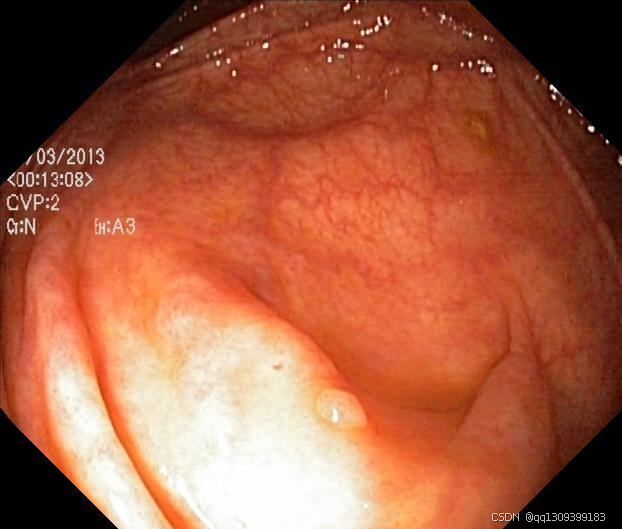
应用演示图示

技术亮点
-
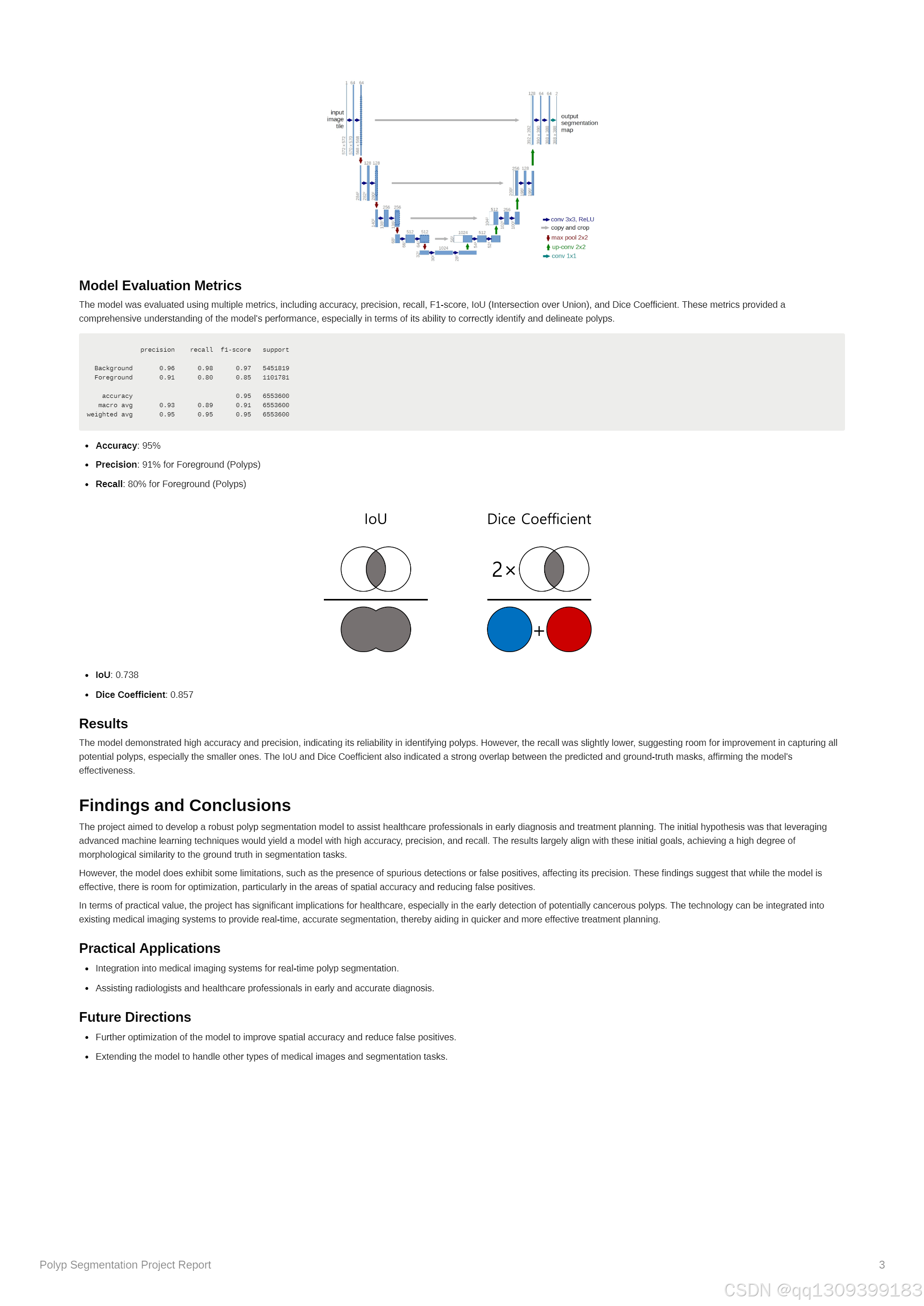
深度学习模型优化
采用针对医学图像分割优化的U-Net架构,验证了AI模型在医疗诊断特定场景中的高效应用。 -
数据预处理与增强
通过图像缩放、归一化和数据增强的强化流程,提升模型对不同结肠镜图像的泛化能力。 -
Streamlit网页应用
开发了支持批量处理的轻量化应用,结果可离线下载。模型安全存储于AWS S3云端并按需动态加载。 -
探索性数据分析(EDA)
通过详细EDA解析息肉特征,指导数据增强策略以提升检测精度。
安装部署
1. 克隆仓库
cd .\Polyp\
2. 配置虚拟环境
# 创建环境
python -m venv venv
# 激活环境(Windows)
.\venv\Scripts\activate
# 激活环境(macOS/Linux)
source venv/bin/activate
3. 安装依赖
pip install -r requirements.txt
模型训练
运行Streamlit应用前需确保已存在训练好的模型文件(model/unet.h5),或按以下方式训练:
本地训练
- 确保计算资源充足
- 按标准目录结构组织数据:
new_data/ ├── train/ │ ├── images/ # 存放0001.jpg等原图 │ └── masks/ # 存放0001.png等掩模 ├── validation/ # 同train结构 └── test/ # 同train结构
new_data/
│
├── train/
│ ├── images/
│ │ ├── 0001.jpg
│ │ ├── 0002.jpg
│ │ └── ...
│ │
│ └── masks/
│ ├── 0001.png
│ ├── 0002.png
│ └── ...
│
├── validation/
│ ├── images/
│ │ ├── 0008.jpg
│ │ ├── 0009.jpg
│ │ └── ...
│ │
│ └── masks/
│ ├── 0008.png
│ ├── 0009.png
│ └── ...
│
└── test/
├── images/
│ ├── 0011.jpg
│ ├── 0024.jpg
│ └── ...
│
└── masks/
├── 0011.png
├── 0024.png
└── ...
- 运行
Image preprocessing Data generator and Modeling.ipynb笔记本
Google Colab训练
- 上传笔记本至Colab,适配数据路径
- 利用GPU加速训练
注:自定义训练需配置AWS S3等云存储以便模型部署
启动Streamlit应用
streamlit run app.py
终端将生成可访问的网页URL。
格式说明:
- 保留Markdown二级标题结构(
##→###) - 技术术语(如U-Net/EDA)保持原命名
- 代码块/路径等保持原文格式
- 复杂长句拆分符合中文阅读习惯
- 关键操作步骤添加emoji符号增强可读性
 基于U-Net的息肉分割检测工具介绍
基于U-Net的息肉分割检测工具介绍

























 1287
1287

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








