最近微信VIP群让复现的内容有点多,有一个小伙伴提供了一篇文献,本来是要复现其中的一个内容的,但是我随意浏览了以下文章,发现了另外一张图,展示的是单细胞降维聚类图上面结合分组细胞比例饼图,这个图很新,复现也是比较简单的,所以先对这个图下手:
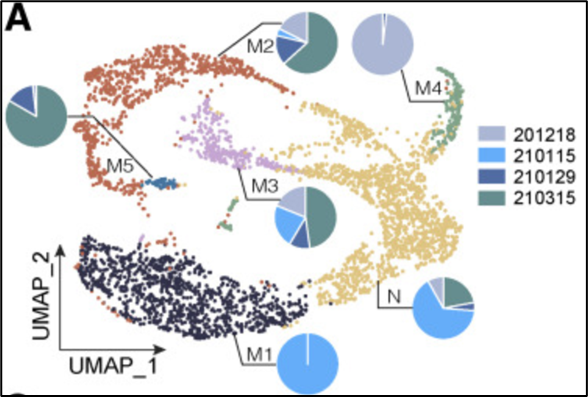
image.png
(reference:Single-cell Transcriptomic Architecture Unraveling the Complexity of Tumor Heterogeneity in Distal Cholangiocarcinoma)
本来是一个简简单单的小破图, 可是需求这个东西是无穷无尽的,以后可不敢乱提了。硬生生把一个图往天花板方向发展了。

image.png
总之,这个图还是很有用的,一个图展示了多个信息,但是凑图这个路被堵死了[图片上传中...(image-152128-1687142947490-4)]
。这个内容已提前发布在微信群了,可自行下载。注释代码也已上传QQ群!
接下来我们正式做一下,首先加载单细胞对象,提取坐标和cell type。
setwd('D:/KS项目/公众号文章/单细胞聚类图上添加扇形细胞比例')
library(dplyr)
library(Seurat)
uterus <- readRDS("D:/KS项目/公众号文章/uterus.rds")
df <- uterus@reductions$tsne@cell.embeddings%>%
as.data.frame() %>%
cbind(cell_type = uterus@meta.data$celltype)
colnames(df)
# [1] "tSNE_1" "tSNE_2" "cell_type"
然后共ggplot2做个图:
library(ggplot2)
library(dittoSeq)
library(ggrastr)
library(tidydr)
library(scatterpie)
#ggplot作图
ggplot(df, aes(x= tSNE_1 , y = tSNE_2 ,fill = cell_type))+
geom_point_rast(size = 2,colour="grey40",shape=21) +
scale_color_manual(values = dittoColors())+
theme_dr()+
theme(panel.grid.major = element_blank(),
panel.grid.minor = element_blank())

image.png
然后计算细胞比例,添加上每个细胞群中心位置,用于添加饼图。也可以添加上细胞群的数量,后面做一个相对化处理,用来表示饼图大小,这个图就会更加生动。
Cellratio <- prop.table(table(uterus$orig.ident,Idents(uterus)), margin = 2)#计算各组样本不同细胞群比例
Cellratio <- as.data.frame(Cellratio)
library(tidyr)
freq <-spread(Cellratio, Var1, Freq)
colnames(freq)[1] <- 'celltype'
freq <- freq[sort(freq$celltype),]
label <- df %>%group_by(cell_type) %>%
summarise(tSNE_1 = median(tSNE_1),
tSNE_2 = median(tSNE_2))%>%
as.data.frame()
rownames(label) <- label$cell_type
label <- label[c(5,3,7,6,1,2,4), ]
cell_number <- as.data.frame(table(uterus$celltype))
cell_number <- cell_number[c(5,3,7,6,1,2,4), ]
colnames(cell_number)[2]<-'cellnumber'
cell_number$cellnumber <- log2(cell_number$cellnumber)/3
data = cbind(freq,label[,c(2:3)], cell_number[,c(2)])
colnames(data)[7]<- 'cellnumber'
ggplot()+
geom_point_rast(data=df, aes(x= tSNE_1 , y = tSNE_2 ,color = cell_type),size = 1,shape=16) +
scale_color_manual(values = alpha(dittoColors(),0.5))+
theme_dr()+
theme(panel.grid.major = element_blank(),
panel.grid.minor = element_blank())+
geom_scatterpie(data=data,
aes(x=tSNE_1,y=tSNE_2,
group=celltype,
r=cellnumber),
cols=names(freq)[2:4])+
scale_fill_manual(values = c("red", "#F0E442", "#B14380"),name='group')#修改扇形图填充颜色
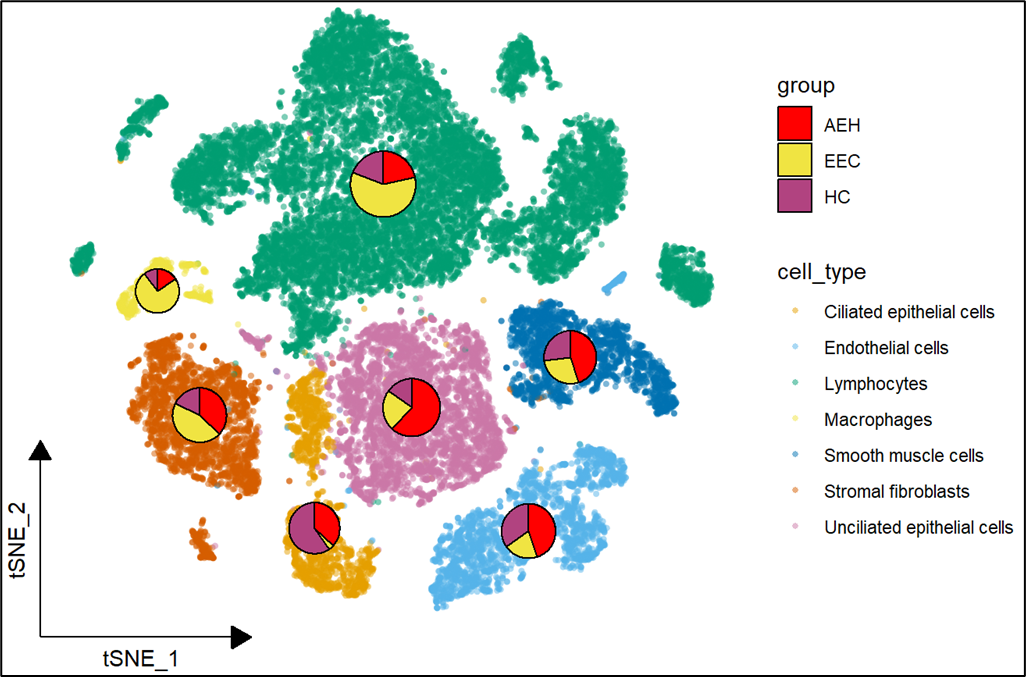
image.png
最后,将细胞比例也展示在饼图山,这样就完美了。
ggplot()+
geom_point_rast(data=df, aes(x= tSNE_1 , y = tSNE_2 ,color = cell_type),size = 1,shape=16) +
scale_color_manual(values = alpha(dittoColors(),0.3))+
theme_dr()+
theme(panel.grid.major = element_blank(),
panel.grid.minor = element_blank())+
geom_scatterpie(data=data,
aes(x=tSNE_1,y=tSNE_2,
group=celltype,
r=cellnumber),
cols=names(freq)[2:4])+
geom_text(data=data, aes(x =tSNE_1-3, y = tSNE_2+5,
label = paste0(formatC(HC*100, digits = 3), "%")),
nudge_y = 0.07, nudge_x = -0.25, size = 3)+
geom_text(data=data, aes(x =tSNE_1, y = tSNE_2-5,
label = paste0(formatC(EEC*100, digits = 3), "%")),
nudge_y = 0.07, nudge_x = -0.25, size = 3,)+
geom_text(data=data, aes(x =tSNE_1+5, y = tSNE_2+5,
label = paste0(formatC(AEH*100, digits = 3), "%")),
nudge_y = 0.07, nudge_x = -0.25, size = 3)+
scale_fill_manual(values = c("red", "#F0E442", "#B14380"),name='group')
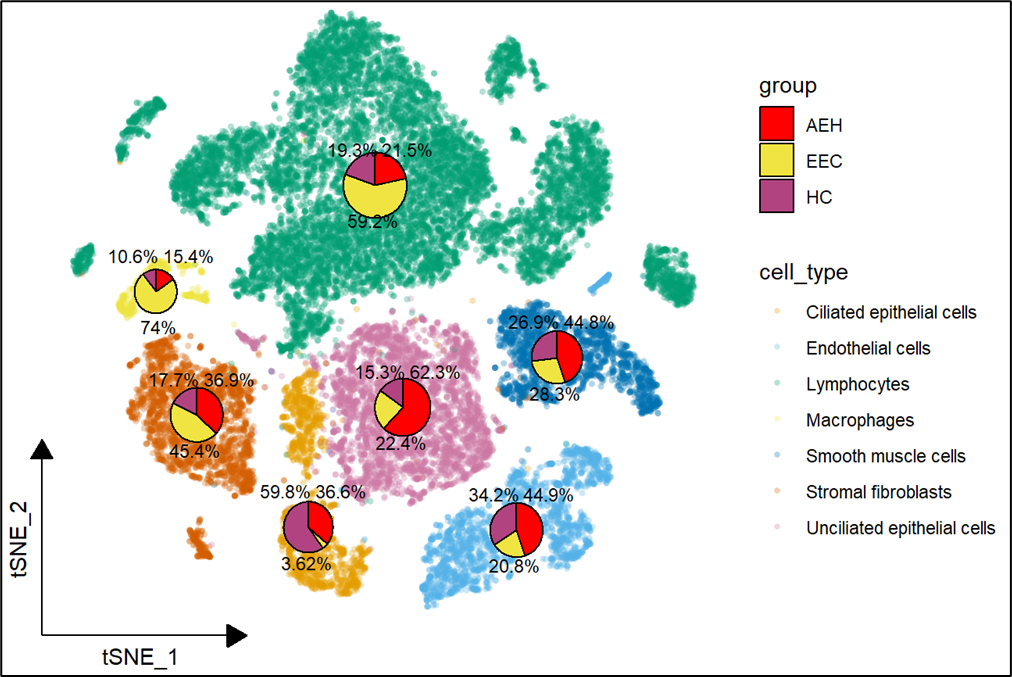
image.png
在这样就完成了,感兴趣的小伙伴可以在自己文章里面展示起来了。觉得分享有用的点个赞再走呗,也可以分享给其他需要的人!





















 824
824











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








