论文信息
题目:SegFormer3D: an Efficient Transformer for 3D Medical Image Segmentation
SegFormer3D:一种高效的3D医学图像分割Transformer
作者:Shehan Perera, Pouyan Navard, Alper Yilmaz
源码:https://github.com/OSUPCVLab/SegFormer3D.git
论文创新点
- 轻量级架构设计:作者提出了SegFormer3D,这是一个轻量级的Transformer架构,它在保持性能的同时显著减少了模型的参数数量和计算复杂性。SegFormer3D拥有33倍更少的参数和13倍减少的GFLOPS,相比于当前的最先进(SOTA)模型。
- 多尺度体积特征注意力计算:SegFormer3D通过一个层次化的Transformer来计算多尺度体积特征上的注意力,这使得模型能够捕获从粗到细粒度的输入特征,增强了对全局上下文的理解。
- 全MLP解码器:与传统的复杂解码器不同,SegFormer3D采用了一个全MLP解码器来聚合局部和全局注意力特征,以产生高度准确的分割掩码,这种方法简化了解码过程并提高了效率。
摘要
视觉Transformer(ViTs)架构的采用代表了3D医学图像(MI)分割领域的重要进步,通过增强全局上下文理解超越了传统的卷积神经网络(CNN)模型。尽管这种范式转变显著提高了3D分割性能,但最先进的架构需要极其庞大和复杂的架构以及大规模计算资源进行训练和部署。此外,在医学成像中经常遇到的有限数据集的背景下,更大的模型可能在模型泛化和收敛方面带来障碍。为了应对这些挑战,并证明轻量级模型在3D医学成像中是一个有价值的研究领域,我们提出了SegFormer3D,这是一个层次化的Transformer,它在多尺度体积特征上计算注意力。此外,SegFormer3D避免了复杂的解码器,并使用全MLP解码器聚合局部和全局注意力特征以产生高度准确的分割掩码。所提出的内存高效的Transformer在紧凑设计中保留了显著更大模型的性能特征。SegFormer3D通过提供具有33倍更少参数和比当前最先进(SOTA)模型减少13倍GFLOPS的模型,使深度学习在3D医学图像分割中得以普及。我们在三个广泛使用的Synapse、BRaTs和ACDC数据集上对SegFormer3D进行了基准测试,取得了有竞争力的结果。
1. 引言
深度学习在医疗保健中的兴起是变革性的,提供了前所未有的学习和分析复杂医学数据模式的能力。医学图像分析中的一个基本任务是3D体积图像分割,这对于诊断和治疗中的肿瘤和多器官定位至关重要。传统的方法涉及使用编码器-解码器架构,其中图像首先被转换为低维表示,然后解码器将表示映射到体素级分割掩码。然而,这些架构由于其有限的感受野而难以生成准确的分割掩码。最近,基于Transformer的技术由于ViT利用注意力层捕获全局关系的能力而展示了优越的分割性能。这与CNN表现出的局部归纳偏置特性形成了鲜明对比。继TransUnet[5]和UNETR[11]的开创性工作之后,医学界的大量研究致力于设计利用ViT的强大编码能力和CNN在解码阶段的特征细化能力的基于Transformer的架构。例如,[10, 11, 31]结合了卷积的局部感受野和全局注意力。尽管它们有优势,但ViT在从头开始训练时未能匹配CNN的泛化能力,并且由于缺乏归纳偏置,通常依赖于大规模数据集进行预训练[7],这在医学图像领域并不常见。此外,ViT的计算效率受到多头自注意力块中的浮点运算和逐元素函数数量的限制[17]。在3D医学成像任务中,这个问题更加突出,因为3D体积输入转换后的序列长度相当长。此外,医学成像数据经常表现出重复结构[6],这表明它可以被压缩,这是3D SOTA ViT架构在医学领域经常忽视的考虑因素。
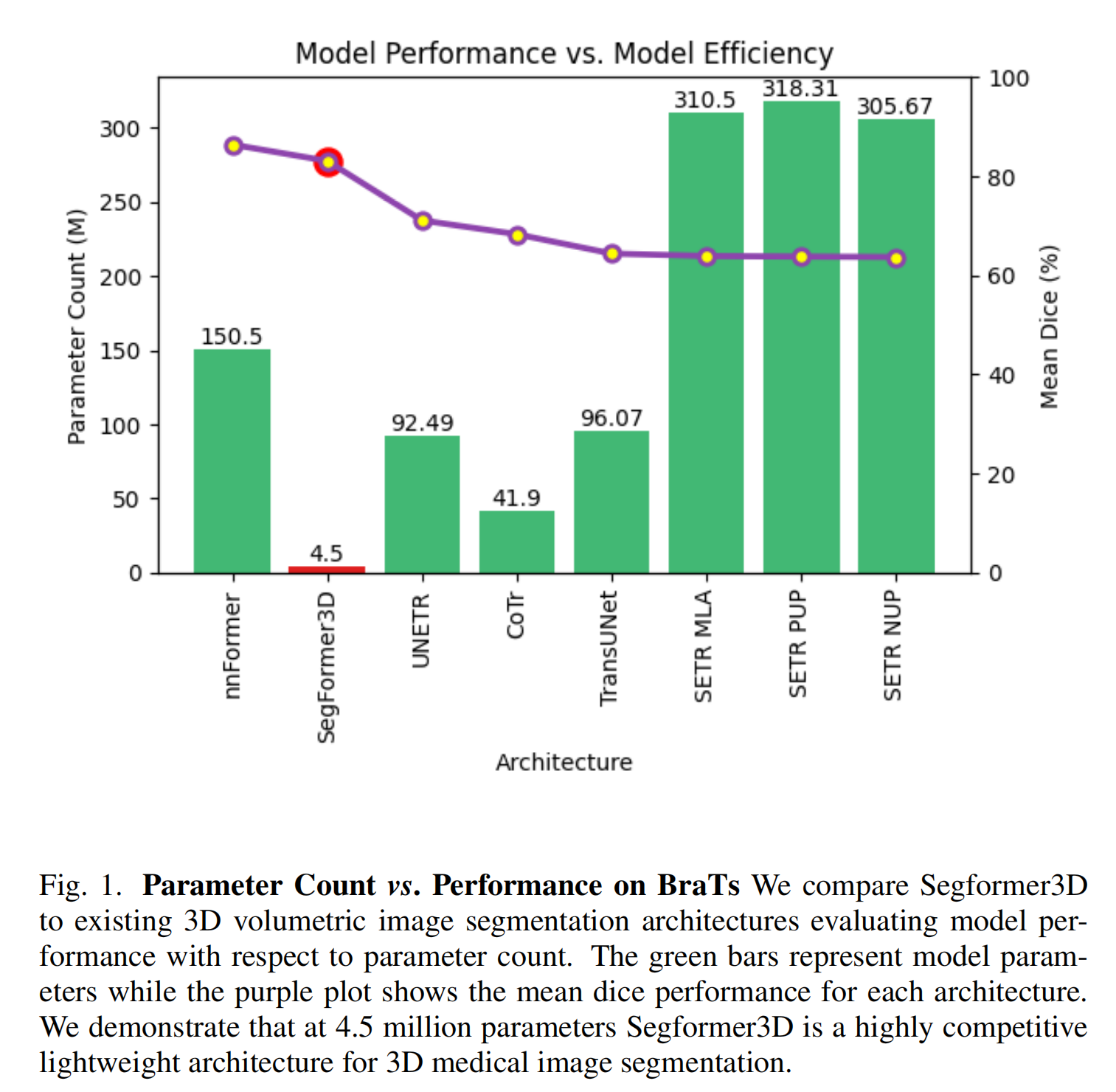
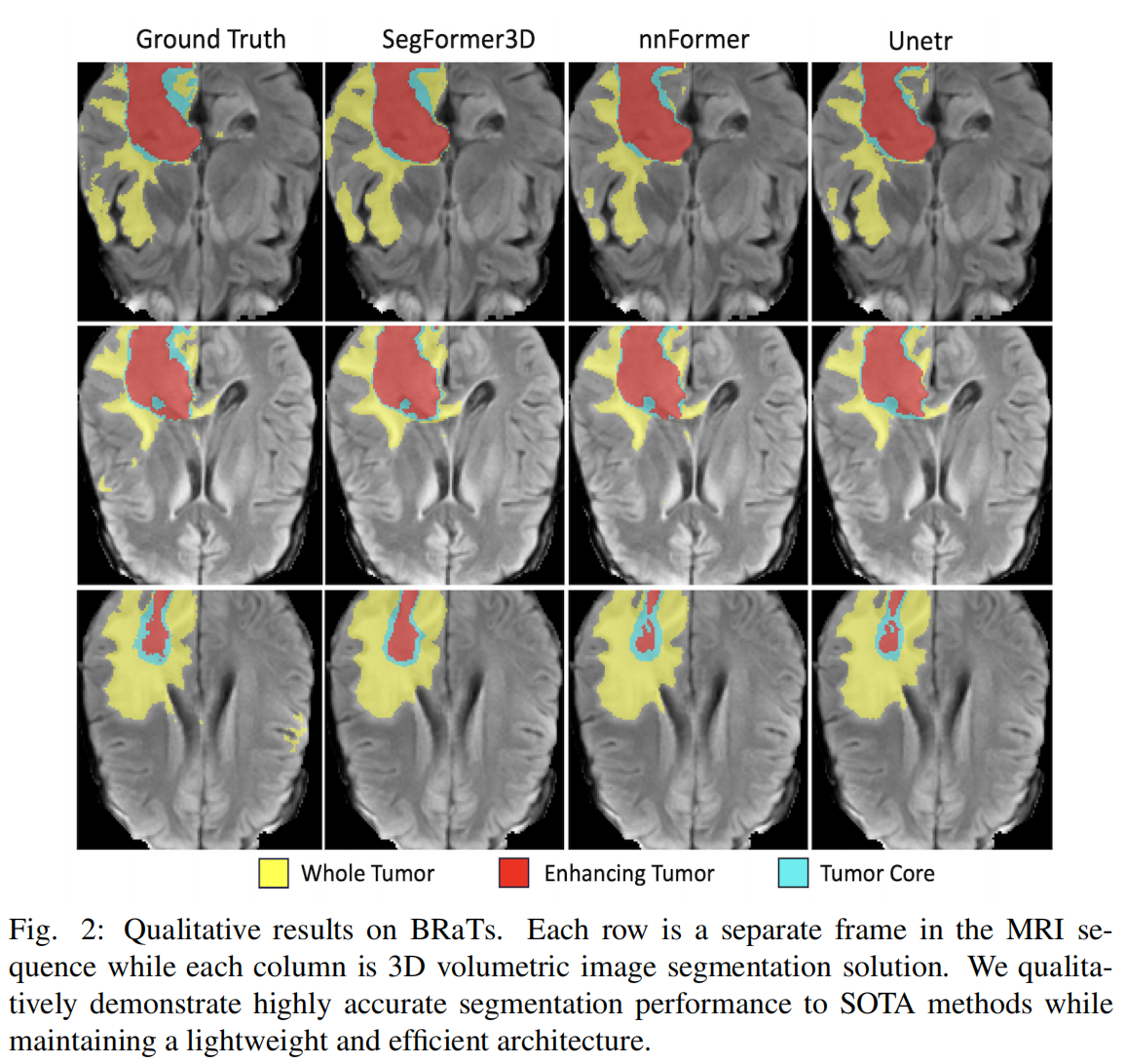
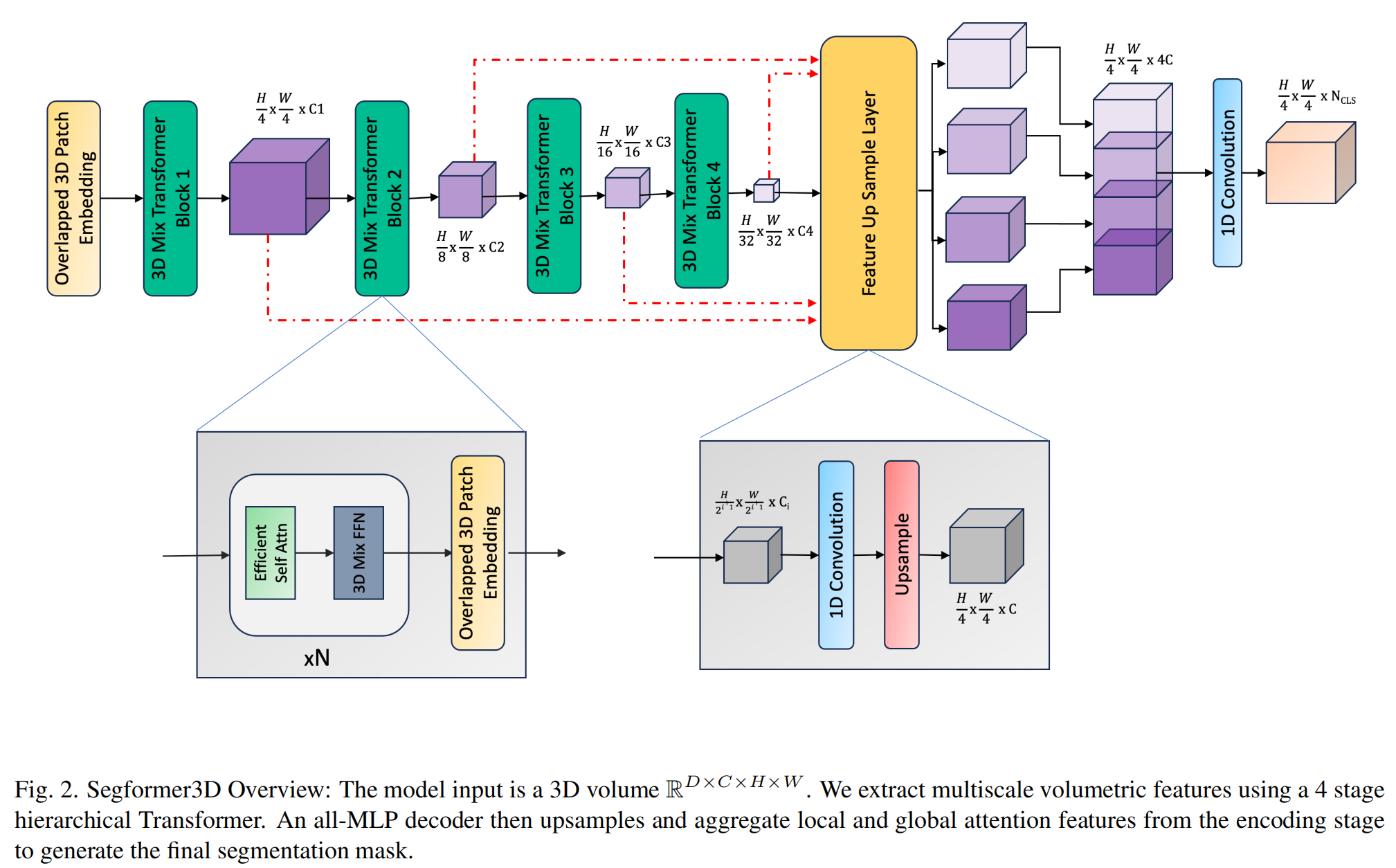
本文介绍了SegFormer3D,这是一个体积层次ViT,将[26]扩展到3D医学图像分割任务。与在固定尺度上渲染特征图的普通ViT[7]不同,SegFormer3D在输入体积的不同尺度上编码特征图,遵循Pyramid Vision Transformer[25]。设计使Transformer能够捕获输入的粗到细粒度特征。SegFormer3D还使用在[26]中使用的高效自注意力模块,将嵌入序列压缩到固定比例,显著降低模型复杂性而不失性能。此外,SegFormer3D利用在[26]中使用的重叠补丁嵌入模块,保留了输入体素的局部连续性。这种嵌入使用无位置编码[14],防止了在训练和推理过程中常见的分辨率不匹配时的准确性损失,这在医学图像分割中很常见。为了高效生成高质量的分割掩码,SegFormer3D使用了在[26]中引入的全MLP解码器。在三个基准数据集Synapse[15]、ACDC[1]和BRaTs[20]上的综合实验验证了SegFormer3D的定性和定量有效性。作者的贡献可以总结为:
- 我们引入了一个轻量级内存高效的分割模型,保留了大型模型在3D医学成像中的性能特征。
- 凭借450万参数和17 GFLOPS,SegFormer3D在参数数量和模型复杂性方面比SOTA减少了34倍和13倍。
- 我们展示了在没有预训练的情况下具有高度竞争力的结果,强调了轻量级ViT的泛化能力,探索像SegFormer3D这样的架构是医学成像中一个有价值的研究领域。
3. 方法
Transformer的采用极大地提高了体积医学图像分割的性能。然而,当前高性能架构为了模型性能而优先过度参数化,牺牲了效率。为了证明轻量级和高效的Transformer的好处,而不影响性能,我们引入了Segformer3D。凭借450万参数和17 GFLOPS,我们展示了参数数量和复杂性的34倍和13倍减少,展示了所提出架构在3D医学图像分割中的重要性。
编码器:
在Transformer框架内使用3D医学图像会导致长序列长度,增加了模型的计算复杂性。例如,一个标准的3D MRI体积,尺寸为128³,会产生一个序列长度为32,768,而一个典型的2D RGB图像,尺寸为256²,会产生一个序列长度为256。我们的层次化Transformer包含三个关键元素,以提高计算效率并减少总参数数量,同时保持SOTA级别的性能。首先,我们引入了重叠补丁合并,以克服在体素生成过程中的邻域信息丢失。这种技术与ViT[7]中看到的补丁机制不同,允许模型更好地理解体素之间的过渡点,并已被证明可以提高整体分割精度[26]。接下来,为了解决序列长度瓶颈而不影响性能,我们集成了一个高效的自注意力机制[25]。这种方法使模型能够更有效地捕获长距离依赖性,促进了改进的可扩展性和性能。传统的自注意力以形状为[Batch, Sequence, Features]的向量序列作为输入,并生成3个独特的投影,Query、Key和Value向量。生成后,注意力分数计算为(Q, K, V) = Softmax( Q K T d h e a d \frac{QK^T}{\sqrt{d_{head}}} d








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章


















 559
559

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










