小罗碎碎念
今日顶刊:Nature
今天精读的这篇文章于24-09-04发表于Nature,作者来自哈佛大学、斯坦福大学。

| 作者角色 | 作者姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Xiyue Wang | Department of Biomedical Informatics, Harvard Medical School | 哈佛医学院生物医学信息学系 |
| 第一作者 | Junhan Zhao | Department of Radiation Oncology, Stanford University School of Medicine | 斯坦福大学医学院放射肿瘤学系 |
| 通讯作者 | Sen Yang | College of Biomedical Engineering, Sichuan University | 四川大学生物医学工程学院 |
| 通讯作者 | Kun-Hsing Yu | Department of Pathology, Brigham and Women’s Hospital | 布莱根妇女医院病理科 |
通讯作者:Kun-Hsing Yu

Kun-Hsing Yu是一位在生物医学信息学和计算机科学领域拥有博士学位的学者,同时在台湾国立大学获得了医学博士学位。
他的研究重点是将定量的组织病理学图像模式与多组学(基因组学、表观基因组学、转录组学和蛋白质组学)资料整合,以推进癌症研究和临床实践。
他的团队开发了全自动算法来大规模分析全幻灯片的组织病理学图像,发现了肿瘤细胞显微表型的分子机制,并识别了对患者预后有影响的新型细胞形态。他的研究兴趣包括定量病理学、机器学习和转化生物信息学。
DBMI(生物医学信息学系)的研究领域包括:
- 人工智能
- 临床决策制定
- 计算组学
DBMI提供的课程有:
- BMI 707 - 生物医学数据的深度学习
目前,该系还提供了机器学习赋能病理学的博士后研究机会。
作者其他文章推荐
| 文章 | doi | 期刊 | IF |
|---|---|---|---|
| Prognostic role of inflammatory diets in colorectal cancer overall and in strata of tumor‐infiltrating lymphocyte levels | 10.1002/ctm2.1114 | Clinical and Translational Medicine | 7.9 |
| Prediction of early-stage melanoma recurrence using clinical and histopathologic features | 10.1038/s41698-022-00321-4 | npj Precision Oncology | 6.8 |
文献速览
这篇文章介绍了一种用于癌症诊断和预后预测的病理学基础模型——Clinical Histopathology Imaging Evaluation Foundation (CHIEF)。

-
研究背景:
- 问题:病理学图像评估在癌症诊断和亚型分类中至关重要。然而,现有的标准人工智能方法通常针对每个诊断任务进行优化,缺乏对不同数字化协议或样本来源的泛化能力。
- 难点:这些方法在面对来自不同人群和组织实验室的样本时,往往表现不佳,难以提供可靠的病理评估。此外,数据量和标注成本也是限制其广泛应用的重要因素。
- 相关工作:现有工作主要集中在为每个特定诊断任务训练专门的深度学习模型,但这些模型通常缺乏跨样本来源和组织实验室的泛化能力。自监督学习虽然显示出强大的特征表示能力,但在处理不同癌症类型和样本来源时仍存在局限性。
-
研究方法:
- CHIEF模型概述:CHIEF是一个通用的弱监督机器学习框架,通过两种互补的预训练方法提取多样的病理学表示:无监督预训练用于瓦片级特征识别,弱监督预训练用于全切片模式识别。
- 数据集和预训练:使用来自19个解剖部位的60,530张全切片图像进行预训练,数据量达44TB。预训练过程包括将全切片图像分割成非重叠的瓦片,并使用对比语言-图像预训练(CLIP)嵌入方法编码每个瓦片的解剖部位信息。
- 模型架构:CHIEF模型整合了显微成像和解剖部位信息,增强了特征表示。图像编码器使用自监督CTransPath骨干网络提取病理学图像特征表示,文本编码器采用CLIP模型的预训练文本编码器。通过注意力机制融合特征,并设计了特征聚合网络以整合瓦片间的上下文信息。
-
实验设计:
- 癌细胞检测:使用15个独立数据集进行外部验证,涵盖11种癌症类型,CHIEF在多种癌症类型的识别任务中表现优异,AUROC值在0.9098到0.9943之间。
- 肿瘤起源识别:在独立的CPTAC数据集上进行验证,CHIEF的预测准确率达到0.9853,显著高于其他方法。
- 基因组谱预测:系统预测癌症类型的常见基因突变,CHIEF在多个基因的突变状态预测中表现出较高的AUROC值,例如TP53在低级别胶质瘤中的AUROC值为0.8756。
- 生存预测:在17个数据集上进行生存预测,CHIEF区分不同生存结果的 concordance index (c-index)平均为0.74,比现有方法提高了12%。
-
结果与分析:
- CHIEF的泛化能力:CHIEF在处理来自不同中心、扫描仪和临床程序的多样病理样本时表现出高度的适应性。与现有的最先进深度学习方法相比,CHIEF在多种任务上表现出色,平均AUROC值最高可达0.9671。
- 基因组变异预测:CHIEF在预测多个致癌基因和抑癌基因的突变状态方面表现优异,AUROC值均大于0.8。
- 生存分析:CHIEF建立的生存预测模型能够准确区分不同生存风险的癌症患者,c-index在所有癌症类型中均值为0.74,显著优于现有方法。
-
总体结论:
- CHIEF是一个适用于多种病理评估任务的通用深度学习框架,展示了其在不同临床应用中的泛化能力。
- 通过最小化的图像注释和详细的定量特征提取,CHIEF为系统分析形态模式、分子异常和重要的临床结果提供了基础。
- 准确的、稳健的和快速的病理样本评估将有助于个性化癌症管理的发展。
一、绪论
组织病理学图像评估对于癌症及其亚型的诊断至关重要。
先前的人工智能(AI)在组织病理学图像分析研究主要依赖于针对每个使用案例优化特定的训练模型[1,2]。
例如,已开发专门的深度神经网络用于癌细胞识别[4,5]、组织学和分子亚型分类[6–10]、预后评估[11–14]以及利用吉帕像素全切片图像(WSIs)进行治疗反应预测[15–17]。
此外,最先进的计算病理学分析揭示了与临床重要分子标志物相关的定量形态信号[18,19],展示了AI方法在识别人眼无法察觉的细胞特征方面的潜力[20]。
尽管这些进展为提高癌症评估提供了有希望的道路,但几个局限性仍然困扰着定量病理图像分析。
首先,标准的深度学习方法需要大量数据来训练每个任务的性能模型。由于难以获得涵盖多样组织微环境异质性的全面病理表现,现有方法主要侧重于单独解决每个狭窄的诊断任务[1,7]。
其次,大多数用于病理成像分析的AI模型是从用于分类宏观对象(例如,动物、汽车和公交车)的通用计算机视觉模型定制而来[2]。这些传统方法在训练特定诊断模型时,没有利用普遍的组织病理学模式。
再者,仅通过单一来源图像训练的AI模型往往过度拟合训练数据分布,并在应用于不同病理实验室处理的图像时,性能大幅下降[3,21]。这些局限性阻碍了先进AI模型在可靠病理评估中的有效应用。
自监督学习作为一种有前景的方法,已出现用于在多样化设置中收集的样本上进行广泛预测任务,获得稳健的图像特征表示[22,23]。
由于多样化的未标记训练数据相对容易收集,且模型训练过程与任务无关,自监督学习在不同任务和数据分布上实现了稳健的性能,如图像检索[24–26]和弱监督WSI分析[27]。
近期,自监督学习在病理图像分析方面的进展进一步利用了图像及其文本描述来增强计算机视觉模型的性能[28,29]。然而,这些方法存在两个主要局限性。
首先,它们主要关注WSIs中的单个图像块,没有考虑同一组织的不同区域之间的相互作用。
其次,先前的研究专注于狭窄的诊断任务,并未评估在不同癌症类型和多个来源样本的预测任务中提取的定量成像特征的一般适用性。
由于病理学家经常面对各种疾病样本,并需要从组织微环境中融合上下文信息,开发一个能够适应广泛组织类型和评估任务的通用病理AI系统至关重要。
为解决这些紧迫的临床需求,作者建立了CHIEF模型,这是一个通用机器学习框架,为各种病理诊断和预测任务提供了基础(图1a)。

作者利用了两种互补的AI模型预训练方式:使用1500万病理图像块进行自监督预训练,以获得块级特征表示;在19个解剖部位的60,530个WSIs上进行弱监督预训练,以获得组织上下文表示。
此外,作者还设计了一个高效框架,用于大规模WSI分析中的块级特征聚合。作者进一步在包含19,491个弱注释WSIs的32个独立数据集上验证了CHIEF在癌症检测、肿瘤起源特征描述、基因组突变识别和生存预测方面的能力。
作者的方法挑战了传统的基于注意力的块聚合方法,提供了WSI特征的全面表示。CHIEF使得系统性的显微特征识别成为可能,并为可靠的病理评估奠定了基础。
二、CHIEF模型概览
作者建立了CHIEF模型,这是一个用于弱监督组织病理学图像分析的通用机器学习框架。
与常用的自监督特征提取器[27,30]不同,CHIEF采用了两种预训练程序:在1500万个未标记的图像块上进行无监督预训练,以及在超过60,000个WSIs上进行弱监督预训练。
块级别的无监督预训练建立了一个针对苏木精-伊红染色的组织病理学图像的通用特征提取器[30],这些图像来自异质性的公开可用数据库,捕获了显微细胞形态的多样化表现。随后的WSI级别的弱监督预训练通过刻画癌症类型之间的相似性和差异性,构建了一个通用目的模型。
作者在广泛的病理评估任务中检验了CHIEF的性能,包括癌症检测、肿瘤起源预测、基因组谱识别和生存预测(图1a)。
模型设计与实施的具体细节小罗不做详细描述。
2-1:CHIEF增强的癌细胞检测
从病理图像中检测恶性细胞对于癌症诊断至关重要[4,5]。
目前最先进的人工智能(AI)方法在癌细胞检测方面主要集中训练针对特定癌症类型的模型,而没有利用跨癌症恶性细胞形态的共同性。因此,这些模型不易扩展到其他癌症类别。
为解决这一不足,作者使用CHIEF构建了一个弱监督的癌症检测平台,并评估了其在不同癌症中的泛化能力。作者使用包含13,661个WSIs的15个独立数据集进行了广泛的外部验证。这些数据集涵盖了公共数据源(例如,临床蛋白质组肿瘤分析联盟(CPTAC)、Diagset-B31、Dataset-PT32、诊断参考肿瘤成像数据库(DROID)-乳腺和TissueNet33队列)和机构数据源(例如,深圳妇幼保健院(SMCH)和重庆大学癌症医院(CUCH)的样本),包含活检和手术切除切片,并覆盖了11种不同的原发癌症部位,包括乳腺、子宫内膜、食管、胃、宫颈、结肠、前列腺、肾脏、皮肤、胰腺和肺。
为了更好地评估CHIEF的性能,作者将之与三种弱监督WSI分类方法进行了比较:
- 聚类约束注意力多实例学习(CLAM)[6]
- 基于注意力的深度多实例学习(ABMIL)[34]
- 双流多实例学习网络(DSMIL)[35]。
CHIEF在多种癌症识别任务中一致取得了优越的性能,无论是使用活检切片还是手术切除切片(图2a)。

CHIEF在代表11种癌症类型的15个数据集上实现了0.9397的宏观平均接收者操作特征曲线下面积(AUROC)(图2a),这比DSMIL(宏观平均AUROC为0.8409)高出约10%,比ABMIL(宏观平均AUROC为0.8233)高出12%,比CLAM(宏观平均AUROC为0.8016)高出14%。
在从独立队列收集的所有五个活检数据集中,CHIEF在包括食管(CUCH-Eso)、胃(CUCH-Sto)、结肠(CUCH-Colon)和前列腺(Diagset-B和CUCH-Pros)在内的几种癌症类型中取得了超过0.96的AUROC。
在跨越五种癌症类型(即结肠(Dataset-PT)、乳腺(DROID-Breast)、子宫内膜(SMCH-Endo和CPTAC-子宫内膜癌(UCEC))、肺(CPTAC-肺鳞状细胞癌(LUSC))和宫颈(SMCH-Cervix和TissueNet)的七个手术切除切片数据集上进行独立验证时,CHIEF取得了超过0.90的AUROC。
尽管CHIEF和基准方法在CPTAC上的性能较低,但CHIEF在这些数据集中的癌细胞识别显著优于所有其他方法(DeLong检验P值<0.001)。这些结果证明了CHIEF在国际上不同癌症组织和样本来源中的泛化能力。
作者使用全切片注意力可视化来识别CHIEF模型利用的诊断信号。
图2b、扩展数据图2和补充图1展示了原始WSIs、病理学家标注的像素级真实情况(方法)和CHIEF输出的注意力图。
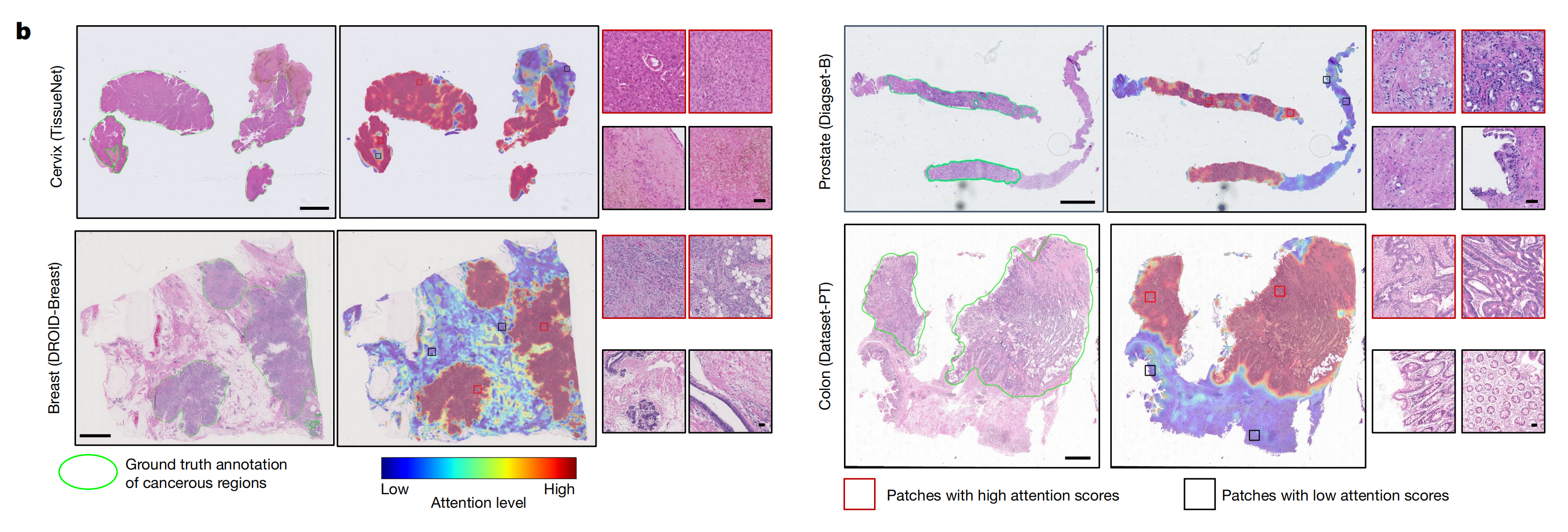
CHIEF将其大部分注意力集中在癌变区域,尽管仅使用切片级标签进行训练,但在像素级与真实情况标注表现出显著的一致性。
值得注意的是,CHIEF给予高度注意的图像块包含了具有恶性典型细胞学和结构模式的组织(例如,增加的核/细胞质比、不规则形状的细胞核、细胞多形性和组织结构紊乱),显示了模型使用弱监督方法识别关键诊断特征的能力。
2-2:CHIEF识别肿瘤来源
作者成功地利用CHIEF预测了癌症的组织来源,并使用来自CPTAC的独立测试集验证了结果。
扩展数据图1和补充表5-7展示了详细的成果。
2-3:CHIEF预测基因组谱
癌症样本的基因组谱预示了患者的治疗反应,对于制定治疗方案至关重要[19]。
由于额外的成本和时间投入,全球范围内并未常规进行癌症患者的全面基因组分析[18]。
从常规的苏木精-伊红染色切片中识别指示基因组谱的定量形态模式,提供了一个即时且成本效益高的基因组测序替代方案。
作者检验了CHIEF在系统预测癌症样本分子谱方面的能力,专注于四个临床重要的预测任务:
- 跨癌症类型系统性预测常见遗传突变;
- 识别与靶向治疗相关的突变;
- 针对WHO分类的胶质瘤预测异柠檬酸脱氢酶(IDH)状态;
- 预测结直肠癌(CRC)患者免疫检查点阻断治疗的益处,即微卫星不稳定(MSI)预测。
2-4:常见遗传突变分析
作者进行了一项系统性分析,将常见遗传突变与组织病理学图像相关联(图3和扩展数据图3)。
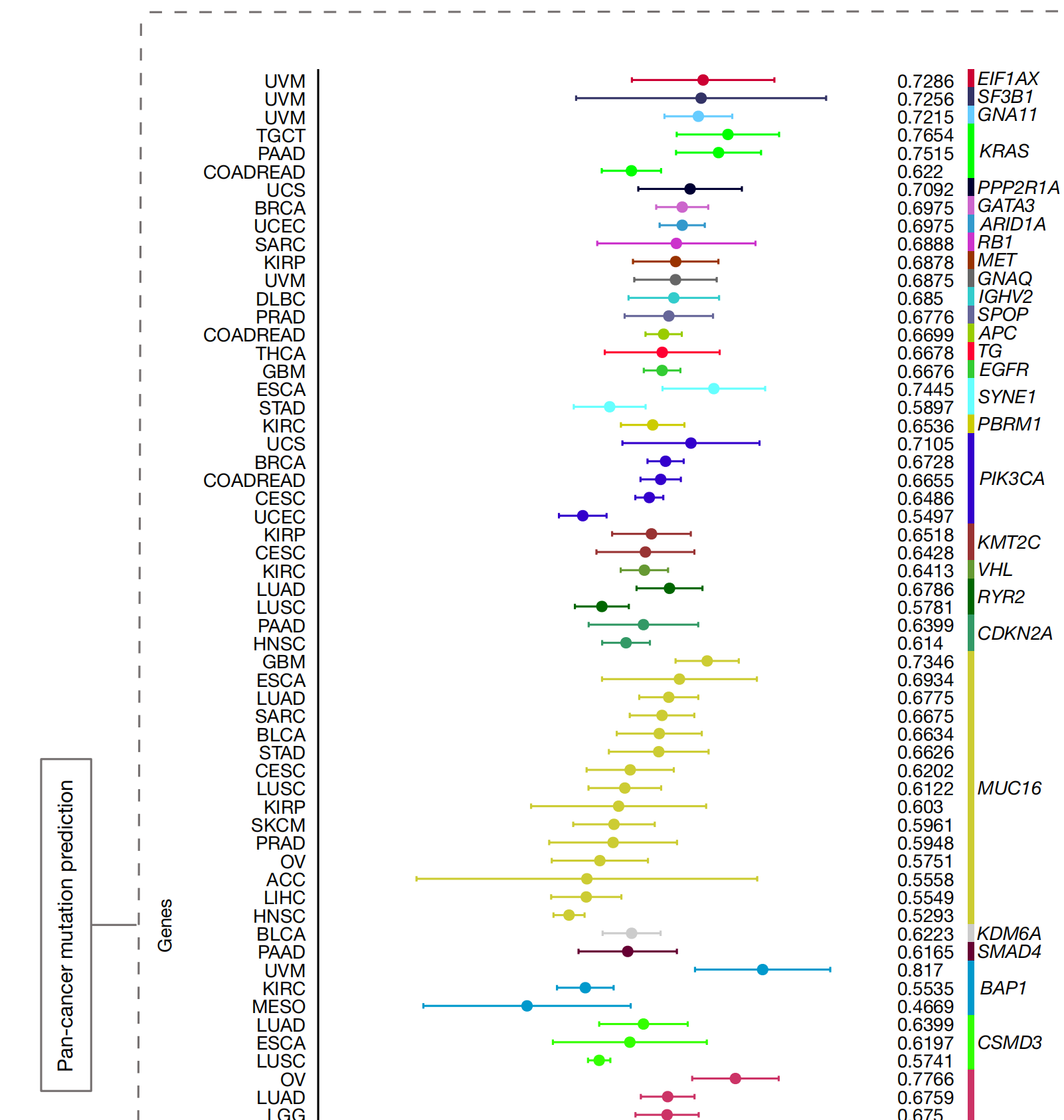
研究涉及30种癌症类型中的13,432个WSIs,以及每种癌症类型中突变率最高的前五个基因,共53个基因。CHIEF在作者的系统性泛癌症遗传突变分析中预测了九个基因的突变状态,AUROCs超过0.8(图3)。
与先前的研究[18,36]一致,病理图像在19种癌症类型中包含与TP53突变强烈相关的信号,其中低级别胶质瘤(LGG;AUROC为0.8756,95%置信区间(CI)为0.8624–0.8888)、肾上腺癌(AUROC为0.8119,95% CI为0.7488–0.8751)和子宫内膜癌(UCEC;AUROC为0.8115,95% CI为0.7971–0.8259)的AUROCs较高。
CHIEF还识别了GTF2I基因的突变,该突变在43.4%的胸腺上皮肿瘤患者中发生[37],AUROC为0.9111(95% CI为0.8935–0.9287)。
此外,CHIEF预测了葡萄膜黑色素瘤中BAP1基因的突变(AUROC为0.817,95% CI为0.7668–0.8672),该突变在大约45%的葡萄膜黑色素瘤病例中出现[38]。
作者在来自CPTAC的独立患者队列中测试了CHIEF。
CHIEF在这些新患者队列中为各种基因持续保持了相似的AUROCs(扩展数据图4)。与基于组织病理学的基因组突变预测的最先进方法(即泛癌症计算组织病理学(PC-CHiP)方法[36];补充图2)相比,CHIEF显示出显著更高的性能(Wilcoxon符号秩和检验P值<0.001),宏观平均AUROC为0.7043(范围0.51–0.89)。
相比之下,PC-CHiP方法的宏观平均AUROC为0.6523(范围0.39–0.92)。
2-5:靶向治疗相关突变预测
作者进一步使用CHIEF来预测与FDA(食品和药物管理局)批准的靶向治疗相关的基因,这些治疗信息收录于OncoKB39(www.oncokb.org),涵盖了15种癌症类型中的18个基因(图3)。
CHIEF预测了所有18个基因的突变状态,AUROCs均超过0.6(图3)。预测性能较高的突变包括弥漫性大B细胞淋巴瘤中的EZH2(AUROC=0.9571,95% CI 0.9321–0.9822),胃腺癌中的NTRK1(AUROC=0.8192,95% CI 0.7767–0.8618),前列腺腺癌中的BRCA2(AUROC=0.8938,95% CI 0.8310–0.9567),甲状腺癌中的BRAF(AUROC=0.8889,95% CI 0.8715–0.9064),肺鳞状细胞癌(LUSC)中的ERBB2(AUROC=0.8211,95% CI 0.7597–0.8826)以及膀胱尿路上皮癌中的FGFR3(AUROC=0.8161,95% CI 0.7921–0.8402)。
在独立验证中,CHIEF在CPTAC队列中实现了相似水平的性能(扩展数据图4)。在这些基因中,乳腺癌(BRCA)中的ESR1,肺腺癌(LUAD)中的EGFR,以及结肠腺癌和直肠腺癌(COADREAD)中的BRAF在预留和独立测试集上的AUROCs均超过0.7。
2-6:IDH状态预测
世界卫生组织中枢神经系统肿瘤的第五版分类将胶质母细胞瘤(GBM)与低级别胶质瘤(LGG)区分开,基于IDH状态而非传统的组织学特征[8,40]。
因此,在诊断时识别患者的IDH状态至关重要。为了独立于组织学分级识别与IDH突变相关的信号,作者将研究队列按组织学分级分层,并使用CHIEF在每个分层中预测IDH状态。
作者在六个数据集上进行了IDH状态预测分析:癌症基因组图谱(TCGA)-LGG、TCGA-GBM、维也纳医科大学(MUV)-LGG和MUV-GBM、哈佛医学院和宾夕法尼亚大学(HMS)-LGG和HMS-GBM,共计2,718个WSIs。
CHIEF模型在预留和独立测试集上均表现出优于其他基准方法的性能(Wilcoxon符号秩和检验P值<0.01;图4a和补充图3)。
为了增加可解释性,作者可视化了定量图像特征向量,并检查了由CHIEF确定的注意力分数的分布(扩展数据图5和9b)。
结果显示,在识别IDH野生型胶质瘤时,坏死区域显著吸引了更多的注意力(Mann–Whitney U检验P<0.0001;扩展数据图9b)。
2-7:MSI状态预测
MSI是结直肠癌(CRC)对免疫检查点阻断治疗反应的一个公认的生物标志物[27]。
为了在诊断时实现快速的治疗个性化,作者检查了CHIEF在预测MSI状态方面的性能。CHIEF在TCGA-COADREAD数据集和两个独立队列(PAIP202042和CPTAC-COAD)中显著优于表现最佳的基准方法(DSMIL),AUROC提高了约12%,15%和26%,分别(图4b)。
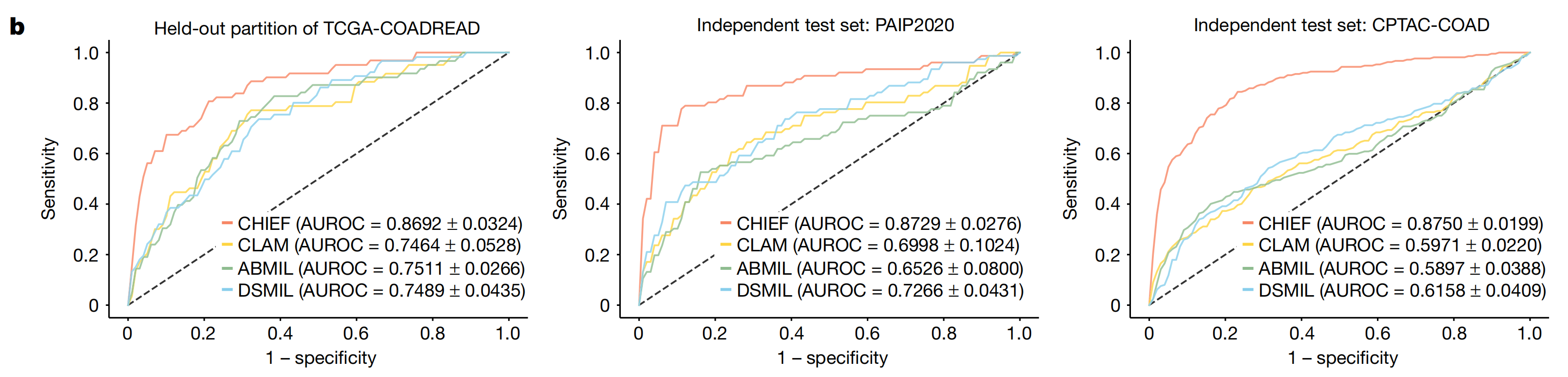
注意力分析显示,CHIEF对包含实体肿瘤、腔内坏死和肿瘤浸润淋巴细胞的区域给予了高度的关注(扩展数据图6)。
三、CHIEF预测生存结果
由于对标准治疗的反应不同,癌症患者的疾病特异性生存结果在初始诊断后存在差异[43]。
尽管提出了许多临床和基因组生物标志物,但它们并不能完全预测每个患者的预后。
为了解决这一挑战,作者将CHIEF框架扩展,建立了针对每种癌症类型的阶段分层生存预测模型。作者使用了总共9,404个WSIs,来自17个数据集(包括公共可获得和机构样本来源),并专注于7种具有可靠预后信息的癌症类型(COADREAD、LUSC、BRCA、GBM、UCEC、LUAD和肾细胞癌(RCC))。
CHIEF成功使用初次诊断时获得的组织病理学图像预测了患者的生存结果。在所有癌症类型和所有研究队列中,CHIEF能够区分长期生存患者和短期生存患者(对数秩检验P<0.05;图5展示了I期和II期癌症患者的预测结果)。
相比之下,最先进的深度学习方法(例如,病理组学研究平台整合生存估计(PORPOISE)[12]和DSMIL[35])在相同条件下无法可靠地区分不同生存结果的患者(对数秩检验P>0.05的队列有11个;补充图4)。
此外,CHIEF产生的Kaplan-Meier曲线具有较窄的置信区间。总的来说,CHIEF在预留测试集中的平均一致性指数(c-index)为0.74,在所有癌症类型中,比PORPOISE和DSMIL分别高出12%和7%。值得注意的是,CHIEF与基准方法之间的性能差异在未参与模型开发过程的独立队列中更为显著。
在这些患者人群中,CHIEF的平均c-index为0.67(比所有基准模型高出9%),并在所有数据集中区分了不同生存结果的患者,而PORPOISE和DSMIL的平均c-index分别为0.54和0.58。
作者观察到,在III期(补充图6)和IV期癌症(补充图7)患者中,CHIEF的性能趋势与I期和II期类似,其性能优于其他方法多达10%。
由于一些先前发表的方法关注混合阶段的结果,作者计算了混合阶段分析的结果,并显示CHIEF在这些研究设置中优于基准方法。
此外,作者进行了多变量分析,将模型生成的风险评分、患者年龄、性别和阶段(补充表9和10)纳入考虑。结果显示,CHIEF生成的风险评分是一个独立于已知生存结果指标的显著预后因素。
作者的单变量分析还显示,CHIEF生成的风险评分与所有癌症类型中所有患者队列的生存结果在统计学上显著相关(补充表11和12)。相比之下,其他基于病理成像的方法在大多数患者队列中,无论是多变量还是单变量分析,都不能区分患者的生存结果。
为了更好地理解与患者生存结果相关的组织学特征,四位主治病理学家独立审查了CHIEF生成的注意力热图(方法)。
在长期生存者和短期生存者中,高注意力区域都包含各种癌症类型的恶性组织(扩展数据图8和9以及补充图8和9)。
长期生存者的高注意力区域比高风险死亡率患者区域含有更多的浸润性免疫细胞。在短期生存者的癌症样本中,高注意力区域显示出较大的核/细胞质比、更明显的核异型、较少的间质纤维化和较弱的细胞间粘附。
四、讨论
作者开发了CHIEF作为一种通用的、泛癌症的深度学习框架,用于定量病理评估。
CHIEF利用了无监督的块级预训练、弱监督的全切片图像(WSI)级预训练,并使用了来自几个国家的44TB组织病理学成像数据,以实现稳健的病理图像分析。
CHIEF框架成功描述了肿瘤来源,预测了临床上重要的基因组谱,并分层的将患者分为长期生存和短期生存组。此外,作者的方法建立了一个能够进行广泛预测任务的一般病理特征提取器,即使样本量较小。
作者的结果显示,CHIEF对多样化的病理样本具有高度的适应性,这些样本来自不同的中心、使用各种扫描仪数字化,并来自不同的临床程序(即活检和手术切除)。
这个新框架显著提高了模型的泛化能力,这是传统计算病理学模型临床渗透的一个关键障碍[1,3]。
CHIEF有效地利用了解剖部位信息作为先验知识,并考虑了WSIs中不同图像区域之间的上下文交互,这使得其泛化能力远优于标准方法。
作者成功地在各种WSI级别的预测任务中使用了CHIEF框架,并且作者的模型在性能上优于最先进的方法。例如,CHIEF展示了识别未参与训练过程的患者队列中肿瘤原发病灶起源的稳健能力。
此外,CHIEF在预测基因组变异方面显著优于基准方法[36]。特别是,CHIEF预测了多种癌基因和肿瘤抑制基因的突变状态,表现出较高的性能(AUROCs>0.8),例如TP53、GTF2I、BTG2、CIC、CDH1、IGLL5和NRAS。
由于世界卫生组织诊断指南将分子标记纳入肿瘤分类,作者进一步展示了CHIEF预测与主要诊断类别相关的关键突变,并在多个患者人群中验证了结果。CHIEF还准确预测了CRC患者的MSI状态,这可能有助于临床决定是否给予免疫检查点抑制剂[18,19,27]。
最后,CHIEF提取的成像特征为生存结果预测模型奠定了基础。这些模型将所有癌症类型的患者分为高风险和低风险死亡组,并在17个队列中得到验证。
作者进一步通过可视化CHIEF模型给予高注意力的成像区域来解释CHIEF模型。
CHIEF采用了一种弱监督的机器学习方法,该方法通过比较正例和负例自动识别感兴趣的区域,从而消除了对像素级或区域级注释的需求。这种方法使得可以利用大规模的公共可获得和机构数据集来捕捉成千上万样本中病理表现的异质性。
例如,生存结果预测模型的可视化显示,来自癌症患者低死亡率风险样本中包含更多的浸润性免疫细胞和丰富的间质,具有清晰的腺体和筛状结构。
最后,作者展示了CHIEF在统计上显著优于最近发布的通用基础模型和基于补丁的病理基础模型[26,44–46](补充图10和补充表25和26)。额外的弱监督预训练方法利用大规模WSI数据集可能有助于提高其性能。
作者的研究有一些局限性。
首先,尽管CHIEF是在来自世界各地多家医院和研究队列的大量样本上进行训练的,但包括更多的非恶性切片和罕见疾病的切片可能会进一步提高作者通用病理特征提取器的性能。
此外,作者的预后预测模型专注于接受标准治疗的患者的疾病特异性和总体生存预测。未来的研究可以扩展作者的方法,研究新癌症治疗预测的好处和不良影响。
总的来说,CHIEF是一种适用于多种癌症类型的广泛病理评估任务的通用基础模型。作者已经展示了这个基础模型在全球24家医院和患者队列中收集的样本中的泛化能力。
CHIEF需要最少的图像注释,并从WSIs中提取详细的定量特征,这使得可以系统地分析形态学模式、分子异常和重要临床结果之间的关系。CHIEF提供的准确、稳健和快速的病理样本评估将有助于个性化癌症管理的发展。

























 801
801

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








