同一作者的两篇配准文章,考虑到是比较久之前的论文,将它们放到同一篇博客中
第一篇论文链接:
基于深度学习的分割法对腹部CT进行高效的两步多器官配准 - ScienceDirect

一、摘要
三维(3D)腹部计算机断层扫描(CT)的配准对于计算机辅助疾病的诊断和治疗至关重要,但腹部的非刚性呼吸运动(non-rigid respiratory movements )增加了其难度。该文提出一种用于腹部CT扫描的两步多器官自动配准方法。首先,将轻量级挤压激励(squeeze-and-excitation ,SE)注意力块和基于全连接条件随机场(conditional random field,CRF)的后处理集成到基于全卷积网络(FCN)的模型中,对肝、肾、脾等腹部多器官获得更准确的分割结果;然后,结合非刚性局部相关系数(LCC)相似度量和各向同性全变正则化来记录多器官区域,从而减少计算时间,避免变形场过平滑问题。
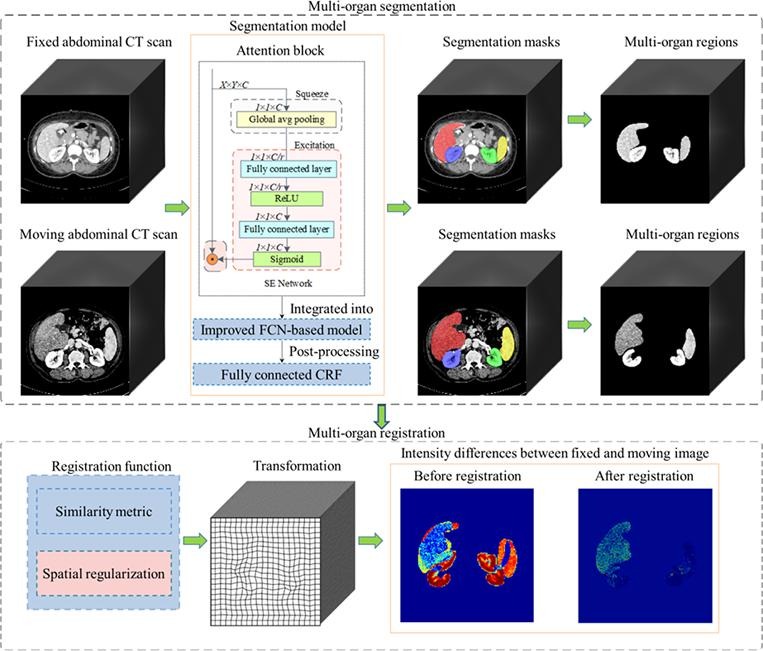
二、方法
考虑到图像内容的完整性,基于体素的方法优于基于表面的方法。然而,基于体素的方法的性能会受到目标器官周围噪声和强度的影响,导致计算负担沉重,配准结果不理想。因此,我们提出了一种基于分段的策略,以在基于体素和基于表面的方法之间进行折衷。它可以保留目标器官的关键图像内容,并以更少的计算负担提高配准性能。所提出的方法由两部分组成:腹部多器官分割和配准。首先,采用改进的基于FCN的模型从固定和移动腹部CT扫描中进行初始多器官分割,然后使用完全连接的CRF来微调分割掩码并提取多器官区域。然后,将基于强度的相似度量和空间正则化项集成到配准函数中,以找到最优变换场并注册成对的腹部多器官区域;
1.基于FCN的腹部CT多器官分割
本文提出了一种改进的基于FCN的三维腹部多器官分割模型。所提模型的主要模块包括卷积层、反卷积层、密集块和注意力块。首先,从V形网络模型中学习卷积层和解卷积层的整体结构。在框架的左侧,对输入图像进行卷积运算,得到72^个特征图,用于减少计算。然后,将密集块、注意力块和卷积层级联以生成三种分辨率的特征图(72^3, 36^3和 18^3),其中卷积层在每个分辨率的末端执行,以减少特征图的数量。在框架的右侧,对每种分辨率的特征图应用基于双线性插值的反卷积操作,然后通过卷积连接三种分辨率的特征图。最后,将生成的分割结果上采样至输入图像的大小。该模型中的 3D 卷积操作包括用于特征学习的卷积内核、用于减少内部协变量偏移然后防止梯度扩散的批量归一化 ,以及参数校正线性单元 (pReLU) 的激活函数 ,用于非线性表示。在卷积过程中进一步采用分批空间dropout策略解决该模型的过拟合问题。此外,密集块的卷积层通过前馈方式相互密集连接,其中不同层的特征图可以在仅存储一次的情况下重复使用。这种前馈方式可以提高密集块的内存效率,性能更好。

诸如全局上下文(global context,GC)网络和挤压和激励(squeeze-and-excitation,SE)网络之类的注意力块在深度神经网络中集成为高效,轻量级和多功能块,可以提高分割精度。GC 块具有有效建模全局上下文的能力,SE 块利用卷积特征通道之间的独立性来重新调整权重以突出显示重要特征和削弱不重要特征。为了从腹部CT图像中精确提取ROI,我们尝试将SE或GC块集成到所提出的基于FCN的框架中,分别称为SE_DVnet模型和GC_DVnet模型。(a)所示的SE模块由三个模块组成:(a)将整个空间特征编码为通道上的全局特征的挤压模块,(b)学习每个特征通道之间的关系并获得这些通道权重的激励模块,以及(c)融合模块将加权通道全局特征乘以原始空间特征。所示的GC块主要分为三个模块:(a)聚合所有位置特征形成全局上下文特征的上下文建模模块,(b)捕获特征通道间依赖关系的转换模块,以及(c)添加从上述两个模块获得的特征的融合模块。如图所示,X×Y表示空间维度,C通道数,以及r作为超参数的减速比率。

注:挤压-激励(Squeeze-and-Excitation,简称SE)网络是一种用于提高卷积神经网络性能的注意力机制。它通过对每个通道的特征进行重新加权来增强网络对输入图像中重要特征的响应。SE网络包括两个部分:挤压(Squeeze)和激励(Excitation)。挤压操作通过全局平均池化来获取每个通道的全局信息,然后激励操作通过全连接层来学习每个通道之间的关系并计算每个通道的权重。最后,这些权重被用来对原始特征图进行重新加权。在本文中,作者将SE网络作为注意力模块集成到改进的基于FCN的模型中,以提高模型的特征学习能力并获得更精确的目标器官轮廓细节。
基于FCN的模型中的反卷积可能会导致轮廓特征的丢失,从而导致目标分类边界的模糊。为了优化分类图像的粗糙度和不确定性,捕捉分割区域的精细细节,并获得更详细的分割边界,本研究提出了一种完全连接的CRF作为后处理策略。此模块的能量函数为:

CRF(条件随机场,Conditional Random Field)是一种用于序列标注和分割任务的概率图模型。它能够对输入序列中的每个元素进行分类,同时考虑相邻元素之间的依赖关系。CRF模型通常由两部分组成:(1)特征函数,用于描述输入序列中每个元素的特征;(2)权重参数,用于控制特征函数对最终预测结果的贡献。在训练过程中,CRF模型通过最大化训练数据的对数似然来学习权重参数。在预测过程中,CRF模型使用维特比算法来寻找最优的标注序列。
完全连接的CRF是指每个像素都与其他像素相连,这样可以更好地捕捉像素之间的关系。在这篇论文中,完全连接的CRF被用作后处理策略,以进一步提高分割掩模的精度。
CRF模型的能量函数是一个关于标签变量的函数,它是由一组特征函数的加权和构成的,每个特征函数都定义在一个局部的标签配置上。这些特征函数可以是任意的函数,但通常是指示函数或者高斯核函数。
高斯双核是一种用于图像处理的方法,用于边缘检测和图像分割。它由两部分组成:高斯核和高斯拉普拉斯(LoG)核。高斯核用于平滑图像,而LoG核用于检测边缘。高斯核是一种低通滤波器,用于从图像中去除高频噪声。LoG内核是一个带通滤波器,它在抑制噪声的同时增强边缘。这两个内核的组合允许精确的边缘检测和图像分割。
2.基于强度的多器官腹部CT配准
相似度度量
对于非刚性配准,通过优化两幅图像之间的相似性度量来计算变形场,捕获的变形质量在很大程度上取决于度量的选择。差分平方和(SSD)、相关系数(CC)、顺序相似性检测算法(SSDA)和互信息(MI)是基于强度的配准方法的代表性相似性指标。然而,腹部医学图像的偏差通常是局部变化,在这种情况下,上述与全局计算的相似性指标可能会导致对变形场的错误估计。相较于 SSD 和 MI 来说,LCC是一个相当友好的折衷方案,它们的隐藏参数数量较少,但估计可靠。在本文中,将固定图像和运动图像分别划分为以每个体素为中心的斑块,并通过LCC计算这些图像之间的相关系数:

空间正则化
使用各向同性总变分正则化避免过平滑问题:

优化器
结合上述相似度量和空间正则化项,对腹部多器官配准的优化问题进行了研究。我们没有使用流行的梯度下降方法,而是使用乘数交替方向法 (ADMM)来最小化项,并使用搜索空间参数化(一阶 B 样条)优化配准框架以减小维度。这种优化方法可以避免物理上不可信的变换,并且对次优局部最小值具有更大的灵活性。

总结
本文提出了一种用于3D腹部CT图像的两步非刚性配准策略。在第一步中,使用改进的基于FCN的模型从CT图像中分割肝脏、肾脏和脾脏。然后,在第二步中,使用结合局部相关系数(LCC)相似性度量和各向同性总变差正则化项的方法来注册成对的腹部多器官区域。因此,该方法仅配准分割后的目标器官。得到的变形场用来对移动图像进行变形,以便将其与目标图像对齐。
第二篇论文链接:

一、摘要
腹部靶器官配准对于医学诊断和临床治疗至关重要,主要挑战来自器官结构和体积的复杂非刚性变形。本文首先提出一种改进的深度卷积神经网络(DCNN)来自动检测腹部计算机断层扫描(CT)目标器官感兴趣区域(ROIs),其中增加了CoordConv层以获取不同目标器官的更多坐标信息,并采用迁移学习技术在非医学数据库上进行预训练,以应对医学训练数据稀缺的情况。然后,通过基于强度的相异性测量结合标准 Tikhonov 正则化来记录成对目标器官 ROI。最后,在自建临床数据库和多个公共数据库上对所提方法进行了评价。

在临床实践中,治疗师通常关注目标器官或组织感兴趣区域(ROI)的注册,而不是全局图像空间的配准[10]。这些ROI可以用作节省配准时间和跟踪目标局部运动的先验过程[11]。然而,基于ROI的手动配准方法需要专家花费大量额外的时间来绘制目标区域的轮廓[12],[13]。因此,许多研究人员关注基于ROI的自动配准方法[14],[15],[16]。
CoordConvs和迁移学习等几种新技术在提高基于DCNN的检测方法的准确性方面表现出优异的性能[33],[34],[35],[36],[37]。标准卷积层通常缺乏全局和位置信息传输,这可以通过CoordConvs来解决。例如, [33]在基于DCNN的模型中采用了CoordConvs,以减少不同肺叶的错误分类,这比其他竞争模型更好。迁移学习首先在公开标注的大尺度自然场景图像上预训练模型进行特征学习,然后直接在小尺度医学图像数据上训练模型。该技术可以减少医学图像标记的时间消耗并提高模型的性能。例如,巴尔等人。 [38]使用迁移学习技术,通过在众多自然场景图像上训练基于DCNN的模型来检测医学图像的特征,并通过这种方式识别胸部X射线中的各种病理。受CoordConvs和迁移学习技术优势的启发,我们将其集成到我们的检测模型中,旨在减少标记工作的时间消耗,更好地从医学图像中学习复杂特征,从而获得更准确的分类和位置信息。
二、方法
这也是一个两阶段的方法:包括ROI检测和ROI配准,如图所示。对于ROIs检测,采用改进的基于DCNN的两阶段模型,输入腹部CT切片和输出靶器官ROI,而对于配准,结合基于强度的相异性度量和正则化项来记录检测阶段的输出数据。

2.1 基于 DCNN 的靶器官ROIs检测
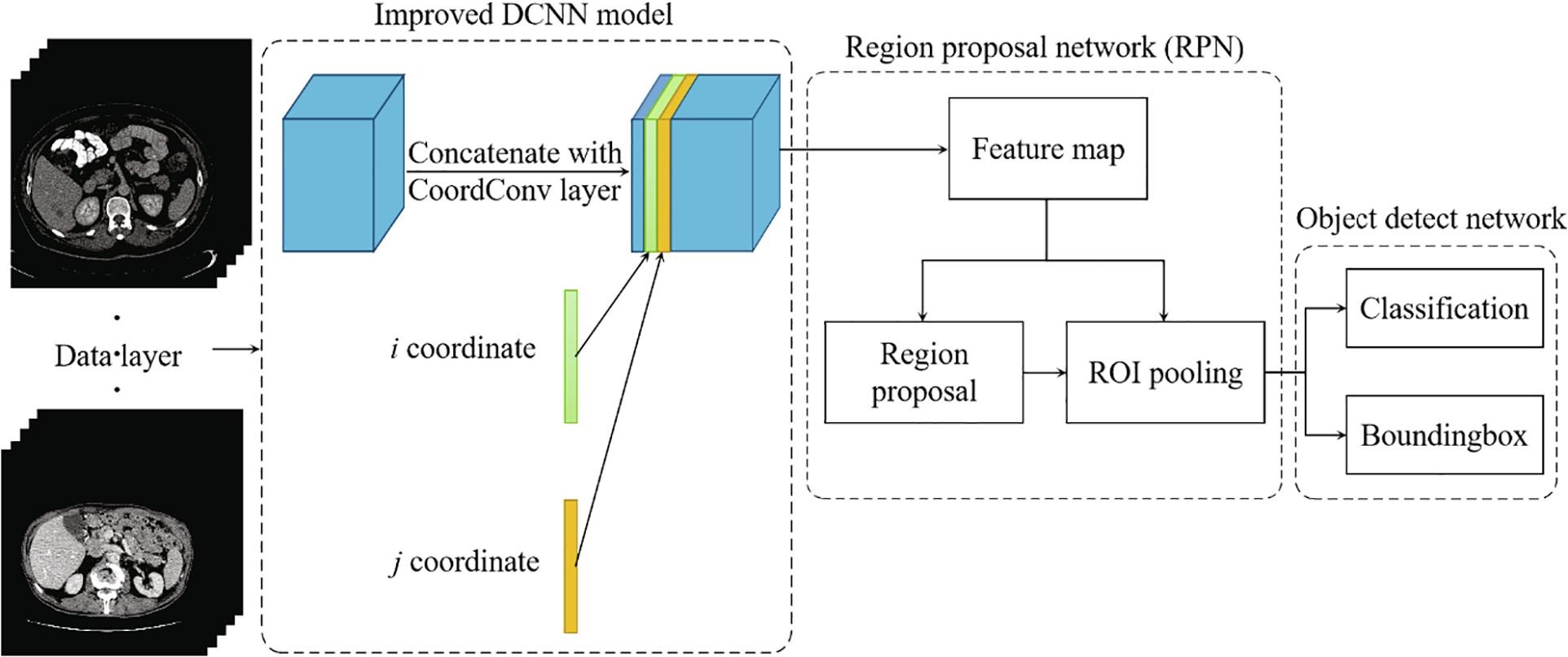
预训练的FasterCoordConv框架由三个模块组成:(a)用于特征学习的改进DCNN模型;(b) 潜在对象框的区域提案网络 (RPN),也称为ROIs生成;以及(c)区域目标检测网络提案。所提出的框架中的数据层包含原始尺寸的腹部CT图像。首先,我们使用改进的DCNN模型从数据层获取特征图。然后,RPN 将这些特征图作为输入,并预测对象的边界和分数以生成区域建议。最后,使用包含回归器和分类器的目标检测网络分别预测边界框并识别其类别。RPN 和对象检测网络都通过经济高效的交替训练技术共享一组大型通用卷积层。也就是说,RPN和目标检测网络首先分别训练,然后交替使用其中一个来微调另一个固定参数。
因为本部分属于目标检测方法,先略过。
2.2 基于强度的靶器官 ROI 配准
相异性度量
通常,平方差和(SSD)、绝对差分之和(SAD)、归一化相关系数(NCC)和归一化梯度场(NGF)被认为是基于强度的配准方法的相异性指标。其中,SSD通常用于单模态图像的配准。对于提取的ROI通常在目标器官周围有严重的强度干扰,SSD差异测量可能更适合ROI配准

正则化
使用标准的Tikhonov正则项

此外,在配准过程中引入了多分辨率策略,在不损失精度的情况下提高了配准速度。固定和运动图像分为三个分辨率级别(四分之一分辨率、半分辨率和全分辨率)。然后,配准过程从最低分辨率开始,计算相对较小的计算,将前一层的配准结果作为下一层的初始配准解,从而可以高效地完成整个从粗到细的配准。
实验
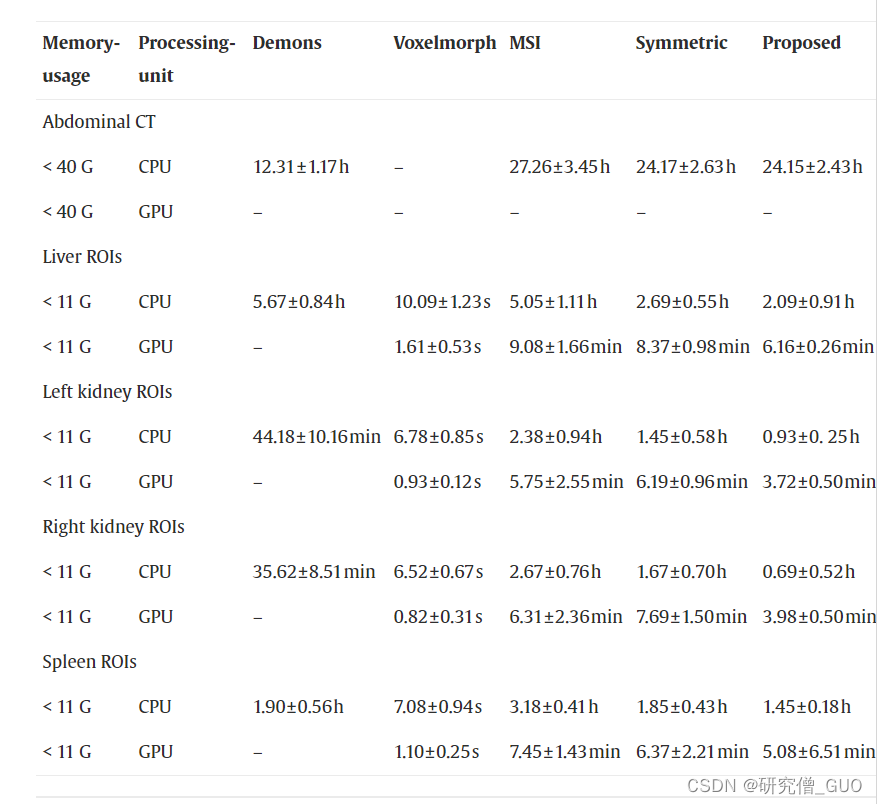
在不同的传统方法上进行原始成对腹部 CT 卷注册需要数小时到数天,因为它们需要大量的计算(内存使用量> 11G,使用多核 CPU)。此外,成对靶器官ROI的配准时间明显少于原始腹部CT体积的配准时间,因为时间消耗与图像大小成正比。例如,所提出的方法的肝脏ROI的平均注册时间为2.09±0.91小时(CPU),仅为原始腹部CT的十分之一左右。还可以观察到,提取目标ROI后,注册中计算的内存需求从40千兆字节(G)下降到11千兆字节(G),这意味着基于ROI的配准策略可以有效地节省单个GPU的计算时间(例如1080 Ti或2080 Ti,内存使用量= 11G)。因此,我们的两步策略解决了大尺寸图像不能通过Voxelmorph方法直接注册的问题。据统计,GPU 的使用可以为我们提供目标器官 ROI 配准速度的质的飞跃。例如,使用上述四种传统方法的GPU的肝脏ROI的注册速度比使用CPU的肝脏ROI快20至30倍。此外,该方法的耗时从3.72±0.50到6.16±0.26 min不等,小于其他传统方法。


总结
本文提出了一种基于ROI的非刚性腹部CT图像配准框架。首先,提出了一种改进的DCNN模型来检测目标器官ROI,其中将CoordConv层连接起来以增强坐标信息,并使用迁移学习技术预训练模型。然后,在成对的目标器官ROI上执行基于强度的非刚性配准。实验结果表明,原始成对腹部CT体积配准方法需要多核CPU(内存使用量> 11G)花费数小时到数天,而我们的方法在单个GPU上成功运行(内存使用量= 11G),仅需几分钟。此外,与一些竞争方法相比,所提出的方法在MSE、PSNR和SSIM的评估指标上具有最优性能,显示出良好的精度和鲁棒性。





















 7039
7039











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








