
MICCAI 2021 FLARE 挑战:快速和低 GPU 内存腹部器官分割
简介
腹部器官分割在临床实践中发挥着重要作用,在某种程度上,这似乎是一个已解决的问题,因为最先进的方法已经在几个基准数据集中实现了观察者间的性能。
然而,现有的腹部数据集大多只包含单中心、单阶段、单一机构或单一疾病的病例,并且尚不清楚是否可以将优异的性能推广到更多样的数据集上。
此外,许多 SOTA 方法使用模型集成来提高性能,但这些解决方案通常具有较大的模型尺寸和大量的计算资源,在临床实践中部署是不切实际的。
为了解决这些限制,我们组织了快速和低 GPU 内存的腹部器官分割挑战,该挑战具有两个主要特征:
- 数据集庞大且多样化, 包括来自 12 个医疗中心的 511 个病例。
- 我们不仅注重分割精度,还注重分割效率,符合实际临床实践和要求。
比赛任务
针对腹部各种期相的CT图像,同时实现肝脏、肾脏、脾脏和胰腺的分割。提供高效率低GPU(fast and low GPU memory)和高准确率(high accuracy)的解决方案。
比赛时间节点
- 2021 年 4 月 20 日(格林威治标准时间上午 12:00):注册开放!
- 2021 年 5 月 1 日(格林威治标准时间上午 12:00):发布训练和验证数据。Docker 和短论文提交验证集开放。
- 2021 年 7 月 31 日(格林威治标准时间上午 12:00): 验证提交截止日期。
- 2021 年 8 月 1 日(格林威治标准时间上午 12:00): Docker 和测试集开放的短论文提交。
- 2021 年8 月20 日(格林威治标准时间上午 12:00):测试提交的截止日期。
- 2021 年 10 月 1 日:将提供结果排名。
由此,可看出该比赛已经进入尾声了,虽然我们不能参加,但也可以学习优秀的解决方案,用于我们的分割任务。
主办方
马骏(南京理工大学)(提供4个基线模型)
顾松(南京信息工程大学)
张姚(中国科学院计算技术研究所;中国科学院大学)
Xingle An, 北京推想科技有限公司
王志和,深圳市海创医疗有限公司
王聪聪(天津工业大学)
程戈(江苏工业大学)
董国强 (南京大学)
朱琼杰(南京大学)
何健(南京大学)
杨小平(南京大学)
数据集
数据包括训练集(361例,全部为门脉期)、验证集(50例,各种期相)和测试集(100例,各种期相)。
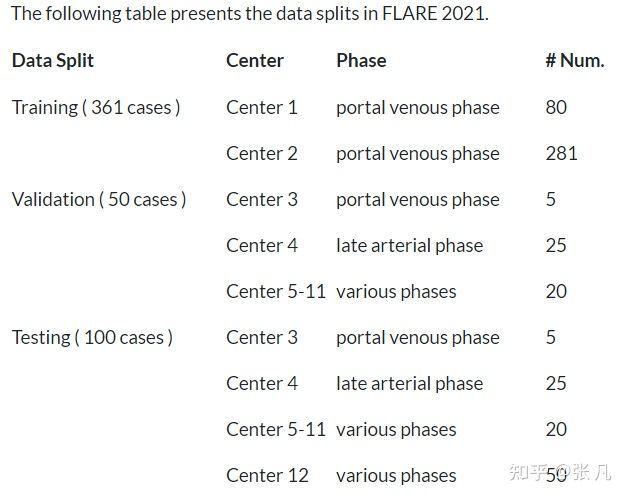
不同期相差异解释参考 CT 不同期相说明
评价指标
- Dice Similarity Coefficient
- Normalized Surface Dice
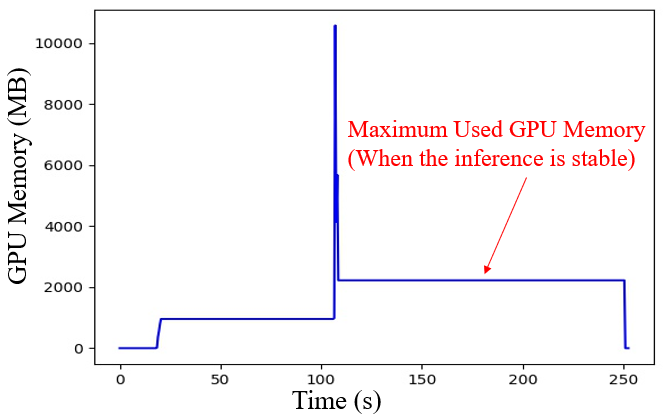
- Running time
- Maximum used GPU memory (When the inference is stable)
知乎作者评价
数据方面:
从以下角度进行统计分析,图像spacing\size\orientation,各个器官的形状、密度、上下文等特征,阴阳性的比例,badcase特点。
算法方面:对算法的pipeline和网络结构进行优化,采用分块/整图、一阶段/多阶段的pipeline,设计高效率的网络结构。解决病理性图像分割失败的问题,提升不同期相的泛化问题。
本次竞赛追求高效率和高准确率的解决方案,我们需要从算法流程上降低推理时间,从网络结构上降低模型的显存占用。针对数据的分布和分割目标的特点,提升分割的准确率。
分割的难点:
训练集、验证集和测试集数据分布差异较大,包括期相、阴阳性的比例等
不同器官的形状、大小分布差异较大,存在不同扫描范围的数据
训练三维的分割模型存在显存不足的问题
优化的出发点:
采用多阶段算法(定位+分割)解决扫描范围的差异
采用均衡的数据采样方法解决分布不一致的问题
采用对比度增强方法,消除不同期相带来的差异
参考实时语义分割场景,设计高效率分割网络
对上下文特征进行学习,提升病理性分割的准确率
采用混合精度训练、分布式训练、模型并行等方法,提升显存利用效率
基线模型
我们(主办方)进一步为全监督、半监督、弱监督和持续学习构建了四个器官分割基准,这些是目前具有挑战性和活跃的研究课题。
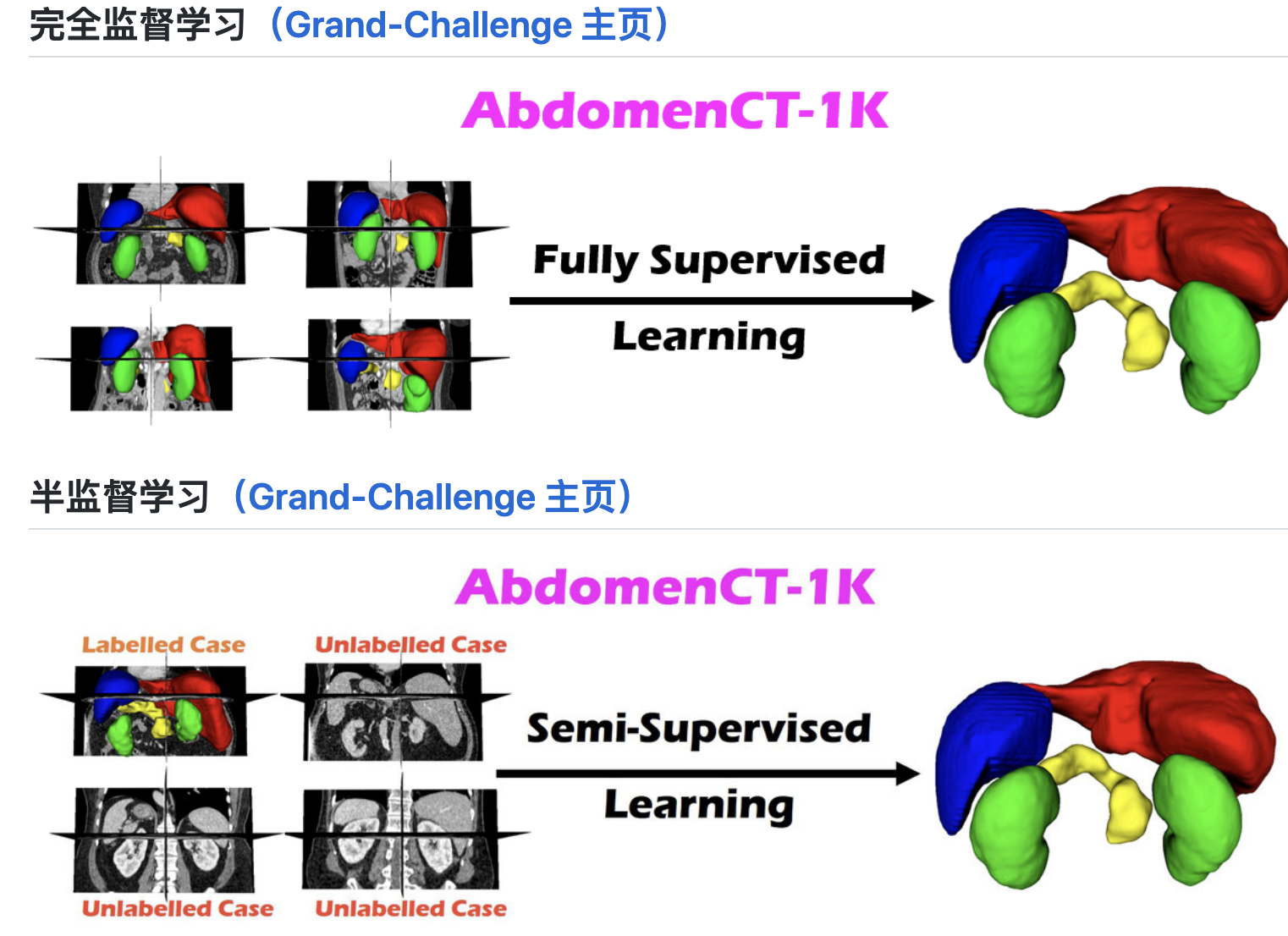
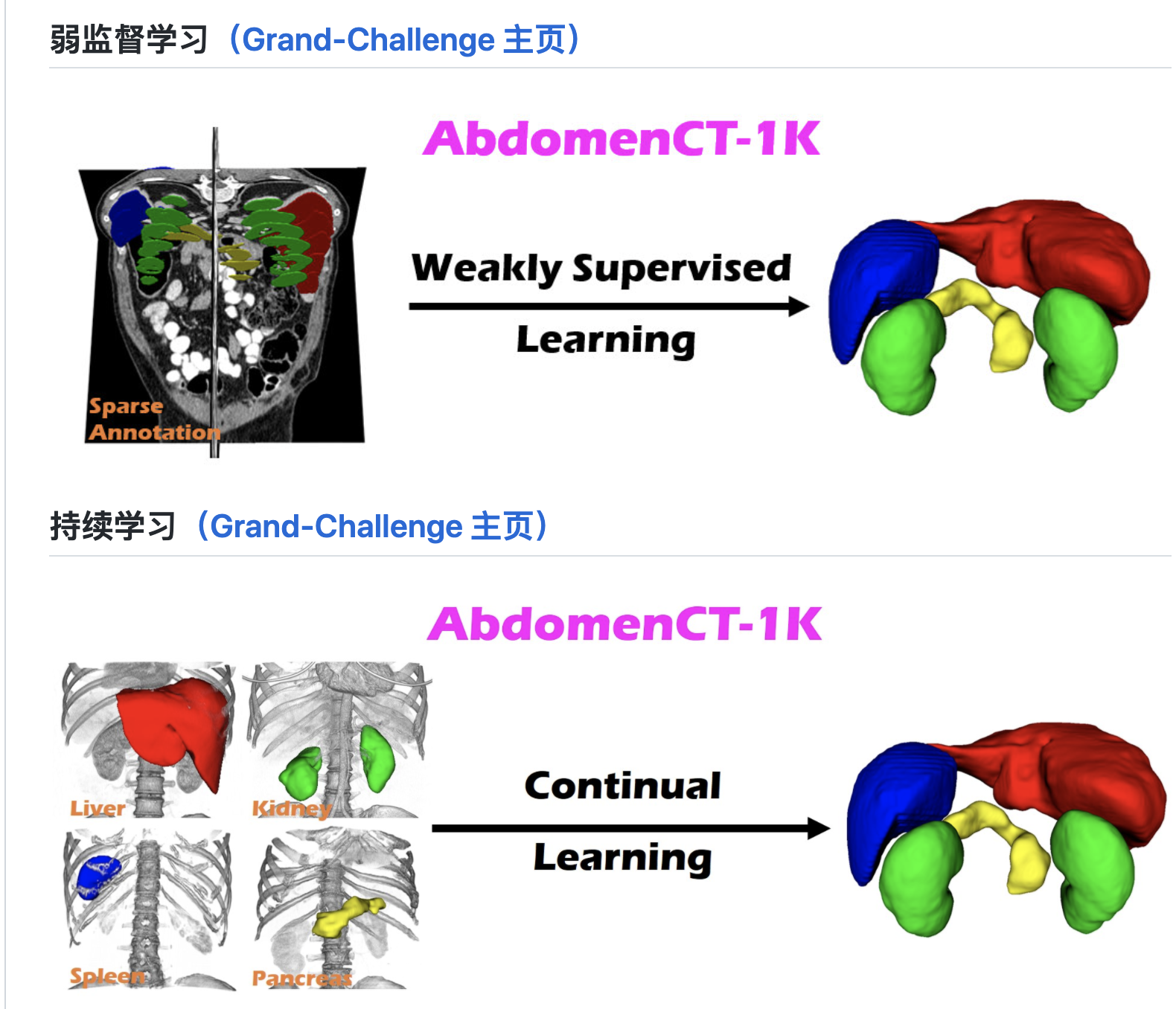
此外,作者还给出了50例12个注释器官
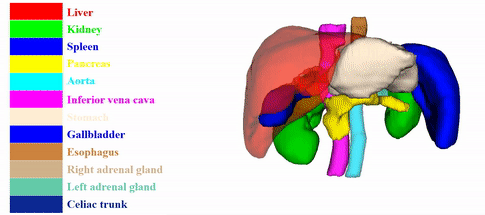
以上基线模型的代码参考链接:
彩蛋
主办方邀请到nnUNet作者【Fabian Isensee 】和 UNet++, ModelGenesis作者【Zhongwei Zhou】在 MICCAI FLARE 分别分享关于nnUNet背后的故事和高效利用医学数据的相关工作:
- nnU-Net - the baseline, the tool, the framework. Insights, analysis and future directions
- Data, Assemble: Towards Efficient Medical Image Analysis
论文解读- nnU-Net: Self-adapting Framework for U-Net-Based Medical Image Segmentation)
MICCAI FLARE将在北京时间 10.1日 17-21点举行,欢迎大家届时参加。(没找到直播链接,应该是有的)
相关的链接:
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连
























 457
457











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










