Deep-DRM a computational method for identifying disease-related metabolites based on graph deep learning approaches论文解析(Deep-DRM:一种基于图形深度学习方法的识别疾病相关代谢物的计算方法)由哈尔滨医科大学 发表在《Briefings in Bioinformatics》
动机: 基因、核糖核酸和蛋白质的功能变化最终将在代谢水平上被选择。越来越多的研究者通过代谢物来研究其机制、生物标志物和靶向药物。然而,与我们对基因、RNAs和蛋白质的了解相比,我们对疾病相关代谢物的了解仍然很少。现有的几种识别疾病相关代谢物的方法都忽略了代谢物的化学结构,未能识别代谢物与疾病之间的关联模式,也未能应用于孤立的疾病和代谢物。
结果:在这项研究中,我们提出了一种基于图形度学习的方法,称为Deep-DRM,用于识别疾病相关的代谢物。首先,代谢物的化学结构被用来计算代谢物的相似性。根据疾病的功能基因网络和语义关联获得疾病的相似性。因此,代谢物和疾病网络都可以建立。接下来,使用图卷积网络(GCN)分别对代谢物和疾病的特征进行编码。然后,利用主成分分析对这些特征进行降维,保留了99%的信息。最后,基于这些特征建立了深层神经网络用于识别真正的代谢物-疾病对。与以前的方法和类似方法相比,在三个测试设置上的10个交叉验证显示了Deep-DR,的突出AUC (0.952)和AUPR (0.939)。前15个预测的疾病和代谢物之间的关联中有10个得到了其他研究的支持,这表明Deep-DRM是一种有效的识别MDPs的方法。
源码:https://github.com/zty2009/GPDNN-for-Identify-ing-Disease-rela
ted-Metabolites
代谢是伴随整个生命周期的重要生化反应,极易发生和发展疾病,进而导致血液和尿液中代谢物的异常[1]。上游的功能变化(核酸、蛋白质等。)大分子最终会在代谢中层面中得到反映,如神经递质的变化、激素调节、受体效应、细胞信号释放、能量传递和细胞间通讯等,所以代谢位于基因调控网络和蛋白质相互作用网络的下游,并提供生物学的末端信息。因此,基因组学和蛋白质组学告诉我们可能会发生什么,代谢组学告诉我们发生了什么[3]。
利用代谢组学数据研究疾病也有以下优势[4]。首先,功能水平上基因和蛋白质表达的微小变化将在代谢物上扩增,这使得检测更容易。第二,基因和蛋白质的许多非功能性变化不会在代谢产物上体现出来,使得代谢产物在向下游传递上游信息的过程中起到‘噪声过滤’的作用;第三,代谢物的种类比基因和蛋白质的数量少得多,物质的分子结构也简单得多,所以通过代谢物研究疾病更容易。此外,常见的代谢产物在各种生物系统(如植物、微生物、动物的初级代谢)中是相似的,因此代谢组学研究中使用的平台技术可以应用于不同的生物系统。
近年来,越来越多的研究者致力于通过代谢物发现疾病机制、生物标志物和靶向药物。Mathewson等人[5]发现丁酸盐修复可以减少肠上皮细胞凋亡,减轻移植物宿主病(GVHD)。他们还指出,微生物代谢物的局部和特异性改变对GVHD靶组织有直接的有益作用,并能减轻疾病的严重程度。马丁内斯-雷耶斯和钱德尔[6]声称,三羧酸循环(TCA循环)是有氧生物中普遍存在的代谢途径,与癌症、免疫和干细胞功能有关。常等人[7]发现克罗恩病相关代谢物中有15种代谢物具有CD4+ T细胞生物活性,相关T细胞的生物活性相当高。此外,研究人员还发现了心血管疾病[8]、神经退行性疾病[9]、肝病[10]、神经免疫性疾病[11]、慢性肾脏疾病[12]等多种疾病。与代谢物有关。
总的来说,代谢物显示了它们帮助理解和对抗疾病的强大力量。然而,与基因、核糖核酸和蛋白质相比,人们对疾病相关的代谢物知之甚少。这主要是两个原因造成的。(1)可通过基于非目标质谱的代谢组学技术识别的人类代谢物的比例通常小于已识别质谱峰的2%[13]。ii)确定每种疾病的相关代谢物既费时又费钱。对于第一个问题,一些非常精确的开放存取和用于电子代谢物和光谱预测的商业工具最近已经可用[14]。然而,对于第二种方法,很少开发出用于识别疾病相关代谢物的工具。
2018年,我们基于疾病的相似性构建了代谢物相似性网络,并通过随机行走(RW)预测了疾病相关的代谢物[15]。根据我们的研究,王等人[16]将文本挖掘分数加入到代谢物相似性网络中,并应用RW遍历该网络以获得疾病的潜在相关代谢物。类似地,雷和铁[17]提出了“MDBIRW”,使用高斯相互作用剖面(GIP)计算疾病相似性,然后推断代谢物相似性网络。此外,还实现了双随机行走来遍历疾病和代谢物网络。
然而,这些方法都有三个缺点。(一)代谢物的相似性仅根据其对应疾病的相似性计算,忽略了代谢物的化学性质。(二)这些方法的原理都是基于“相似代谢物与相似疾病相关联”的假设,没有认识到代谢物与疾病之间的关联模式。(三)这些方法不适用于网络中的孤立疾病/代谢物。具体来说,没有相应疾病的代谢物与其他代谢物之间的相似性无法计算,没有已知相关代谢物的疾病的潜在代谢物也无法预测。
因此,我们提出了一种新的方法“Deep-DRM”来克服这些缺点。首先,代谢物的相似性是基于它们的化学结构获得的。然后,使用图卷积网络(GCN)对代谢物和疾病网络进行编码。因为代谢物是蛋白质的底物或产物,所以代谢物的特征由它们相应的蛋白质通过独热编码来编码。应用主成分分析对特征进行降维。最后,将这些特征输入到深度神经网络(DNN)中,以识别代谢物和疾病之间的关联模式。此外,我们的方法还可以孤立的疾病/代谢物识别相应的疾病和代谢物。
我们提出了一种新的方法,称为Deep-DRM,它是融合GCN,主成分分析和DNN,以确定潜在的疾病相关的代谢物。Deep-DR,包括三个步骤(图1)。 
Metabolites network
化学性质是代谢产物参与生化反应的最重要特征。软件‘PaDEL-Descriptor’[18]的开发给了我们一个基于化学结构计算分子描述符和指纹的机会。它可以为我们提供代谢物的化学性质,如原子型电拓扑状态描述符、克里平的logP和磁共振以及扩展的拓扑化学原子描述符。
1D&2D描述符和指纹是通过帕德尔描述符为每种代谢物计算的。一个2325维的向量(1D&2D描述符为1441,指纹为881)被用来描述每种代谢物的化学性质。由于维度的尺度不同,每个维度都需要归一化。z分数标准化应用如下:
其中,mk表示标准化后ith代谢物的kth维数,mean(mk)表示所有代谢物的原始kth维数的平均值,std(mk)表示所有代谢物的原始kth维数的标准差。
然后,代谢物的相似性可以通过这些载体获得如下: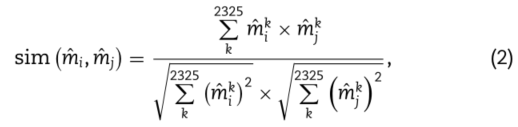
其中sim(mi,mj)表示ith代谢物和jth代谢物之间的相似性。最后,可以构建代谢物网络,其中代谢物是节点,相似性是网络的边缘。
Diseases network
疾病之间的相似性源于疾病之间的语义关联和疾病相关基因之间的功能关联。semfonsim[19]是我们以前提出的一种方法,它不仅使用疾病相关的基因集来计算人类基因功能加权网络中的疾病相似性,还使用疾病本体(DO)中两种疾病之间的关系来计算疾病相似性。最后将两种相似性结合在一起,得到疾病的最终相似性。
一对基因之间的功能相似性得分被定义为FunSim(gi,gj)
其中LLSN(gi,gj是通过人类网络中的对数似然分数(LLS)来测量基因之间功能连接的概率。然后,我们把一个基因G和一个基因集G = {g1,g2,,gk}之间的功能关联定义为FG(g)。![]()
其中k表示g中的基因数量。
如果疾病D1与基因集相关,G1= {g11,g12,,g1m},如果疾病D2与基因集相关,G2= {g21,g22,,g2n}。D1和D2之间的相似性可以计算如下: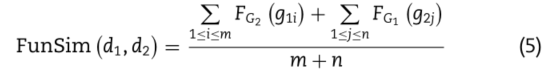
其中g1i∈ G1,g2j∈ G2,m是G1中的基因数,n是G2中的基因数。
然后,疾病对D1和d2之间的语义相似度可以通过等式(6)来计算。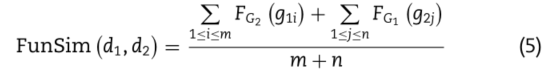
其中gmi care在DO的有向无环图(DAG)中呈现D1和D2的信息最丰富的共同祖先(MICA)。
最后,疾病的相似性是FunSim()和SemSim()的产物。![]()
通过这种方式,可以构建疾病网络,其中疾病是节点,相似性是网络的边。
建立代谢物和疾病网络后,两个网络的每个节点都应该包含其内在特征。然后,GCN可以根据网络结构和节点特征对网络进行编码。最后,通过主成分分析可以实现降维。
Feature extraction
代谢物是蛋白质的底物或产物,因此它们与蛋白质的关系可以代表它们的特征。
独热编码用于编码代谢物的特征。假设存在k蛋白,那么代谢物的特征可以表示如下:![]()
其中Fm表示代谢物的特征,Pi表示代谢物和第ith蛋白之间的关系(1< i < k)。如果代谢物是ith蛋白的底物或产物,则Pi= 1,否则Pi= 0。
由于疾病的相似性已经包含了足够的信息,我们只使用一个独热编码来编码疾病的索引。
假设有n种疾病,那么疾病的特征可以表示如下:![]()
这是疾病的特征。D1到Dn中,只有Di= 1,其他元素都是0。
Graph encoding
GCN算法是一种神经网络算法,可以直接提取网络的结构信息和节点信息。它已被广泛应用于生物信息学的许多不同领域[20,21]。
接矩阵可以分别从疾病和代谢物网络中获得。描述节点之间的连接。
由于代谢物和疾病的特征应包含各自的信息,因此应进一步处理邻接矩阵。![]()
其中I是单位矩阵。
下一步是求逆矩阵D′。![]()
最后,网络的特征可以提取如下: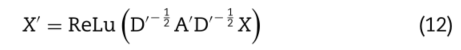
其中X是每个节点的信息。对于代谢物网络,X可以通过公式(8)获得。对于疾病网络,X可以通过公式(9)获得。另外,ReLu是整流后的线性单位。其公式是![]()
最后,通过GCN编码,分别得到代谢物和疾病的特征。
Reduction of dimension
由于蛋白质和疾病的数量很大,代谢物和疾病的规模也很大。因此,引入主成分分析(PCA)来降低特征维数。
作为一种公认的方法,我们就不详细解释PCA的过程了。我们保留了代谢物和疾病的99%的特征信息。
3.Classification of metabolites-diseases pairs
由于代谢物和疾病的特征都可以通过上述处理表示为一个向量,因此我们通过组合它们的特征来构建代谢物-疾病对的特征(MDP)。
MDP的特点可以表述如下:![]()
其中Fm1,Fm2,.。。,Fmi i和 Fd1,Fd2,.。。Fdj i是代谢物和疾病的特征,分别在GCN和PCA编码之后,而FMDP是MDP的特征。如公式(14)所示,MDP的特征是相应的代谢物和疾病的组合。
然后,我们可以将MDP的特征及其标签输入DNN,以识别真正的MDP。我们建立了一个有四层的DNN模型。参数如表1所示。
DNN模型有两个输出。一个是测试MDP为真的概率,另一个是为假的概率。
Dataset
人体代谢组学数据库(HMDB) [13]是关于人体代谢组学的最全面的网络资源。我们从HMDB获得了代谢物和疾病之间的联系。此外,还从HMDB获得了代谢产物的化学结构和相关蛋白。
我们从DO中总共获得了3524种疾病来计算疾病的相似性。
我们把任务分成三个目标。第一个目标是确定已知代谢物和已知疾病之间的新联系。“已知”是指代谢物/疾病具有相关的疾病/代谢物。我们称这个为aim Ki。第二个目的是确定已知代谢物和未知疾病之间的联系。“未知疾病”指没有已知相关代谢物的疾病。我们称这个目标为Ud。最后一个目的是确定未知代谢物和已知疾病之间的联系。“未知代谢物”代表那些没有已知相关疾病的代谢物。我们称这个目标为Um。表2显示了在这三个目标中使用的数据数量。
如表2所示,我们从HMDB获得了1436种代谢物和242种疾病之间的3124种已知关联。因此,这些代谢物与疾病之间未知的关联应为1436×242 3124 = 344 388。对于aim Ud,我们只选择了与242种已知疾病中至少一种相似度高于0.3的疾病,因为疾病与242种已知疾病的关系越密切,我们的方法就越有可能准确找到与其相关的代谢物。最后,我们从3524种疾病中发现了160种具有高潜力的未知疾病。因此,未知关联数应为160 × 1436 = 229 760。对于目标Um,我们选择了与至少120种蛋白质相关的代谢物作为未知代谢物,因为代谢物和蛋白质之间的关联越多,代谢物参与疾病相关生化反应的可能性就越大。最后,从HMDB的114 100个代谢物中获得了857个未知代谢物。因此,未知关联数应为857 × 242 = 13 794。
Training and testing
为了Deep-DRM的有效性,我们分别对三个目标进行了10次交叉验证。
由于未知MDP的数量远远大于已知MDP的数量,我们从具有相同数量的正集合(3124个样本)的未知MDP中随机选择负样本。因此,对于每个目标,3124个阳性样本和3124个阴性样本被构建为新的数据集。然后,我们对这个新数据集进行了10次交叉验证。
为了测试Deep-DRM的稳定性,上述所有过程(通过随机选择负样本和10次交叉验证来构建新数据集)针对每个目标重复5次
1.Verifying the validity of Deep-DRM
由于我们的方法是融合GCN,PCA和DNN,我们比较了Deep-DRM与两个类似的方法。由于降维功能,选择了主成分分析。然而,由受限玻尔兹曼机(RBM)构造的深度信念网络(DBN)也具有降维能力。为此,我们融合了GCN、DBN和DNN的思想,构建了一种新的方法——广义递归神经网络。我们将Deep-DRM与GRDNN进行了比较,以显示主成分分析和DBN之间的差异。然后,为了展示GCN的力量,我们只使用了主成分分析和DNN来确定MDPs。我们称这种方法为“PDNN”。
为了展示这三种方法的性能,我们绘制了图2。
如图2所示,所有这三种方法都在三个目标上进行了测试。红色条表示Deep-DRM的性能,蓝色和绿色条分别表示GRDNN和PDNN。由于我们通过构建不同的数据集重复了5次10交叉验证,误差条显示了曲线下面积和精确召回曲线下面积的标准偏差(AUPR)。深度数字版权管理在三个目标中表现最好,稳定性高,GRDNN最差。通过对Deep-DRM和GRDNN的对比实验进行分析,可以看出DBN的性能比主成分分析差,因为DBN提取了高度抽象的特征,这使得它比Deep-DRM和PDNN更适合于图像处理,代谢物和疾病的相似性在识别代谢物和疾病的关联模式中起着重要的作用。
总的来说,Deep-DRM在这个比较实验中显示了较高的AUC和AUPR,这表明了它识别潜在的多学科设计方案的有效性。
2.Comparison with previous methods
代谢物作为生物过程的最终产物,是非常有前途的生物标志物,也是理解发病机制的重要组成部分。然而,尽管人们对疾病相关基因、核糖核酸和蛋白质有了很多了解,但迄今为止对疾病相关代谢物的了解却很少。
虽然已经开发了一些基于相对湿度的计算方法来识别疾病相关的代谢物,但是它们都存在诸如不能有效利用代谢物的化学特性、不能识别分离的代谢物/疾病相关的疾病或代谢物等问题。
1、本文利用代谢物的化学结构计算代谢物的相似性,构建代谢物网络。根据疾病的基因网络和语义获得疾病的相似性,构建疾病网络。然后使用了GCN对网络进行编码,PCA用来降维,DNN用来分类。
2、作者在使用图卷积来提取特征的时候,并没有使用AXW的公式,一般图卷积分为两步,AX聚合节点的邻居信息,然后乘可训练参数W来映射特征。作者只用了AX,然后在使用PCA降维,相当于作者前半部分在做的时候,其实并没有一个需要训练的部分,和后面在拼接成节点对特征,应用分类器没有一个整体。效果可能还有提升的空间。如果只使用AX就有点类似矩阵分解的感觉了。
3、按照我之前来做的看的话,如果先分别对代谢物和疾病来进行提取特征,在构造节点对。没有先构造节点对,在提取特征效果会更好。
4、技术算不上新颖吧,算是图卷积在disease-related metabolites上的一个新的应用。
5、随着图神经网络的发展,越来越多的使用图神经网络在生物网络上,从之前写的几篇文章中也可以看出来。





















 1098
1098











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










