在看单细胞文章的时候,总会在文章里面碰到一些非常酷炫的热图,比如说下面这张图来自于最近的NBT上单细胞ATAC-seq的聚类展示。
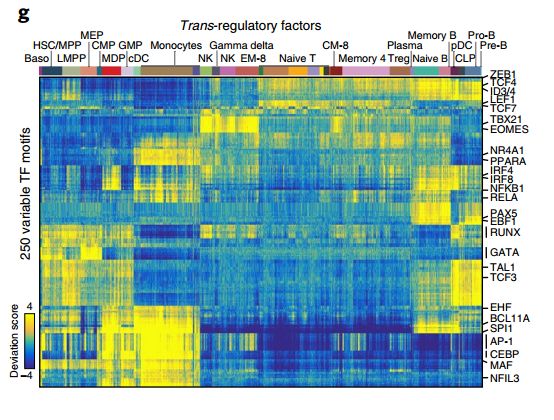
但是用Seurat自带的热图函数DoHeatmap绘制的热图,其实是没有这个效果。于是我尝试使用ComplexHeatmap这个R包来对结果进行展示。
个人觉得好的热图有三个要素
聚类: 能够让别人一眼就看到模式。如果本来就无法聚类,那图也不好看。
注释: 附加注释能提供更多信息,比如说一些标记基因名
配色: 要符合直觉,比如说大部分都会认为红色是高表达,蓝色是低表达
在正式开始之前,我们需要先获取一下pbmc的数据,Seurat提供了R包SeuratData专门用于获取数据
devtools::install_github('satijalab/seurat-data')
library(SeuratData)
InstallData("pbmc3k")
加载数据并进行数据预处理,获取绘制热图所需的数据
library(SeuratData)
library(Seurat)
data("pbmc3k")
pbmc pbmc[["percent.mt"]] "^MT-")
pbmc 200 & nFeature_RNA < 2500 & percent.mt < 5)
pbmc "LogNormalize", scale.factor = 10000)
pbmc "vst", nfeatures = 2000)
all.genes pbmc all.genes)
pbmc pbmc 1:10)
pbmc 0.5)
pbmc.markers only.pos = TRUE,
min.pct = 0.25,
logfc.threshold = 0.25)
先感受下Seurat自带热图
top10 % group_by(cluster) %>% top_n(n = 10, wt = avg_logFC)
DoHeatmap(pbmc, features = top10$gene) + NoLegend()
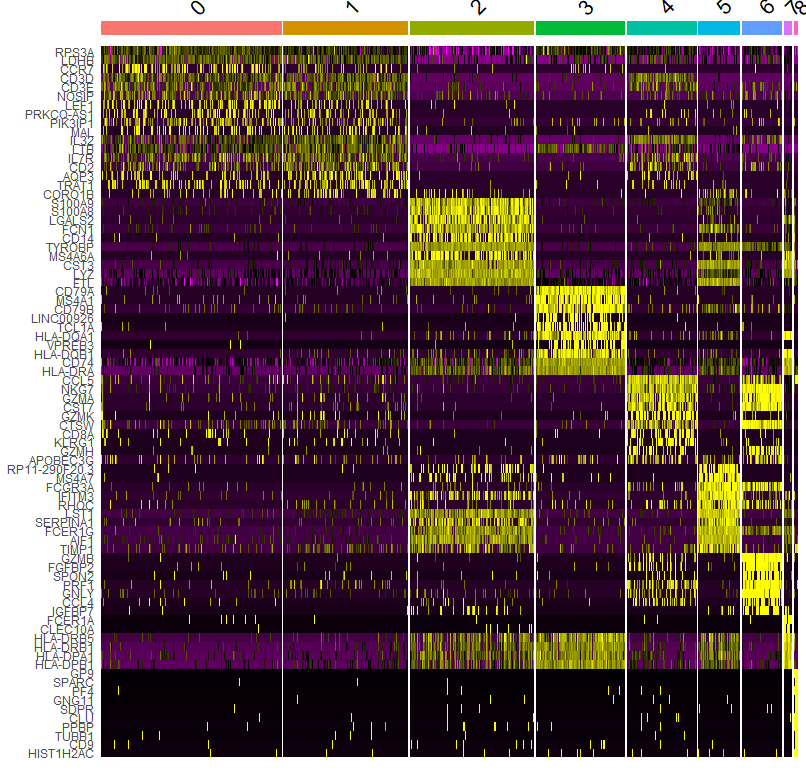
作为探索性分析,这张图是可用的,但是可能无法直接用于最后文章的展示。
下面则是介绍如何用R包ComplexHeatmap进行组图,虽然这个R包名带着Complex,但是并不是说这个R包很复杂,这个Complex应该翻译成复合,也就是说这个R包能在热图的基础上整合很多信息。
先安装并加载R包。
BiocManager::install("ComplexHeatmap")
library(ComplexHeatmap)
为了手动绘制一个热








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 681
681











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








