
点击关注,桓峰基因
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
124篇原创内容
公众号
OncodriveCLUST 利用体细胞突变的位置聚类来识别癌症基因
摘 要
研究动机**:**功能获得突变通常聚集在特定的蛋白质区域,这表明这些突变为癌细胞提供了适应性优势,因此在肿瘤的克隆进化过程中被积极选择。我们试图确定该特征在癌症中的总体程度,以及使用该特征识别司机的可能性。
**结果:**我们开发了OncodriveCLUST,一种识别蛋白质序列中具有显著突变聚类倾向的基因的方法。该方法通过评估编码沉默突变来构建背景模型,这些突变被认为不是正选择,因此可以反映体细胞突变聚类的基线趋势。OncodriveCLUST对《癌症体细胞突变目录》的分析检索了癌症基因普查丰富的基因列表,优先考虑了那些具有显性表型的基因,但也突出了一些隐性癌症基因,这些基因显示出更广泛但仍有限制的突变集群。对癌症基因组图谱数据集的评估表明,OncodriveCLUST选择了基于频率和功能影响标准的方法所遗漏的癌症基因。这强调了结合基于互补原则的方法来识别驱动突变的好处。我们建议OncodriveCLUST作为实现这一目的的有效工具。

执行的步骤的示意图表示OncodriveCLUST评估两个虚拟基因的突变聚类主要步骤分为5步:
(I) 输入数据以影响蛋白质的基因突变直方图表示,x轴为蛋白质序列位置,y轴为该位置发生的基因突变的百分比。
(II) 有意义的位置被选择为那些具有大量突变且不被偶然预期的位置。这是根据二项累积分布函数对每个基因的长度和突变数量进行计算的,根据我们的突变非均匀分布假设,这是一个松散的标准(默认51%的概率将某个位置表示为有意义的)。在本图中,作为有意义阈值的每个基因计算的最小突变数由标记为Th的虚线表示。(III) 对步骤(II)中发现的有意义的位置进行分组,形成突变簇(在展示的面板中灰色阴影部分)。如果两个连续的位置之间的默认最大距离为5个氨基酸,则它们被分组在同一簇中。这被认为是一个严格的分界点,以定义不同残基上的两个突变影响相同的致瘤机制,因为事实强调,大多数(95%)编码沉默突变,假设是随机分布的,在COSMIC数据集被较大的距离分开。注意,如果在整个蛋白质序列中没有发现有意义的位置,则认为该基因没有突变簇,因此不进行进一步的分析。在本例中,基因A有一个有意义的位置,因此它形成一个单簇;在B基因中,有6个有意义的位置,它们被组合成两个单独的簇。
(IV) 没有发生在(II)中有意义的位置,但在(III)中定义的集群边界内或与其相邻的突变也应计入该集群所包含的突变数量的最终数字。
(V) 之后,对每个聚类进行评分。这个分数如图1所示。继续与封闭突变的数量成正比,与簇长度成反比(参见方法部分了解更多关于聚类分数计算的细节)。每个基因的最终值是通过将该基因中发现的每个聚类的得分相加得到的(基因A为C1,基因B为C1 + C2)。
(VI) 最后,为了估计(V)中每个基因得分的显著性,将其与背景模型的分布进行比较。背景模型是通过计算所有数据集基因的聚类分数,然后分析编码沉默突变得到的。注意,这种比较可以在背景模型的正态分布得到验证后获得一个标准分数(即Z分数),然后得到一个校正后的P值。其实图示如下:

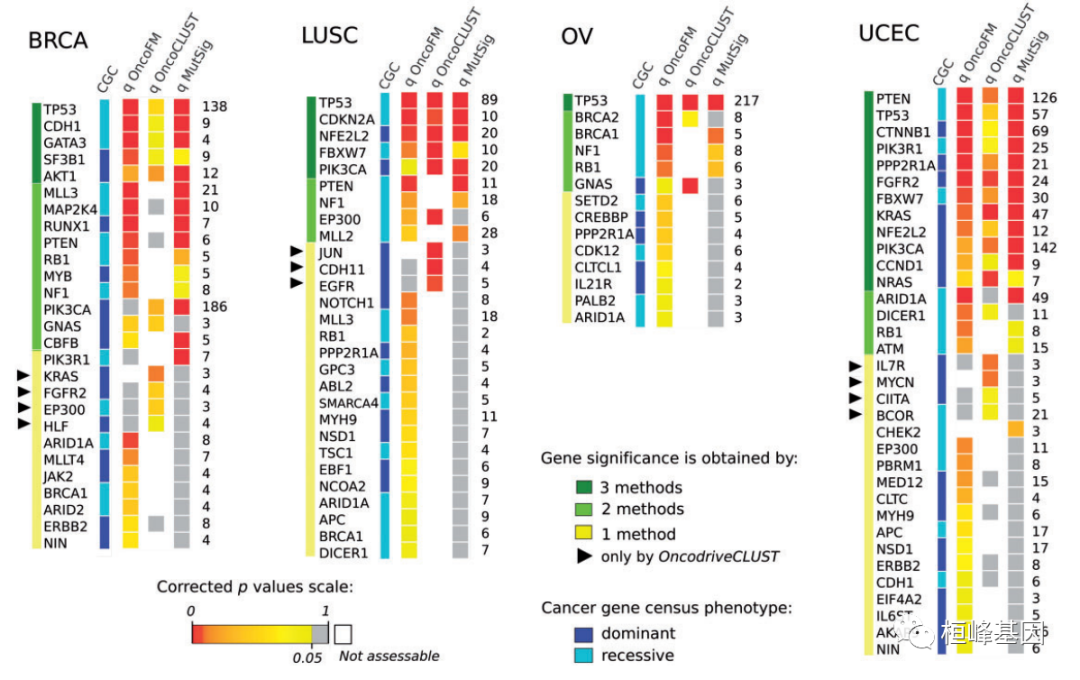
从对COSMIC的OncodriveCLUST分析中选择top基因,如下:

软件安装
OncodriveCLUSTL依赖于Python 3.5和一些外部库。我们建议使用Anaconda Python发行版来安装:
$ conda install -c bbglab oncodriveclustl
OncodriveCLUSTL也可以使用pip来安装:
$ pip install oncodriveclustl
也可以从存储库获取最新的代码,并使用pip安装。这里出现一个bug,大家可以参考,就是git clone时出现如下bug:
git clone git@bitbucket.org:bbglab/oncodriveclustl.git
Cloning into 'oncodriveclustl'...
git@bitbucket.org: Permission denied (publickey).
fatal: Could not read from remote repository.
问题原因:
permission denie(publickey) 翻译过来就是公匙不对导致不能下载,我们知道通过ssh的方式去下载的话,是需要持有私匙和公匙,才能正常下载。
这个地方提示的就是ssh key缺失的问题,那么有的小伙伴可能疑惑说,我明明配置了,为什么不能使用,其实我刚开始也是这么想的,但是忽略了一个问题,我要下载的包在github,而项目是存放在gitlab上的,这两个是不通用的。在登录github上进行配置一下即可。
解决方案:
1、生成ssh key
在我们的 Git Bash here 打开的控制台上面进行命令操作:
-
先输入ssh-keygen -t rsa -C “你的github账号”
-
然后选择保存key的位置,基本就是默认C:/Users/admin/.ssh 目录下面的id_rsa文件(如果已经存在这个文件的话,可以选择其他位置)
-
输入你的passphrase,这个应该可以理解为密码,正常我这边是直接回车,就是没有密码
-
重新确认一下passphrase
-
然后会生成你的rsa文件。
2、复制ssh key
3、登录github
4、new ssh key
没有此类问题的可以直接运行下面安装软件:
$ git clone git@bitbucket.org:bbglab/oncodriveclustl.git
$ cd oncodriveclustl
$ conda install -c anaconda pycurl
$ pip install .
当你第一次使用给定的参考基因组运行OncodriveCLUSTL时,将从他们的服务器下载。默认情况下,下载的数据集位于~/.bgdata。如果希望将这些数据集移动到另一个文件夹,则必须使用导出命令定义系统环境变量BGDATA_LOCAL:
$ oncodriveclustl --help
官网提供了测试数据集,下载方式如下:
$ curl -o oncodriveclust.tar.gz https://bitbucket.org/bbglab/oncodriveclust/get/0.3.tar.gz
#或者
wget -c oncodriveclust.tar.gz https://bitbucket.org/bbglab/oncodriveclust/get/0.3.tar.gz
$ tar -xvf oncodriveclust.tar.gz
$ cd bbglab-oncodriveclust-60e75d0cb432
###文件还有的文件
$ ls
agpl-3.0.txt
data/
distribute_setup.py
examples/
.gitignore
logo.png
oncodriveclust/
README.md
setup.py
###exmples
tcga.BRCA.nonsyn.txt
tcga.BRCA.syn.txt
$ less tcga.BRCA.nonsyn.txt
symbol gene transcript Sample ct position
SCYL3 ENSG00000000457 ENST00000367772 tcga_Breast_invasive_carcinoma_TCGA-AR-A0U4-01A-11D-A117-09 non-synonymous 136
SCYL3 ENSG00000000457 ENST00000367772 tcga_Breast_invasive_carcinoma_TCGA-C8-A12U-01A-11D-A10Y-09 non-synonymous 571
$ less tcga.BRCA.syn.txt
CFH ENSG00000000971 ENST00000367429 tcga_Breast_invasive_carcinoma_TCGA-AO-A0J2-01A-11W-A050-09 synonymous 498
CFH ENSG00000000971 ENST00000367429 tcga_Breast_invasive_carcinoma_TCGA-BH-A0HA-01A-11D-A12Q-09 synonymous 706
LAS1L ENSG00000001497 ENST00000374811 tcga_Breast_invasive_carcinoma_TCGA-B6-A0I8-01A-11W-A050-09 synonymous 701
SEMA3F ENSG00000001617 ENST00000002829 tcga_Breast_invasive_carcinoma_TCGA-AR-A1AY-01A-21D-A12Q-09 synonymous 87
###data
$ ls
CGC_phenotype.tsv
gene_transcripts.tsv
$ less CGC_phenotype.tsv
Symbol Cancer Molecular Genetics
ABL1 Dom
ABL2 Dom
ACSL3 Dom
AF15Q14 Dom
AF1Q Dom
$ less gene_transcripts.tsv
Symbol Ensembl Transcript ID CDS Length
MADD ENST00000311027 4944
MAST2 ENST00000361297 5397
ZNF32 ENST00000374433 822
LDB2 ENST00000304523 1122
HIST1H1E ENST00000304218 660
C9orf150 ENST00000319264 687
实例解析
- 数据读取
OncodriveCLUSTL只需要两个主要的输入,突变文件和注释文件及基因在CGC数据库中对应的注释信息。
1. 突变的文件
TSV文件包含映射到参考基因组(例如,人类hg19)的SNV。如果存在其他突变类型(插入、缺失、双碱基替换等),则在分析过程中将其过滤掉。这个文件必须至少包含以下5个带标题的列。
-
CHROMOSOME: 1, 2,…, X, Y
-
POSITION: Integer indicating the position of the mutation
-
REF: Reference nucleotide
-
ALT: Alternate nucleotide
-
SAMPLE: Identifier of the sample
Additional columns are:
-
CANCER_TYPE: Type of tumor. When specified, OncodriveCLUSTL will calculate one mutational profile for each cancer type and mutations will be randomized accordingly.
-
SIGNATURE: User-defined group to compute k-mer nucleotide mutational probabilities. When specified, OncodriveCLUSTL will calculate one mutational profile for each group and will randomize each mutation accordingly.
对应exmaple文件夹里面的 tcga.BRCA.nonsyn.txt和tcga.BRCA.syn.txt
2. 注释文件
注释文件包括基因组元素坐标(GEs)的TSV文件。至少以下5列带标题:
-
CHROMOSOME: 1, 2,…, X, Y
-
START: Starting position of the genomic region
-
END: Final position of the genomic region
-
ELEMENT: Identifier of the GE
-
SYMBOL: Symbol of the GE. OncodriveCLUSTL will analyze GEs as SYMBOL + ELEMENT.
Additional columns are:
- STRAND: Strand of the GE coordinates (“+” or “-”).
对应data文件夹里面的gene_transcripts.tsv。该文件最重要的是第一列和最后一列,第一列表示基因,最后一列表示突变所在的蛋白质位置。
3. 基因在CGC数据库中对应的注释信息
--cgc参数指定的是基因在CGC数据库中对应的注释信息,是可选的,对应exmaple文件夹里面的CGC_phenotype.tsv
运行操作
运行十分简单,主要就是准备输入文件而已,实例运行:
oncodriveclust \
-m 3 \
--cgc \
data/CGC_phenotype.tsv \
examples/tcga.BRCA.nonsyn.txt \
examples/tcga.BRCA.syn.txt \
data/gene_transcripts.tsv
这是分析BRCA(乳腺癌)数据集,OncodriveCLUST输出将放置在使用-o参数指定的路径(如果没有指定,那么将使用当前目录)。注意,在对输入的分析中,我们使用了肿瘤队列中至少10个突变的任意值来将该基因纳入到COSMIC数据集的分析中,并在TCGA数据集的分析中使用了3图。这可以通过使用-m参数来定义。使用可选标志-c,突变集群的坐标将包含在输出文件中。
默认生成结果文件oncodriveclust-results.tsv,如下:
$ head oncodriveclust-results.tsv
GENE CGC GENE_LEN GENE_NUM_MUTS MUTS_IN_CLUST NUM_CLUSTERS GENE_SCORE ZSCORE PVALUE QVALUE
KRAS Dom 190 3 3 1 1.0 4.17671455905 1.47874885544e-05 0.0005077037737
KLRG2 410 3 3 1 1.0 4.17671455905 1.47874885544e-05 0.0005077037737
KRT38 457 3 3 1 1.0 4.17671455905 1.47874885544e-05 0.0005077037737
AKT1 Dom 481 12 11 1 0.916666666667 3.79776654893 7.30028674072e-05 0.00187982383574
PIK3CA Dom 1069 186 178 7 0.855624278036 3.52018384847 0.00021562388305 0.00444185199083
VASN 674 5 4 1 0.8 3.26723933475 0.000543009128935 0.00932165671338
KIF19 999 3 3 1 0.784521187523 3.1968513525 0.000694682523388 0.0100330867162
ZNF598 905 4 3 1 0.75 3.03987052868 0.00118339934876 0.0100330867162
FGFR2 Dom 823 4 3 1 0.75 3.03987052868 0.00118339934876 0.0100330867162c
最后根据pvalue和qvalue对结果进行筛选,挑选显著性的驱动基因既可以完成,若是需要进行完美的展示可以根据文章中的方式进行展示,如下:
References:
-
Dees N D, Zhang Q, Kandoth C, et al. MuSiC: identifying mutational significance in cancer genomes[J]. Genome research, 2012, 22(8): 1589-1598.
-
Tamborero D, Gonzalez-Perez A, Lopez-Bigas N. OncodriveCLUST: exploiting the positional clustering of somatic mutations to identify cancer genes[J]. Bioinformatics, 2013, 29(18): 2238-2244.
-
Mularoni, Loris, et al. “OncodriveFML: a general framework to identify coding and non-coding regions with cancer driver mutations.” Genome biology 17.1 (2016): 128.
-
Arnedo-Pac C, et al. OncodriveCLUSTL: a sequence-based clustering method to identify cancer drivers. Bioinformatics. 2019;35(22):4788–4790.






















 1311
1311











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








